Nature sub-journal, 96%의 정확도로 AI가 서열로부터 단백질-리간드 상호작용 예측
- PHPz원래의
- 2024-07-11 12:56:20836검색
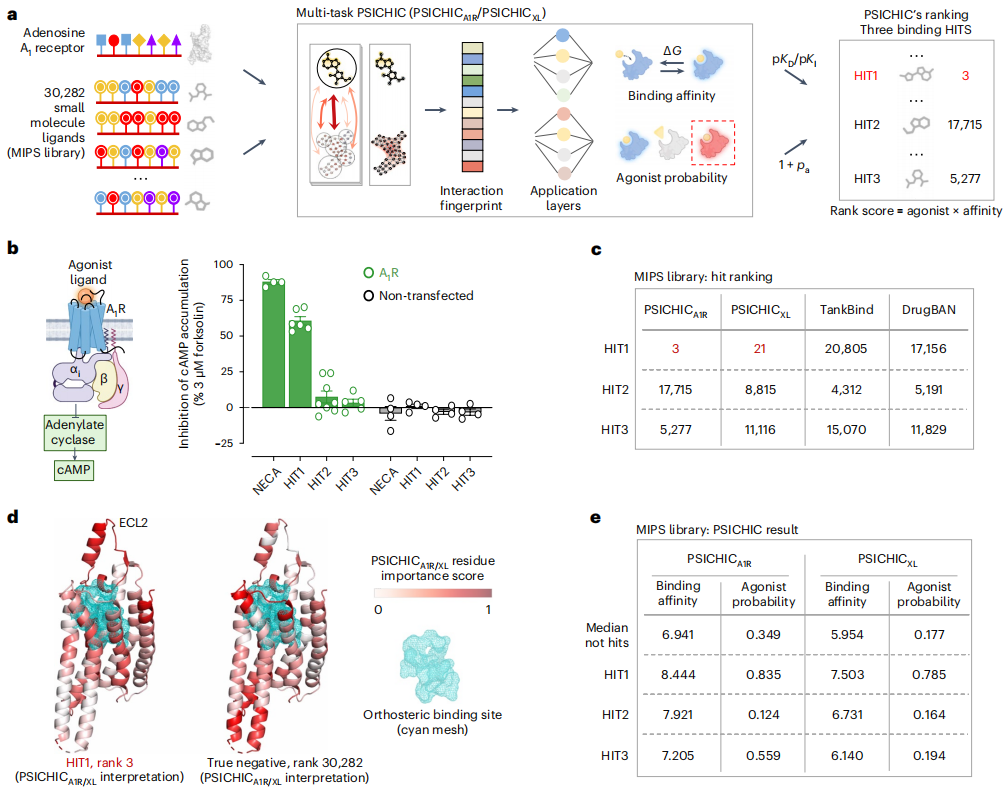
약물 개발에서는 단백질에 대한 저분자 리간드의 결합 친화력과 기능적 효과를 결정하는 것이 중요합니다. 현재의 계산 방법은 이러한 단백질-리간드 상호 작용 특성을 예측할 수 있지만 고해상도 단백질 구조가 없으면 정확도가 손실되는 경우가 많으며 기능적 효과를 예측할 수 없습니다.
Monash University와 Griffith University의 연구원들은 데이터 디코딩 상호 작용 지문 시퀀스에서 직접 물리화학적 제약 조건을 결합하는 프레임워크인 PSICHIC(PhySIcoCHhemICal Graph Neural Network)을 개발했습니다. 이를 통해 PSICHIC은 단백질-리간드 상호 작용 뒤에 있는 메커니즘을 해독하여 최첨단 정확성과 해석 가능성을 달성할 수 있습니다.
구조적 데이터 없이 동일한 단백질-리간드 쌍에 대해 훈련된 PSICHIC은 결합 친화도 예측에서 선도적인 구조 기반 방법과 동등하거나 심지어 그 이상으로 수행되었습니다.
PSICHIC의 해석 가능한 지문은 상호 작용에 관여하는 단백질 잔기와 리간드 원자를 식별하고 단백질-리간드 상호 작용의 선택성 결정 요인을 밝히는 데 도움이 됩니다.
이 연구의 제목은 "시퀀스 데이터에서 단백질-리간드 상호 작용 지문을 학습하기 위한 물리화학적 그래프 신경망"이며 2024년 6월 17일 "Nature Machine Intelligence"에 게재되었습니다.

약물 발견 과정에서는 리간드와 특정 단백질의 선택적 상호작용이 단백질에 대한 저분자 리간드의 결합 친화도와 기능적 효과를 이해하는 것이 중요합니다. 약의 기대효과.
그러나 현재의 계산 방법은 단백질-리간드 상호 작용 특성을 예측할 수 있지만 고해상도 단백질 구조가 없으면 예측 정확도가 떨어지는 경우가 많으며 기능적 효과 예측에도 어려움이 있습니다.
시퀀스 기반 방법은 비용과 리소스 측면에서 더 많은 장점이 있지만(예: 값비싼 실험 구조 결정 프로세스가 필요하지 않음) 이러한 방법은 종종 패턴 일치에서 과도한 자유도 문제에 직면하여 쉽게 과적합으로 이어질 수 있습니다. 일반화 기능이 제한되어 구조 기반 또는 복합 기반 방법에 비해 성능 격차가 발생합니다.
물리화학 그래프 신경망
모나쉬 대학과 그리피스 대학의 연구팀은 신체 및 화학적 원리에 따라 서열 데이터에서 단백질-리간드를 직접 해독하는 방법인 PSICHIC(물리화학 그래프 신경망)을 개발했습니다. 지문 방식. 이전 시퀀스 기반 모델과 달리 PSICHIC은 물리화학적 제약 조건을 고유하게 통합하여 최첨단 정확성과 해석성을 달성합니다.
2D 시퀀스 기반 방법인 PSICHIC은 클러스터링 알고리즘을 적용하여 2D 플롯에 이러한 제약 조건을 생성하고 부과하므로 PSICHIC은 훈련 중에 단백질-리간드 상호 작용을 결정하는 합리적인 기본 패턴에 주로 적응할 수 있습니다.

(출처: 논문)
성능 검증 및 비교
구조 데이터 없이 동일한 단백질-리간드 쌍에 대한 훈련 후 PSICHIC은 결합 친화도 예측에서 뛰어난 성능을 발휘합니다. -예술 구조 기반 및 복합 기반 방법은 이에 필적하거나 능가합니다.
PDBBind v2016 및 PDBBind v2020 데이터 세트에 대한 실험 결과에 따르면 PSICHIC은 여러 지표에서 TransCPI, MolTrans 및 DrugBAN과 같은 다른 시퀀스 기반 방법보다 성능이 뛰어난 것으로 나타났습니다.
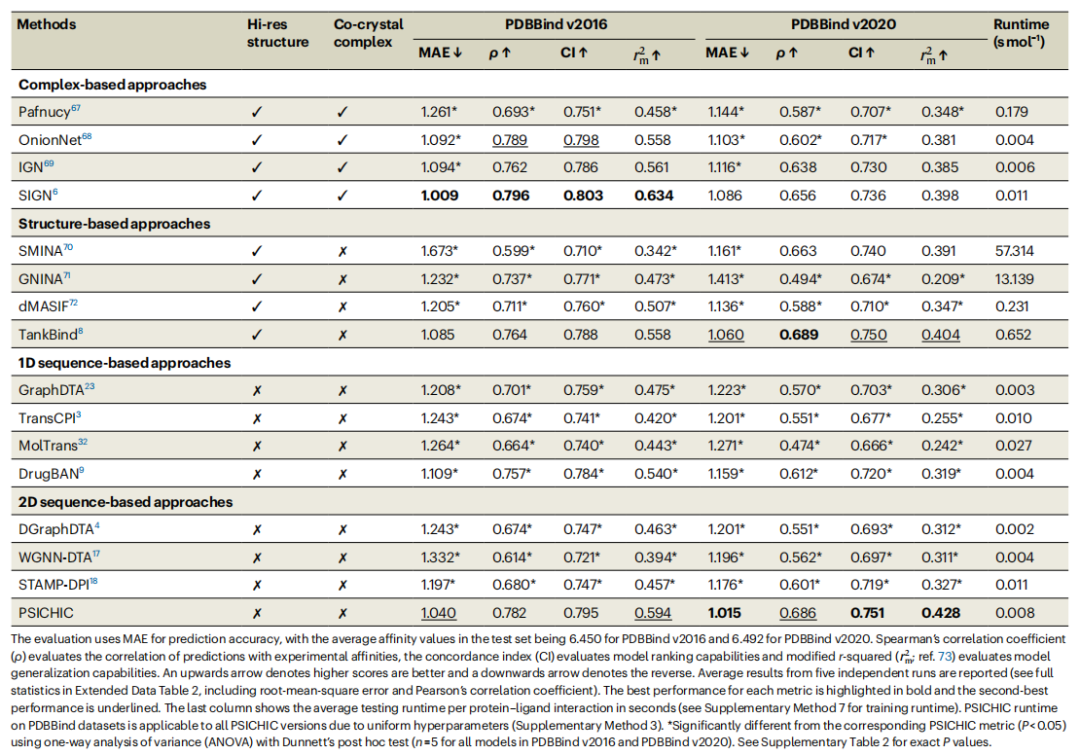
PDBBind v2016 및 PDBBind v2020 벤치마크에 대한 단백질-리간드 결합 친화도 예측의 성능 통계 요약입니다. (출처: 논문)
구체적으로:
- PSICHIC은 특히 예측 정확도 및 일반화 능력 측면에서 예측 오류가 낮고 상관 지수가 높음을 나타냅니다.
- PSICHIC은 기능적 효과 예측에서 최대 96%의 정확도를 달성합니다.
또한:
- PSICHIC은 결합 부위와 주요 리간드 작용기를 식별하는 데 탁월합니다.
- 다중 단백질-리간드 복합체 구조(예: PDB 6K1S 및 6OXV) 분석에서 PSICHIC은 중요한 결합 잔기와 리간드 작용기를 정확하게 찾아 서열 데이터에서 단백질-리간드를 직접 디코딩하는 능력을 검증할 수 있었습니다. 신체 상호작용 패턴.
- 이 능력은 특히 서열 데이터에서 단백질-리간드 결합 부위와 주요 잔기를 예측하는 능력에 반영됩니다.
그림: 대화형 지문을 사용한 가상 검사. (출처: 논문)
흥미롭게도 PSICHIC의 해석 가능한 지문은 서열 데이터만으로 단백질-리간드 상호 작용의 기본 메커니즘을 해독하고 결합 부위 단백질 잔기와 관련 리간드 원자 능력을 식별하는 능력을 얻었음을 보여줍니다. 결합 친화도 레이블은 있고 상호 작용 정보는 없는 시퀀스 데이터입니다.
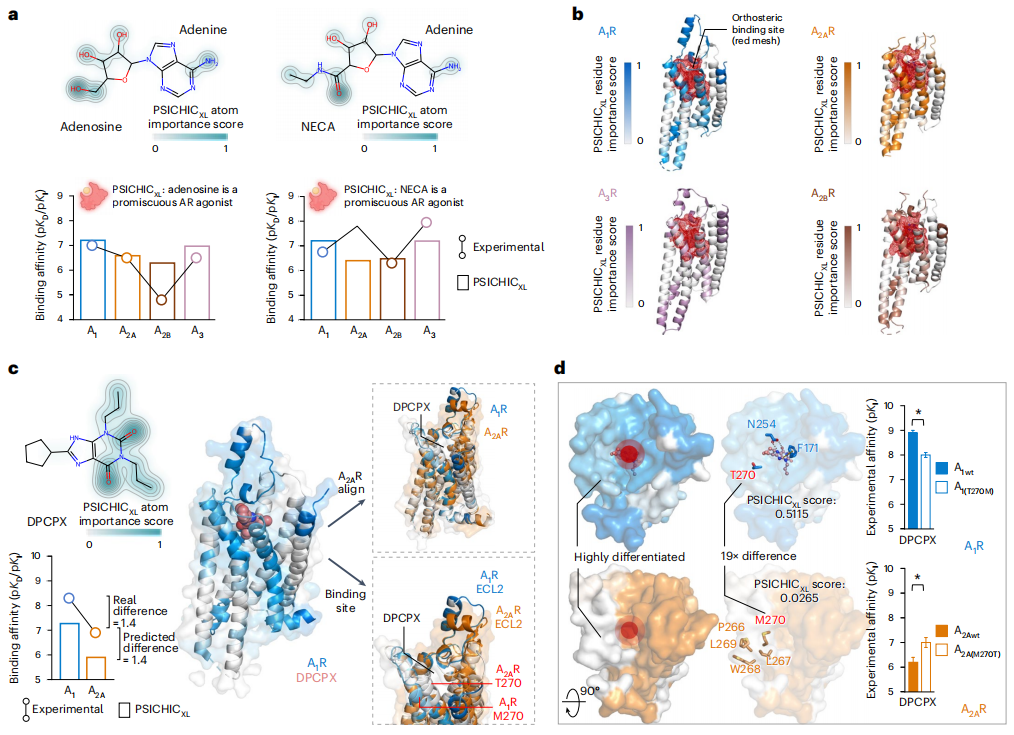
- 단백질-리간드 상호작용 지문은 리간드와 단백질 잔기 사이의 특정 상호작용 특성을 설명합니다.
- PSICHIC은 시퀀스 데이터만 사용하여 해석 가능한 상호 작용 지문을 얻는 독특한 접근 방식을 제공합니다.
- PSICHIC은 제약 조건을 통합하고 단백질-리간드 상호 작용 메커니즘을 밝히고 상호 작용 특성을 효율적으로 예측하는 새로운 기능을 보여줍니다.
- PSICHIC은 3D 데이터의 필요성을 제거하여 대규모 시퀀스 데이터베이스에서 강력한 학습을 위한 기반을 마련합니다.
미래 전망
- PSICHIC 분석을 이종삼합체 G 단백질과 복합체를 이루는 GPCR과 같은 단백질 복합체로 확장합니다.
- 알로스테릭 조절과 같은 복잡한 상호작용을 탐구하면 알로스테릭 리간드가 단백질 표적 내에서 오르토스테릭 리간드를 어떻게 조절하는지 이해하는 데 도움이 됩니다.
- PSICHIC은 다양한 응용 분야에서 견고성과 효율성을 입증했으며 향후 개발 가능성이 넓습니다.
위 내용은 Nature sub-journal, 96%의 정확도로 AI가 서열로부터 단백질-리간드 상호작용 예측의 상세 내용입니다. 자세한 내용은 PHP 중국어 웹사이트의 기타 관련 기사를 참조하세요!