단 몇 초 만에 단백질 역학 정보를 정확하게 추론할 수 있는 산동대학교, 베이징 공과대학 등의 AI 모델 RMSF-net이 네이처(Nature) 저널에 게재되었습니다.
- 王林원래의
- 2024-07-10 14:55:00898검색

단백질의 역학은 그 메커니즘을 이해하는 데 중요합니다. 그러나 단백질 동역학 정보를 계산적으로 예측하는 것은 어렵습니다.
여기서 산동 대학교, BioMap, 베이징 공과 대학, 후베이 의과 대학, 닝샤 의과 대학교 및 압둘라 과학 기술 대학교(KAUST)의 연구팀은 이전 방법보다 성능이 뛰어나고 대규모 단백질 역학 데이터 세트에서 최상의 결과를 생성하며, 모델은 단 몇 초 만에 단백질의 역학 정보를 정확하게 추론할 수 있습니다.
실험적 단백질 구조 데이터와 cryo-EM 데이터의 통합을 통해 효과적으로 학습함으로써 이 방법은 cryo-EM 이미지와 PDB 모델 간의 대화형 양방향 제약 조건과 감독을 정확하게 식별하여 역학 예측의 효율성을 극대화할 수 있습니다.
RMSF-net은 단백질 역학 연구에서 중요한 역할을 하는 무료 도구입니다.
이 연구는 "복잡한 원자 구조와 Cryo-EM 밀도 정보를 통합하는 딥 러닝을 통한 단백질 구조 유연성의 정확한 예측"이라는 제목으로 "Nature Communications"에 7월 2일 게재되었습니다.

- https://www.nature.com/articles/s41467-024-49858-x
RMSF-net GitHub 주소:
- https://github. com/XintSong/RMSF-net
단백질 역학
단백질 역학은 메커니즘을 이해하는 데 중요합니다. 저온전자현미경(cryo-EM) 기술은 거대분자 구조가 3D 밀도 맵으로 표현되는 대부분의 단백질을 분해할 수 있습니다.
저온전자현미경의 한계
원본 2D 입자 이미지의 낮은 해상도와 신호 대 잡음비로 인해 저온전자현미경 분석은 재구성 과정에서 작은 형태 변화를 확인할 수 없습니다.
저온전자현미경에 딥러닝 적용
딥러닝 방법은 저온전자현미경 이미지의 자동 분석에 널리 사용됩니다. 고해상도 저온 EM 맵을 사용하면 저온 EM 맵에서 단백질 데이터 뱅크(PDB) 모델을 구성할 수 있습니다.
RMSF-net 개요
RMSF-net은 저온전자현미경 밀도 맵을 위한 신경망 모델입니다. 극저온-EM 밀도와 PDB 모델 정보를 활용하여 단 몇 초 만에 단백질 동적 정보를 정확하게 추론합니다.
RMSF
RMSF는 분자 역학(MD) 분석에서 분자 구조의 유연성을 평가하기 위해 널리 사용되는 측정 방법입니다. 주요 목적은 단백질 내의 국소 구조(잔류물, 원자)의 RMSF를 예측하는 것입니다.
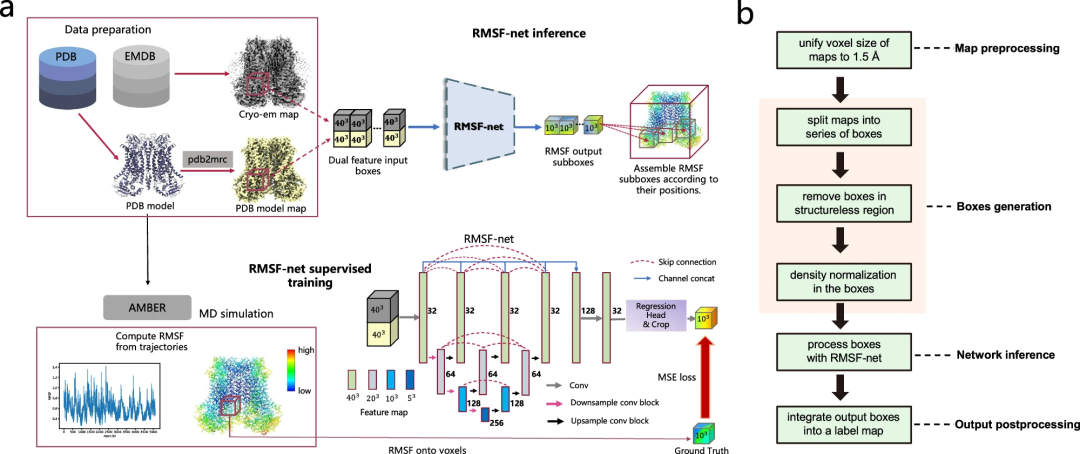
cryo-EM 이미지 외에도 RMSF-net은 PDB 모델을 추가 입력으로 활용하여 MD 시뮬레이션 결과에 매우 가까운 RMSF 예측을 생성합니다.
RMSF-net은 두 개의 상호 연결된 모듈을 포함하는 3차원 컨벌루션 신경망입니다. 메인 모듈은 Unet+(L3) 아키텍처를 사용하여 입력 밀도 상자의 기능을 인코딩 및 디코딩합니다. 또 다른 모듈은 1x1 컨볼루션을 활용하여 Unet+백본에서 생성된 기능 맵의 채널을 회귀합니다. 그런 다음 중앙 클리핑이 회귀 모듈 출력에 적용되어 중앙에 위치한 RMSF 하위 상자를 얻습니다. 여기서 복셀 값은 그 안에 포함된 원자의 RMSF에 해당합니다. 마지막으로 RMSF 하위 상자는 병합 알고리즘을 사용하여 RMSF 맵에 공간적으로 병합됩니다.
또한 연구원들은 RMSF-net의 훈련 및 검증을 위한 대규모 단백질 역학 데이터 세트를 구축했습니다. 여기서 PDB 모델이 적합한 335개의 극저온 EM 구조 항목을 선택하고 해당 MD 시뮬레이션을 수행했습니다. 종합적인 실험 결과는 RMSF-net의 효율성과 효과를 입증합니다.
표: 데이터 세트에 대한 다양한 RMSF 예측 방법의 성능. (출처: 논문)
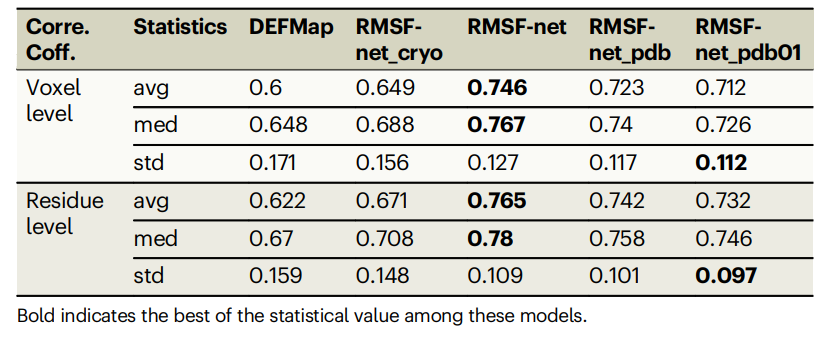
RMSF-net은 MD 시뮬레이션 결과와의 상관계수 0.746±0.127로 엄격한 5겹 교차 검증에서 좋은 성능을 보였습니다. RMSF-net의 상관계수는 DEFMap에 비해 15%, Baseline 방법에 비해 10% 향상되었다.
역학 예측의 해석성
연구원들은 비교 실험을 통해 RMSF-net 역학 예측의 해석성을 향상시켰습니다. RMSF 예측 프로세스는 두 단계로 나뉩니다.
- 구조 정보 추출(Occ2RMSF-net)
- 추출된 구조 정보를 기반으로 한 운동학 예측
연구 결과에 따르면 저온 전자 현미경 스펙트럼(예: DEFMap 또는 RMSF-net_cryo)을 기반으로 한 모델의 역학 예측은 주로 다음과 같은 방식으로 이루어집니다. 단백질 구조 구현 해석. 이는 구조-기능 관계의 첫 번째 원칙과 일치하는 단백질 토폴로지와 역학 간의 연결을 강조합니다.
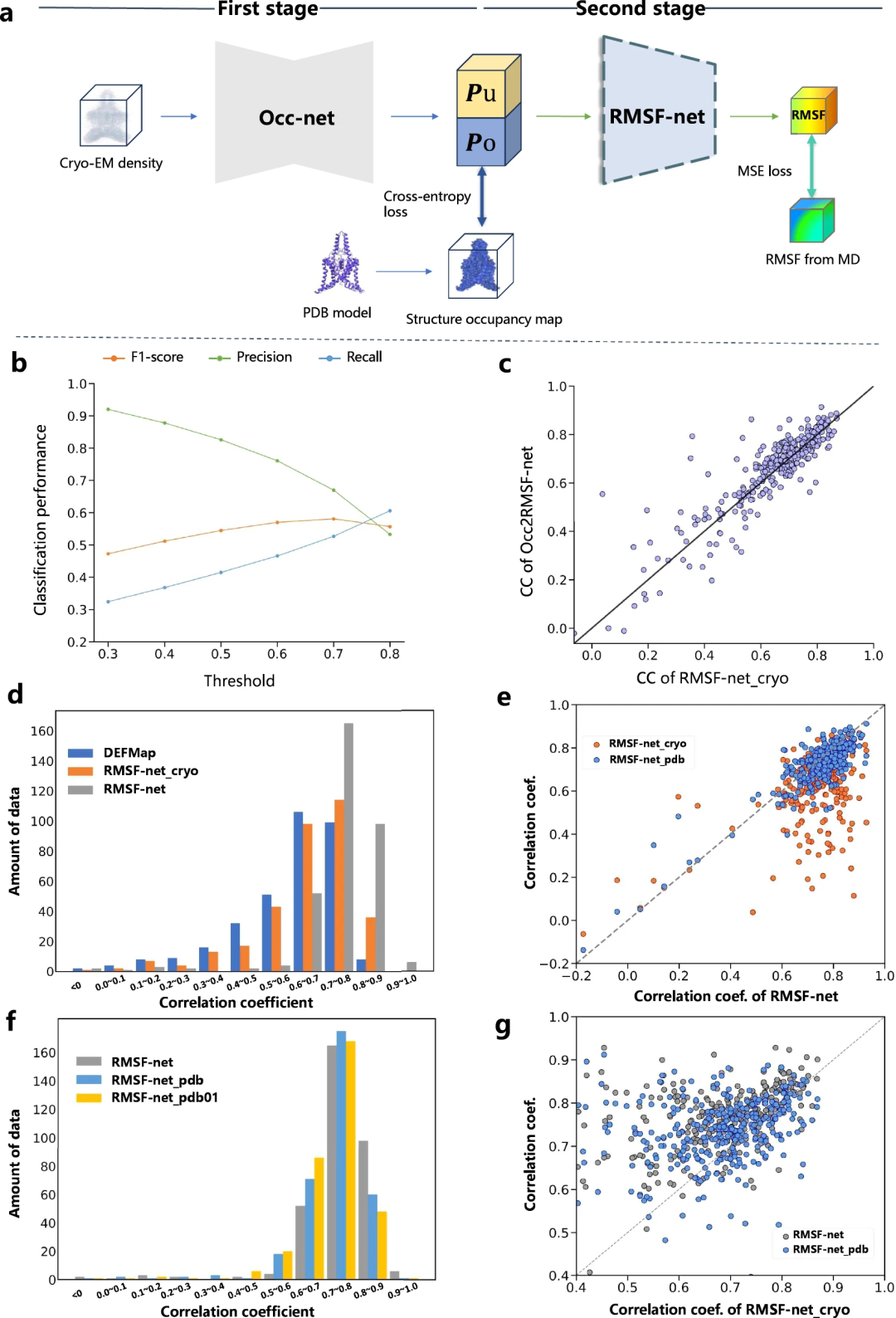
또한 RMSF-net_cryo, RMSF-net_pdb 및 최종 이중 조합 RMSF-net의 포괄적인 비교를 통해 다음이 입증되었습니다. 한편으로는 PDB 모델의 구조 정보가 재생됩니다. 심층 모델이 MD 시뮬레이션에서 구조 토폴로지와 유연성 사이의 패턴을 학습하는 RMSF-net 역할의 주요 역할이며, 반면에 모델은 저온 전자 현미경의 이질적인 밀도 분포에 포함된 운동 정보에 의해 더욱 향상됩니다. 지도. 이러한 결과는 RMSF-net의 단백질 역학 예측에 대한 저온 EM 맵 및 PDB 모델 정보의 보완적인 역할을 검증합니다.
제한 사항 및 향후 방향
RMSF-net이 주로 용액 내 순수 단백질과 그 복합체의 유연성을 예측하는 데 제한된다는 것은 부인할 수 없습니다. 이 접근법은 소분자 리간드에 결합되거나 막 환경에서 단백질의 동적 특성과 관련하여 특정 국소 영역에서 부정확성을 나타낼 수 있습니다.
RMSF-net의 뛰어난 성능은 이 방향에 대한 추가 연구의 타당성을 보여줍니다. 이 연구는 핵산과 단백질-핵산 복합체로 확장되지 않았습니다. 다중 구조 예측 및 전이 분석을 포함하여 거대분자 역학의 모든 측면에 대한 포괄적인 특성화에는 향후 더욱 광범위하고 심층적인 연구가 필요합니다.
그럼에도 불구하고, RMSF-net은 단백질 역학을 예측하는 도구로서 뛰어난 성능과 초고속 처리 속도로 인해 단백질 구조 및 역학 연구에 여전히 큰 응용 가능성을 가지고 있습니다.
참고: 표지는 인터넷에서 가져온 것입니다
위 내용은 단 몇 초 만에 단백질 역학 정보를 정확하게 추론할 수 있는 산동대학교, 베이징 공과대학 등의 AI 모델 RMSF-net이 네이처(Nature) 저널에 게재되었습니다.의 상세 내용입니다. 자세한 내용은 PHP 중국어 웹사이트의 기타 관련 기사를 참조하세요!

