
편집자 | 무 껍질
단백질은 신체가 병원균과 싸우는 데 있어 확고한 도구이며 실험적 테스트를 위한 잠재적인 치료법을 좁히는 데 사용됩니다. 고품질의 단백질 구조가 필요하며, 단백질은 종종 완전히 또는 부분적으로 단단한 것으로 간주됩니다.
여기서 베를린 자유대학교(Freie Universität Berlin)의 연구원들은 서열 정보로부터 직접 단백질-리간드 복합체의 완전히 유연한 전체 원자 구조를 예측할 수 있는 인공 지능 시스템을 개발했습니다.
고전적인 도킹 방법이 여전히 우수하지만 이 역시 표적 단백질의 결정 구조에 따라 달라집니다. 유연한 전체 원자 구조를 예측하는 것 외에도 예측 신뢰도 측정법(plDDT)을 사용하여 정확한 예측을 선택하고 강한 결합제와 약한 결합제를 구별할 수 있습니다.
이 연구의 제목은 "Umol을 이용한 서열 정보로부터 단백질-리간드 복합체의 구조 예측"이며 2024년 5월 28일 "Nature Communications"에 게재되었습니다.

단백질-단백질 표적 접촉은 신약을 평가하고 알려진 물질을 재배치하는 데 중요한 문제입니다. 기존 접촉 방법에는 고품질의 단백질 구조가 필요하고 정확한 접촉 자세를 결정하기가 어렵고 대부분 결합 능력(친화도) 평가에 기반을 두고 있어 구조적 안정성과 같은 다른 요소를 반영하기 어렵습니다. 그러나 기존 접촉 방법은 고품질 단백질 구조, 정확한 접촉 포즈 및 다중 기반 친화력 평가의 필요성으로 인해 제한됩니다. 따라서 새로운 리간드의 탐색은 단백질 조립과 구조 평가의 결합된 접근 방식에 의해 제한됩니다.
이 분야에 기계 학습이 적용되었지만 알려진 대상 영역에서의 성능은 여전히 채점 기능을 기반으로 하는 고전적인 방법을 능가하지 못했습니다. 더욱이, 예측된 단백질 구조는 리간드 도킹에 직접 사용하기에 적합하지 않은 경우가 많습니다.
또한 평가 세트의 구조를 유사성보다는 릴리스 시간을 기준으로 나누면 편향이 도입되며, 특히 훈련에서 볼 수 없는 수용체 구조를 마주할 때 성능이 절반으로 떨어지게 됩니다.
단백질 유연성은 결합 상태에 도달하고 성공적인 도킹에 중요합니다. RoseTTAFold All-Atom은 단백질을 예측할 때 리간드를 결합할 수 있지만 PoseBusters 테스트 세트의 성공률은 42%에 불과하며 눈에 보이지 않는 단백질의 경우 매우 어렵습니다. 단백질 거동은 알려져 있지 않으며, 이는 단백질-리간드 복합체 구조 예측의 과제가 아직 완전히 해결되지 않았음을 나타냅니다.
베를린 자유대학교(Freie Universität Berlin) 팀은 AlphaFold2에 EvoFormer를 확장하여 서열 정보를 기반으로 단백질-리간드 복합체의 구조를 예측할 수 있는 AI 방법을 개발했습니다. 이 네트워크는 3D 궤적이 포함되지 않고 템플릿 구조 또는 추가 결정학적 리간드 데이터가 입력으로 또는 훈련 중에 사용된다는 점을 제외하면 RFAA와 유사합니다.
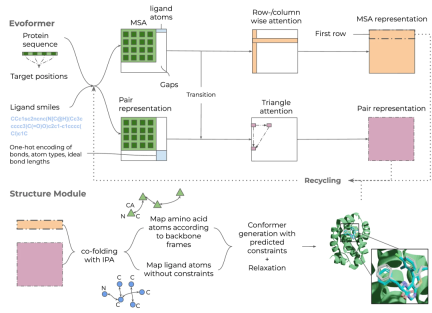
그림: Umol 개요. (출처: 논문)
단백질 서열에서 시작하여 대체 단백질 표적(포켓) 및 리간드 SMILES는 다중 서열 정렬(MSA) 및 결합 매트릭스를 생성합니다. 이를 통해 네트워크 내에서 특징이 생성되고 3D 구조가 생성됩니다. 최종 단백질-리간드 복합체 구조를 생성하는 데 구조적 정보가 필요하지 않으므로 단백질이나 리간드 유연성에 대한 제한이 없습니다.
Umol은 가장 가까운 RoseTTAFold All-Atom 및 NeuralPlexer1과 비교하여 PoseBusters 테스트 세트에 포켓 정보를 포함할 때 각각 45%, 42%의 더 높은 성공률(SR, 리간드 RMSD ≤ 2Å)을 달성하여 24%를 달성합니다. 이는 단백질-리간드 구조 예측에서 가장 성능이 좋은 방법입니다.

그림: 예측 정확도. (출처: 논문)
Umol에서 포켓 정보를 제거하고 RFAA에서 템플릿 정보를 제거하면 SR이 각각 18%, 8%로 떨어집니다. AF 예측과 함께 DiffDock을 사용하는 경우 정확도는 21%이지만 매우 정확한 인터페이스 예측(포켓 RMSD
2Å 성공 임계값 바로 위에 있는 많은 리간드 포즈는 비교할 수 있을 가능성이 높으며, 이는 보다 유연한 채점 시스템이 필요할 수 있음을 시사합니다. Umol의 성공률은 2.35Å 임계값에서 AutoDock Vina를 초과합니다. 기본 단백질 구조가 채점에 사용되지 않으면 작은 정렬 오류라도 문제가 될 수 있습니다.
공접힘 단백질-리간드 복합체는 약물 재배치를 가속화할 가능성이 있습니다. 특히 연구자들은 리간드(plDDT)의 예측된 lDDT가 정확한 도킹 포즈를 선택하는 데 사용될 수 있는 반면, 단백질 포켓의 pIDDT는 정확한 인터페이스를 선택하는 데 적합하다는 것을 발견했습니다.

그림: 신뢰도 측정항목 및 정확성. (출처: 논문)
리간드 plDDT는 또한 친화도가 높은 리간드와 친화도가 낮은 리간드를 분리하는데, 이는 Umol 및 Umol-포켓 불확실성에 대한 예측 중 일부가 약한 결합제가 될 수 있음을 시사합니다. 이는 Umol의 기능을 더욱 입증하고 단백질-리간드 상호작용의 중요한 측면이 이해되는 것으로 나타난다는 점을 강조합니다.

그림: BindingDB 예측. (출처: 논문)
포켓 정보 없이 18%의 정확도에도 불구하고 네트워크는 여전히 강한 바인더와 약한 바인더를 어느 정도 구분할 수 있습니다. 이는 알려지지 않은 복합체에 주석을 달 때 특히 유용하며, 팀은 매우 높은 신뢰도로 336개의 단백질-리간드 구조를 제시했습니다(리간드 plDDT>85). 이러한 구조가 합리적으로 보이고 L-plDDT 점수가 높더라도 여전히 실험적으로 검증해야 한다는 점에 유의해야 합니다.
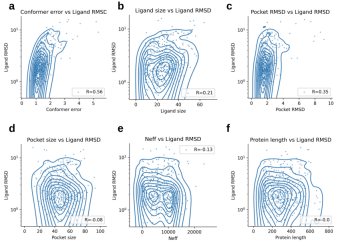
그림: Umol-pocket을 사용하여 PoseBusters 테스트 세트(n=428)에서 예측된 다양한 특징과 리간드 RMSD(LRMSD) 간의 관계를 분석합니다. (출처: 논문)
연구원들은 모델의 예측 성능과 "동일한 단백질 또는 리간드와 관련된 다양한 특징" 사이의 명확한 관계를 찾지 못했습니다.

그림: 가장 어려운 5가지 구조물. (출처: 논문)
그러나 우몰포켓은 다른 방법으로는 예측하기 어려운 5건 중 3건에서 정확했습니다. 훈련된 네트워크를 반전시킴으로써 새로운 리간드 결합 단백질 또는 단백질 결합 리간드를 설계할 수 있습니다. 또 다른 옵션은 전이 학습을 사용하여 동일한 목적을 위한 생성 확산 모델을 만드는 것입니다. 이 경우, 친화력이 높은 결합제를 생성하기 위해 리간드 또는 단백질 plDDT를 최대화할 수 있습니다.
PDBbind의 현재 버전에는 2019년에 PDB에서 처리된 데이터가 포함되어 있습니다. 그 이후로 추가적인 단백질-리간드 복합체가 제출되었으며, 이는 더 큰 정확도를 달성할 수 있음을 시사합니다.
그러나 의미 있는 단백질-리간드 도킹 결과를 얻으려면 어느 정도의 정밀도가 필요한지는 현재 불분명합니다. 단백질 구조 예측의 높은 정확도는 작은 분자나 RNA와 같은 다른 분자와 관련된 작업에서는 달성할 수 없습니다.
단백질 공진화 정보가 없으면 구조 예측의 정확도가 급격히 떨어집니다. 작은 분자나 RNA에 대한 유사한 정보 소스가 없기 때문에 원자 표현에 의존해야 합니다.
표: PoseBuster 벤치마크 세트의 성공률(RMSD가 2Å 이하인 리간드의 백분율)을 PDBBind 2020 버전의 서열 동일성(seqid)으로 나눈 값입니다. (출처: 논문)

연구자들은 포켓 정보가 매우 효과적이라고 믿고 있으며, 포켓 정보가 없으면 딥러닝 방법은 과적합되기 쉬운 것 같습니다. 이 발견은 PoseBusters 테스트 세트의 많은 분자가 훈련 데이터 세트에서 매우 유사한 유사성을 포함하고 있지만 이러한 유사성은 모델 성공과 상관 관계가 없다는 관찰을 더욱 확증합니다.

그림: 일부 테스트. (출처: 논문)
Vina나 Gold와 같은 구조 기반 도킹 방법에서는 동일한 정도의 과적합이 관찰되지 않습니다. 이는 원자 채점 기능을 기반으로 하고 따라서 단백질 상동성에 동일한 정도로 의존하지 않기 때문에 예상됩니다.
딥 러닝 방법은 훈련 세트에서 훨씬 더 높은 성능을 보였으며, 이는 단백질 상동성이 단백질-리간드 도킹에서 중요한 역할을 한다는 것을 나타냅니다. 테스트 세트의 RFAA 성능은 훈련 세트의 성능보다 높으며 이는 훈련 세트와 테스트 세트 사이의 데이터 유출 가능성을 나타냅니다.
결론적으로, 단백질-리간드 상호 작용의 복잡성을 완전히 이해하려면 아직 갈 길이 멀지만, 딥 러닝을 사용하여 전체 복합체의 구조를 예측하면 과학자들은 해결책에 더 가까워질 수 있습니다.
Umol: https://github.com/patrickbryant1/Umol
논문 링크: https://www.nature.com/articles/s41467-024-48837-6
위 내용은 단백질-리간드 복합체의 구조를 직접 예측하기 위해 서열 정보를 사용하는 RoseTTAFold 시리즈보다 성공률이 높습니다.의 상세 내용입니다. 자세한 내용은 PHP 중국어 웹사이트의 기타 관련 기사를 참조하세요!
 California는 AI를 빠른 트랙 산불 복구 허가에 탭합니다May 04, 2025 am 11:10 AM
California는 AI를 빠른 트랙 산불 복구 허가에 탭합니다May 04, 2025 am 11:10 AMAI는 산불 복구 허가를 간소화합니다 기계 학습 및 컴퓨터 비전을 활용하는 호주 기술 회사 인 Archistar의 AI 소프트웨어는 지역 규정 준수를위한 건축 계획 평가를 자동화합니다. 이 예비 검증은 중요합니다
 미국이 에스토니아의 AI 기반 디지털 정부로부터 배울 수있는 것May 04, 2025 am 11:09 AM
미국이 에스토니아의 AI 기반 디지털 정부로부터 배울 수있는 것May 04, 2025 am 11:09 AM에스토니아의 디지털 정부 : 미국의 모델? 미국은 관료적 비 효율성과의 투쟁이지만 에스토니아는 설득력있는 대안을 제공합니다. 이 소규모 국가는 AI가 구동하는 거의 100% 디지털화 된 시민 중심 정부를 자랑합니다. 이것은 아닙니다
 생성 AI를 통한 결혼 계획May 04, 2025 am 11:08 AM
생성 AI를 통한 결혼 계획May 04, 2025 am 11:08 AM결혼식을 계획하는 것은 기념비적 인 일이며, 종종 가장 조직화 된 커플조차도 압도적입니다. AI의 영향에 관한 진행중인 Forbes 시리즈의 일부인이 기사 (여기 링크 참조)는 생성 AI가 결혼 계획에 혁명을 일으킬 수있는 방법을 탐구합니다. 결혼식 pl
 Digital Defense AI 에이전트는 무엇입니까?May 04, 2025 am 11:07 AM
Digital Defense AI 에이전트는 무엇입니까?May 04, 2025 am 11:07 AM기업은 AI 에이전트를 판매하기 위해 점점 더 활용하는 반면 정부는 다양한 기존의 작업에이를 활용합니다. 그러나 소비자 옹호자들은 개인이 자주 조정 된 사람들에 대한 방어로 자신의 AI 에이전트를 소유해야 할 필요성을 강조합니다.
 생성 엔진 최적화에 대한 비즈니스 리더 안내서 (GEO)May 03, 2025 am 11:14 AM
생성 엔진 최적화에 대한 비즈니스 리더 안내서 (GEO)May 03, 2025 am 11:14 AMGoogle은 이러한 변화를 이끌고 있습니다. "AI 개요"기능은 이미 10 억 명 이상의 사용자에게 제공되며, 누군가가 링크를 클릭하기 전에 완전한 답변을 제공합니다. [^2] 다른 플레이어들도 빨리지면을 얻고 있습니다. Chatgpt, Microsoft Copilot 및 PE
 이 스타트 업은 AI 에이전트를 사용하여 악의적 인 광고와 가장하는 계정과 싸우고 있습니다.May 03, 2025 am 11:13 AM
이 스타트 업은 AI 에이전트를 사용하여 악의적 인 광고와 가장하는 계정과 싸우고 있습니다.May 03, 2025 am 11:13 AM2022 년에 그는 사회 공학 방어 스타트 업 도플을 설립하여 바로 그렇게했습니다. 그리고 사이버 범죄자들이 공격을 터보 차지하기 위해 더욱 진보 된 AI 모델을 활용함에 따라 Doppel의 AI 시스템은 비즈니스가 더 빠르게 빠르게 그리고 더 빠르게 그리고 규모로 싸우는 데 도움이되었습니다.
 세계 모델이 생성 AI 및 LLM의 미래를 근본적으로 재구성하는 방법May 03, 2025 am 11:12 AM
세계 모델이 생성 AI 및 LLM의 미래를 근본적으로 재구성하는 방법May 03, 2025 am 11:12 AMVoila는 적합한 세계 모델과 상호 작용하여 생성 AI 및 LLM을 실질적으로 향상시킬 수 있습니다. 그것에 대해 이야기합시다. 혁신적인 AI 혁신에 대한이 분석은
 2050 년 5 월 : 우리는 무엇을 축하하기 위해 떠났습니까?May 03, 2025 am 11:11 AM
2050 년 5 월 : 우리는 무엇을 축하하기 위해 떠났습니까?May 03, 2025 am 11:11 AM노동당 2050 년. 전국의 공원은 전통적인 바베큐를 즐기는 가족들로 가득 차고 향수를 불러 일으키는 퍼레이드는 도시 거리를 통해 바람을 피 웁니다. 그러나 축하 행사는 이제 박물관과 같은 품질을 가지고 있습니다.


핫 AI 도구

Undresser.AI Undress
사실적인 누드 사진을 만들기 위한 AI 기반 앱

AI Clothes Remover
사진에서 옷을 제거하는 온라인 AI 도구입니다.

Undress AI Tool
무료로 이미지를 벗다

Clothoff.io
AI 옷 제거제

Video Face Swap
완전히 무료인 AI 얼굴 교환 도구를 사용하여 모든 비디오의 얼굴을 쉽게 바꾸세요!

인기 기사

뜨거운 도구

에디트플러스 중국어 크랙 버전
작은 크기, 구문 강조, 코드 프롬프트 기능을 지원하지 않음

드림위버 CS6
시각적 웹 개발 도구

SublimeText3 중국어 버전
중국어 버전, 사용하기 매우 쉽습니다.

메모장++7.3.1
사용하기 쉬운 무료 코드 편집기

SublimeText3 Mac 버전
신 수준의 코드 편집 소프트웨어(SublimeText3)





