編集者 | Radish Skin
ディープ生成モデルは、医薬品設計を加速する大きな可能性を秘めています。ただし、既存の生成モデルは、データが限られているために一般化の課題に直面することが多く、その結果、革新的な設計が得られません。
これらの問題に対処するために、韓国の KAIST の研究者らは、標的結合ポケット内での相互作用に基づく相互作用設計を可能にする、相互作用を認識した 3D 分子生成機能フレームワークを提案しました。タンパク質とリガンドの相互作用の一般的なパターンを事前知識として利用することで、モデルは限られた実験データで高度な一般性を達成できます。同時に、タンパク質の質量とリガンドの質量を相互作用の一般的なパターンとして使用することにより、このモデルは一般性と高い特異性の間の良好なバランスを達成することができ、これにより医薬品設計に一般性と予測可能性がもたらされます。
生成されたまだ見ぬターゲットリガンドの性能は、結合姿勢の確実性、親和性、多様性、新規性を分析することによって包括的に評価されました。さらに、潜在的な突然変異選択的阻害剤の効率的な設計は、このアプローチが構造に基づいた薬剤設計に適用できることを示しています。
この研究は「相互作用誘導型薬物設計のための3D分子生成フレームワーク」と題され、2024年3月27日に「Nature Communications」に掲載されました。

データキャプチャと科学的問題において、深層学習モデルの適切な階層的な事前知識は、一般化可能なモデルを開発するために重要です。たとえば、AlphaFold は、共進化情報と残基ペア表現を活用してタンパク質構造を予測することに成功しました。深層生成モデルは医薬品設計のパラダイムを変えていますが、その性能は医薬品分子の活性データの欠如によって制限されており、その結果一般化能力が低くなります。深層生成モデルのパフォーマンスを向上させるには、薬物分子活性データの一般化への適合性を確保するための適切な事前知識が必要です。これは、困難な化合物の構造や特性を予測するために重要です。
最近の生成機能モデルは、結合部位の三次元構造を利用して、活性データに依存せずに構造ベースのリガンド設計を行うことにより、モデルの波形形成機能を向上させています。十分に波形のモデルは、水素結合、塩橋、疎水性相互作用、π-π スタッキングなど、タンパク質とリガンドの相互作用の普遍的な特性を理解している必要があります。これは安定した結合構造を形成し、高い親和性を維持するために不可欠です。これらの遍在する相互作用パターンは、強力な薬剤の設計の基礎となります。
これらの状況に基づいて、KAISTの研究者らは相互作用を認識した3D分子生成フレームワークを提案しました。このフレームワークは、タンパク質とリガンドの相互作用の普遍的な性質を利用して、構造に基づいた医薬品設計を導きます。このフレームワークは、(1) 相互作用センシング条件の設定、および (2) 相互作用する 3D 分子の生成の 2 つの主要な段階で構成されます。
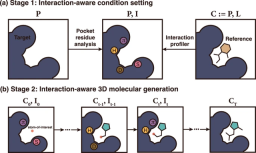
イラスト: フレームワークの概念図。 (出典: 論文)
フレームワークの最初の段階は、特定の結合部位 P のタンパク質原子を研究することによって相互作用条件 I を設定することを目的としています。研究者らは、水素結合、塩橋、疎水性相互作用、π-πスタッキングという4種類のタンパク質-リガンド相互作用を利用した。ここで研究者らは、モデルのトレーニングに PDB から派生した PDBbind 2020 データセットを使用したため、Protein Data Bank (PDB) で最も支配的な 4 つの相互作用タイプのみを考慮しました。
同時に、チームはタンパク質原子相互作用のセンシング制御戦略を開発しました。研究者らは、相互作用条件をタンパク質原子のセットに対する追加の相互作用タイプのワンホット ベクトルとして定義し、これは原子が特定の相互作用に参加できるかどうか、および相互作用におけるその役割を示します。
タンパク質原子は、アニオン、カチオン、水素結合供与体と受容体、芳香族、疎水性、非相互作用原子の 7 つのカテゴリのいずれかに分類されます。インタラクション情報全体を単一のインタラクション フィンガープリントとして表す代わりに、チームの戦略は、インタラクション条件をローカルに確立することを目的としています。
この研究では、研究者らは主に 2 つの戦略を通じてバッグ原子の相互作用カテゴリを決定しました。
生成段階では、受容体とリガンドの相互作用に関する情報が常に利用できるとは限らないため、各タンパク質原子を分析することによって相互作用条件を特定するために、相互作用カテゴリーの基準が事前に定義されます。自由相互作用条件。
トレーニング段階では、タンパク質-リガンド複合体のグラウンドトゥルース構造を使用して相互作用条件を抽出します。
The researchers also proposed a deep generative model called DeepICL for reverse engineering ligands, which gradually generates atoms in the ligand based on the three-dimensional environment of the pocket and the first-stage interaction conditions.
Although target pockets can form different combinations of protein-ligand interaction types depending on the bound ligand and its binding posture; the team's goal was to reverse engineer one using a 3D conditional generative model called DeepICL. For ligands that satisfy specific interaction combinations, the model can be applied to any type of protein. Researchers use local interaction conditions in the subpockets to which ligands should bind, rather than using the entire interaction information, to prevent undesirable biases toward specific pockets or ligand structures.
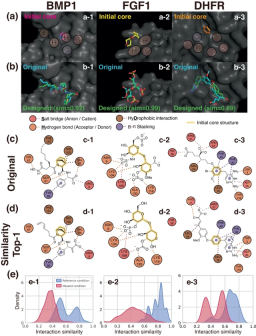
Illustration: Example of interaction-aware conditional ligand elaboration. (Source: Paper)
To demonstrate the framework's ability to perform general structure-based drug design, rather than using typical benchmarks consisting of 105 to 107 computer-generated protein-ligand binding structures, the researchers used only Approximately 104 real crystal structures were selected from the PDBbind database because a good generalization model can successfully extract appropriate features even for small-scale data.
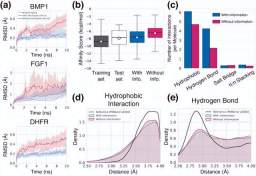
Illustration: Generating the universality of the framework. (Source: Paper)
The researchers evaluated their model by analyzing various aspects of the properties of the generated unseen target ligands—binding stability, affinity, geometric patterning, diversity, and novelty.
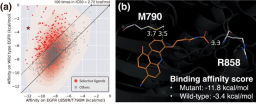
#aIllustration: Modulating selectivity through site-specific interactions controls ligand design. (Source: Paper)
The researchers used the model to solve practical problems where specific interaction sites play a critical role, demonstrating the applicability of their approach to structure-based drug design.
Paper link:https://www.nature.com/articles/s41467-024-47011-2
以上がKAISTは少量のデータで高い汎用性を実現し、医薬品設計のための3D分子生成の新しいフレームワークを開発の詳細内容です。詳細については、PHP 中国語 Web サイトの他の関連記事を参照してください。
 生成エンジン最適化に関するビジネスリーダーのガイド(GEO)May 03, 2025 am 11:14 AM
生成エンジン最適化に関するビジネスリーダーのガイド(GEO)May 03, 2025 am 11:14 AMGoogleはこのシフトをリードしています。その「AIの概要」機能はすでに10億人以上のユーザーにサービスを提供しており、誰もがリンクをクリックする前に完全な回答を提供しています。[^2] 他のプレイヤーも速く地位を獲得しています。 ChatGpt、Microsoft Copilot、およびPE
 このスタートアップは、AIエージェントを使用して悪意のある広告となりすましアカウントと戦っていますMay 03, 2025 am 11:13 AM
このスタートアップは、AIエージェントを使用して悪意のある広告となりすましアカウントと戦っていますMay 03, 2025 am 11:13 AM2022年、彼はソーシャルエンジニアリング防衛のスタートアップDoppelを設立してまさにそれを行いました。そして、サイバー犯罪者が攻撃をターボチャージするためのより高度なAIモデルをハーネスするにつれて、DoppelのAIシステムは、企業が大規模に戦うのに役立ちました。
 世界モデルがどのように生成AIとLLMの未来を根本的に再形成しているかMay 03, 2025 am 11:12 AM
世界モデルがどのように生成AIとLLMの未来を根本的に再形成しているかMay 03, 2025 am 11:12 AM出来上がりは、適切な世界モデルとの対話を介して、生成AIとLLMを実質的に後押しすることができます。 それについて話しましょう。 革新的なAIブレークスルーのこの分析は、最新のAIで進行中のForbes列のカバレッジの一部であり、
 2050年5月:私たちは祝うために何を残しましたか?May 03, 2025 am 11:11 AM
2050年5月:私たちは祝うために何を残しましたか?May 03, 2025 am 11:11 AM労働者2050年。全国の公園は、ノスタルジックなパレードが街の通りを通り抜ける一方で、伝統的なバーベキューを楽しんでいる家族でいっぱいです。しかし、お祝いは現在、博物館のような品質を持っています。
 あなたが聞いたことがないディープフェイク検出器はそれが98%正確ですMay 03, 2025 am 11:10 AM
あなたが聞いたことがないディープフェイク検出器はそれが98%正確ですMay 03, 2025 am 11:10 AMこの緊急かつ不安な傾向に対処するために、TEM Journalの2025年2月版の査読済みの記事は、その技術のディープフェイクが現在存在する場所に関する最も明確でデータ駆動型の評価の1つを提供します。 研究者
 Quantum Talent Wars:The Hidden Crisis Treatenting Tech'の次のフロンティアMay 03, 2025 am 11:09 AM
Quantum Talent Wars:The Hidden Crisis Treatenting Tech'の次のフロンティアMay 03, 2025 am 11:09 AM新薬を策定するのにかかる時間を大幅に短縮することから、より環境に優しいエネルギーを生み出すまで、企業が新境地を破る大きな機会があります。 しかし、大きな問題があります:スキルを持っている人々が深刻な不足があります
 プロトタイプ:これらの細菌は電気を生成できますMay 03, 2025 am 11:08 AM
プロトタイプ:これらの細菌は電気を生成できますMay 03, 2025 am 11:08 AM数年前、科学者は、特定の種類のバクテリアが酸素を摂取するのではなく、電気を生成することで呼吸するように見えることを発見しましたが、どのようにしたのかは謎でした。 Journal Cellに掲載された新しい研究は、これがどのように起こるかを特定しています:微生物
 AIとサイバーセキュリティ:新政権の100日間の計算May 03, 2025 am 11:07 AM
AIとサイバーセキュリティ:新政権の100日間の計算May 03, 2025 am 11:07 AM今週のRSAC 2025会議で、SNYKは「The First 100 Days:How AI、Policy&Cybersecurity Collide」というタイトルのタイムリーなパネルを開催しました。ニコール・ペルロス、元ジャーナリストとパートネ


ホットAIツール

Undresser.AI Undress
リアルなヌード写真を作成する AI 搭載アプリ

AI Clothes Remover
写真から衣服を削除するオンライン AI ツール。

Undress AI Tool
脱衣画像を無料で

Clothoff.io
AI衣類リムーバー

Video Face Swap
完全無料の AI 顔交換ツールを使用して、あらゆるビデオの顔を簡単に交換できます。

人気の記事

ホットツール

SublimeText3 中国語版
中国語版、とても使いやすい

MantisBT
Mantis は、製品の欠陥追跡を支援するために設計された、導入が簡単な Web ベースの欠陥追跡ツールです。 PHP、MySQL、Web サーバーが必要です。デモおよびホスティング サービスをチェックしてください。

MinGW - Minimalist GNU for Windows
このプロジェクトは osdn.net/projects/mingw に移行中です。引き続きそこでフォローしていただけます。 MinGW: GNU Compiler Collection (GCC) のネイティブ Windows ポートであり、ネイティブ Windows アプリケーションを構築するための自由に配布可能なインポート ライブラリとヘッダー ファイルであり、C99 機能をサポートする MSVC ランタイムの拡張機能が含まれています。すべての MinGW ソフトウェアは 64 ビット Windows プラットフォームで実行できます。

mPDF
mPDF は、UTF-8 でエンコードされた HTML から PDF ファイルを生成できる PHP ライブラリです。オリジナルの作者である Ian Back は、Web サイトから「オンザフライ」で PDF ファイルを出力し、さまざまな言語を処理するために mPDF を作成しました。 HTML2FPDF などのオリジナルのスクリプトよりも遅く、Unicode フォントを使用すると生成されるファイルが大きくなりますが、CSS スタイルなどをサポートし、多くの機能強化が施されています。 RTL (アラビア語とヘブライ語) や CJK (中国語、日本語、韓国語) を含むほぼすべての言語をサポートします。ネストされたブロックレベル要素 (P、DIV など) をサポートします。

AtomエディタMac版ダウンロード
最も人気のあるオープンソースエディター

ホットトピック
 7935
7935 15
15 1652
1652 14
14 1412
1412 52
52 1303
1303 25
25 1250
1250 29
29


