ホームページ >テクノロジー周辺機器 >AI >AI はタンパク質の「スイッチ」をゼロから設計し、タンパク質設計における驚くべき画期的な進歩である David Baker の研究が Nature に掲載
AI はタンパク質の「スイッチ」をゼロから設計し、タンパク質設計における驚くべき画期的な進歩である David Baker の研究が Nature に掲載
- 王林オリジナル
- 2024-08-20 13:41:30753ブラウズ

生活の中で、ランプをつけたり照明を調整したりするのは簡単です。しかし、生体分子機能の同様の制御を実現するシステムは複雑であり、ほとんど理解されていません。
生物学では、タンパク質の機能は複雑な方法でオンとオフが切り替えられ、アロステリック制御は重要な生物学的制御メカニズムの 1 つであり、健康な代謝と細胞シグナル伝達に不可欠です。しかし、合成タンパク質システムでアロステリーを作成することには、常に大きな課題が伴います。
最近、ワシントン大学のデビッド・ベイカーのチームは、アロステリック制御によって組み立てと分解を確実かつ正確に切り替えることができるタンパク質を設計しました。研究者らは、AI を使用して自然界に存在しない新しいタンパク質を設計し、複数の動的なタンパク質の配置を設計しました。
デビッド・ベイカー氏は、「命令に従って組み立てたり分解したりできるタンパク質を設計することで、自然の複雑さに匹敵する可能性のある将来のバイオテクノロジーへの道を切り開いている。」と論文の筆頭著者兼責任著者であるアルビンド・ピライ氏は語った。この研究の重要な革新の 1 つは、エフェクター分子に応答して、二量体、リング、ケージなどの異なるオリゴマー状態の間で切り替えることができるタンパク質集合体の設計です」と博士は述べています。適応生体材料と薬物送達システムの開発の可能性を「ネイチャー」誌は「タンパク質設計における驚くべき進歩」と呼んでいる。
関連する研究は「アロステリックに切り替え可能なタンパク質アセンブリのデノボデザイン」というタイトルで、8月14日に「Nature」に掲載されました。
アロステリーとデノボデザイン論文リンク: https://www.nature.com/articles/s41586-024-07813-2
- デザインは、特定の分子シグナルに応答して構造を変化させ、この機能を実行するタンパク質は、
- アロステリック調節因子 と呼ばれます。
De novoデザイン
- De novoデザインのタンパク質は、自然に進化した特性のレパートリーを拡大し、タンパク質機能のより制御可能な制御への扉を開きます。
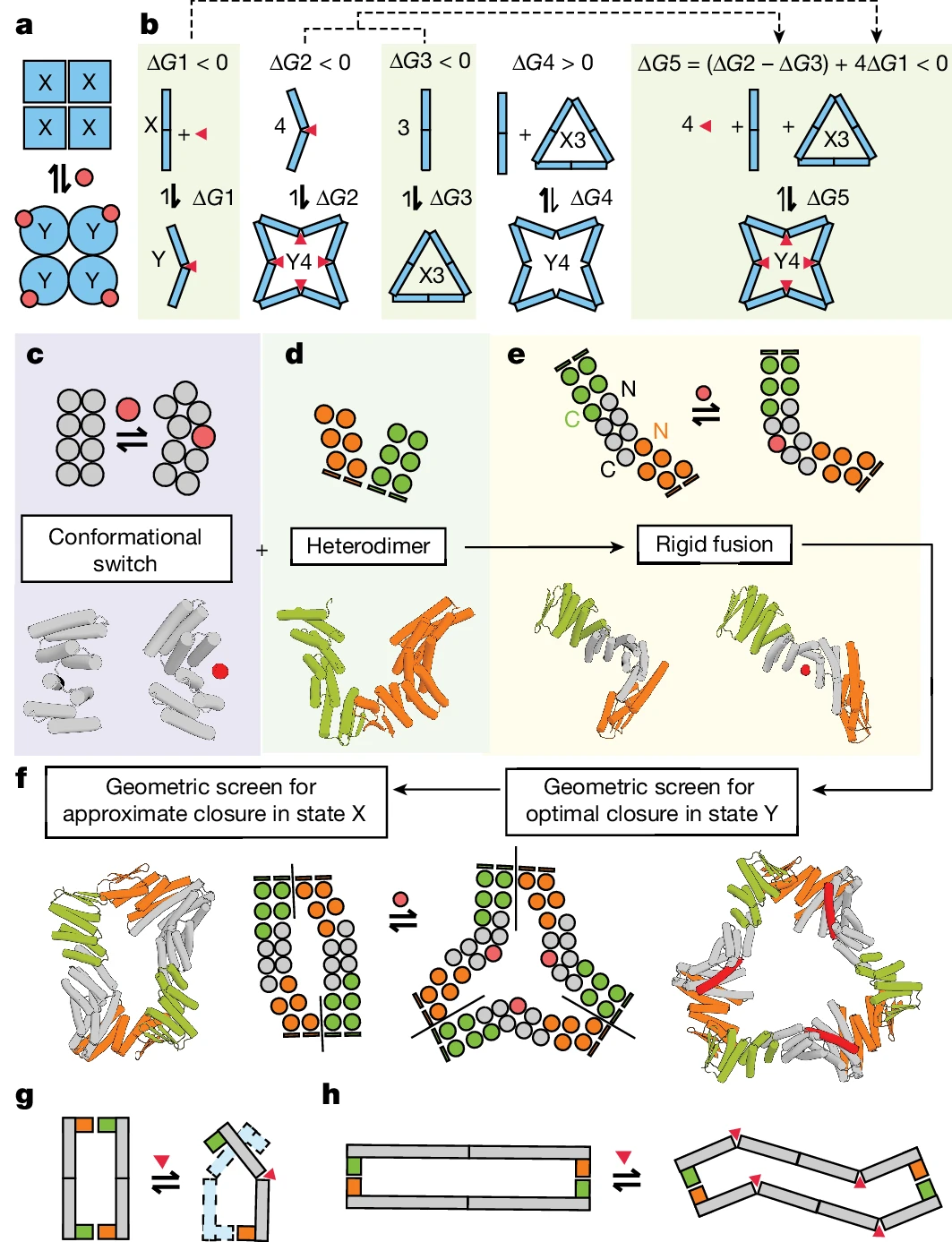
- MWC 相乗効果モデルに触発され、研究者らは、切り替え可能なヒンジ モジュールとタンパク質インターフェイスを剛体と結合することにより、
- アロステリー をゼロから設計しました。
設計戦略
- 設計の開始点としてMWCモデルを使用する
- 設計 構造スイッチ
- 構造的かつエネルギー的に実現可能な方法でタンパク質モジュールを縫製する
- イラスト:
切り替え可能なオリゴマーの設計戦略を構築します。 (出典: 論文)
研究結果:
研究者らは、RFdiffusion、ProteinMPNN、その他の設計ツールを適用して、動的で構造的に切り替え可能な一連のタンパク質集合体を作成することを実証しています。 2 状態ヒンジとカスタマイズされたタンパク質間相互作用モジュールを組み合わせることで、得られるアセンブリはこれまでに見られたものとは大きく異なり、合成生物学の可能性が広がります。
主要な革新:この研究の主要な革新は、タンパク質集合体の設計です。構造の多様性に加えて、チームは新しいタンパク質とそのエフェクターの間の高親和性結合も達成し、信頼性の高いプログラムされたアロステリック制御を確保しました。
エフェクターの選択:「このプロジェクトでは、特定のペプチドをエフェクターとして使用しましたが、適切な条件下では、どのような種類の分子でもタンパク質アロステリーを生成できます」と、共著者であるワシントン大学大学院生のアッバス・イドリス氏は述べています。追加した。
図: アロステリックに制御された周期的アセンブリの設計。 (出典: 論文)研究者らは、設計された多数のタンパク質を合成し、タンパク質の構造とエフェクター分子の結合によって引き起こされるスイッチング動作を特徴付けました。さまざまな数のプロトマーから構成されるリング集合体間を切り替えるように設計された合成タンパク質のほぼ 40% は水溶性であり、予想されるプロトマー化学量論を示します。 
Um noch einen Schritt weiter zu gehen, entwarfen die Forscher Proteine, die Doppelscharniere (zwei Scharniere, die durch kurze Schleifen verbunden sind) enthalten, mit dem Ziel, Strukturen zu schaffen, die auf die Bindung von Effektoren reagieren, ohne die Anzahl der Protomer in der Proteinanordnung zu verändern Das Protein verändert seine 3D-Struktur. Tatsächlich funktionierten diese Proteine wie erwartet und reproduzierten das dominante Verhalten natürlich vorkommender allosterischer Proteine wie Hämoglobin. Schließlich entwarfen die Forscher auch Protomere, die sich zusammensetzen oder zerlegen, wenn sie an Effektormoleküle gebunden sind.
Spezifische De-novo-Proteinanordnungen, die in der Studie entworfen wurden, umfassen Ringe, die durch die Dimerisierung zweier Monomere gebildet werden und beim Zusammenbau eine Lichtabgabe für Biosensoranwendungen auslösen, sowie käfigartige Strukturen, die einer kontrollierten Zerlegung unterliegen, um die Nutzlast für die Arzneimittelabgabe freizugeben. Diese Proteindynamik wurde experimentell in vitro durch Größenausschlusschromatographie, Massenspektrometrie und Elektronenmikroskopie verifiziert.
Pillai betonte, dass Ringstrukturen zusätzliche präzise Eigenschaften wie Kooperativität aufweisen, ein Phänomen, das natürliche Systeme (z. B. Blutproteine, Hämoglobin) aufweisen. In synergistischen Systemen verstärkt die Bindung eines Moleküls die Bindung anderer Moleküle und führt zu schnellen Ein-Aus-Reaktionen, die für eine präzise Steuerung entscheidend sind, beispielsweise das Einfangen von Sauerstoff in der Lunge und seine Freisetzung in das Gewebe.
„In der Vergangenheit haben wir im Labor viel getan, um die Bindungsaffinität einer Substanz zu kontrollieren, beispielsweise indem wir sie immer fester gebunden haben. Aber das ist nicht der einzige Aspekt, der für biologische Systeme relevant ist“, sagte Pillai. „Manchmal möchte man in der Lage sein, über einen sehr engen Konzentrationsbereich zu binden.“ Um das Design zu validieren, charakterisierten die Forscher mehr als 20 Proteinanordnungen mittels Negativfärbung und Kryo-Elektronenmikroskopie. „Dadurch konnten wir bestätigen, welche Designs sich wie erwartet bildeten, und beobachten, wie diese Anordnungen ihre Struktur veränderten, als Effektormoleküle eingeführt wurden“, erklärt Dr. Andrew Borst, Leiter des IPD Electron Microscopy Research Core.
Abbildung: Strukturcharakterisierung von sr312 und sr322 durch Elektronenmikroskopie. (Quelle: Papier) 
Zu den entworfenen Komponenten gehören nanoskalige Behälter, die aus der Ferne geöffnet und geschlossen werden können. Solche Systeme könnten zu neuartigen Medikamentenverabreichungsvehikeln mit fortschrittlichen Kontrollmechanismen führen, einschließlich Geräten, die zelltötende Medikamente binden, bis sie auf Tumore treffen.
Diese Forschung ebnet den Weg für die Gestaltung allosterisch kontrollierter Funktionen, die über den Auf- und Abbau von Proteinen hinausgehen, wie etwa die Regulierung der Enzymaktivität für Stoffwechselfunktionen und Nanomaschinen, die Energie in mechanische Arbeit umwandeln können, ähnlich den Proteinen, die für die Zellbewegung verantwortlich sind .
„Der nächste Schritt besteht darin, festzustellen, ob wir Wechselwirkungen mit kleinen Molekülen eingehen und Reaktionen präzise katalysieren können, was eine größere Herausforderung für das gesamte Gebiet darstellt“, sagte Pillai.
Zukünftig möchte das Forschungsteam die Dynamik dieser manipulierten Proteine in einem breiteren biologischen Kontext bewerten. Zukünftige Arbeiten umfassen die Installation dieser technischen Funktionen auf Zelloberflächen in Gewebekulturen, um wertvolle Werkzeuge für die Feedback-Kontrolle bei Therapeutika wie der adoptiven Zelltherapie bereitzustellen.
Referenzinhalt:
https://www.bakerlab.org/2024/08/14/morphing-protein-assemblies-by-design/- https://www.genengnews.com/topics/artificial- Intelligenz/ai-designed-proteins-morph-on-demand-for-steerable-functionality/
- https://www.nature.com/articles/d41586-024-02242-7
以上がAI はタンパク質の「スイッチ」をゼロから設計し、タンパク質設計における驚くべき画期的な進歩である David Baker の研究が Nature に掲載の詳細内容です。詳細については、PHP 中国語 Web サイトの他の関連記事を参照してください。