ホームページ >テクノロジー周辺機器 >AI >タンパク質と DNA の結合特異性を予測するために、USC チームが新しい幾何学的ディープラーニング手法を開発
タンパク質と DNA の結合特異性を予測するために、USC チームが新しい幾何学的ディープラーニング手法を開発
- PHPzオリジナル
- 2024-08-19 13:34:101316ブラウズ
編集者 | 大根の皮
タンパク質と DNA の結合特異性を予測することは、遺伝子制御を理解するのに役立つ、困難ですが重要な作業です。タンパク質-DNA複合体は通常、選択されたDNA標的に結合しますが、タンパク質はさまざまな程度の結合特異性で広範囲のDNA配列に結合します。この情報は、単一の構造内では直接アクセスできません。
この情報を得るために、南カリフォルニア大学とワシントン大学の研究者は、タンパク質-DNA 構造に基づいて結合特異性を予測するように設計された幾何学的な深層学習モデルである Deep Binding Specificity Predictor (DeepPBS) を提案しました。
DeepPBS は、界面残基の解釈可能なタンパク質重原子重要度スコアを抽出できます。これらのスコアは、タンパク質残基レベルで集計した場合の突然変異誘発実験によって検証されました。 DeepPBS を特定の DNA 配列をターゲットとする設計されたタンパク質に適用すると、実験的に測定された結合特異性を予測できることが示されました。
この研究は「タンパク質-DNA結合特異性の幾何学的深層学習」と題され、2024年8月5日に「Nature Methods」に掲載されました。

転写因子は、特定の DNA 配列に結合することによって生命プロセスを制御します。この結合メカニズムには、静電相互作用、デオキシリボースのスタッキング効果、および水素結合の形成が含まれます。
タンパク質-DNA の構造情報は通常、X 線結晶構造解析、核磁気共鳴分光法、極低温電子顕微鏡などの実験的方法を通じて取得され、タンパク質データ バンク (PDB) に保存されます。これらの構造は通常、結合した DNA 配列とその物理化学的相互作用を示しますが、考えられるすべての結合配列を網羅しているわけではありません。
一方、タンパク質結合マイクロアレイやSELEX-seqなどのハイスループット実験では、潜在的な結合配列の範囲を捕捉できますが、構造情報が不足しています。
したがって、転写因子の結合特異性を完全に理解するには、構造データとハイスループット実験データを組み合わせることが重要です。
現在、タンパク質ファミリー内の特定のタンパク質配列の結合特異性を予測することは、依然として困難で未解決の問題です。この困難は、結合コンテキストの構造変化と広大な機構の多様性によってさらに悪化します。
「タンパク質-DNA複合体の構造には、通常単一のDNA配列に結合するタンパク質が含まれています。遺伝子制御を理解するには、任意のDNA配列またはゲノム領域に対するタンパク質の結合特異性を理解することが重要です。」南カリフォルニア大学のレモ・ローズ教授。
最新の研究では、南カリフォルニア大学とワシントン大学の研究者が結合特異性ディーププレディクター (DeepPBS) を導入しました。
Rohs 氏は次のように説明しました。「DeepPBS は、タンパク質と DNA の結合特異性を明らかにするためのハイスループット シーケンスや構造生物学の実験に代わる人工知能ツールです。」 図: DeepPBS フレームワークの概略図。 (出典: 論文)
この深層学習モデルは、タンパク質-DNA 相互作用の物理化学的および幾何学的コンテキストを捕捉して、特定のタンパク質-DNA 構造に基づいて位置重み行列 (PWM) として表現される結合特異性を予測することを目的としています。 DeepPBS はタンパク質ファミリー全体で機能し、構造決定と結合特異性決定実験の間の橋渡しとして機能します。 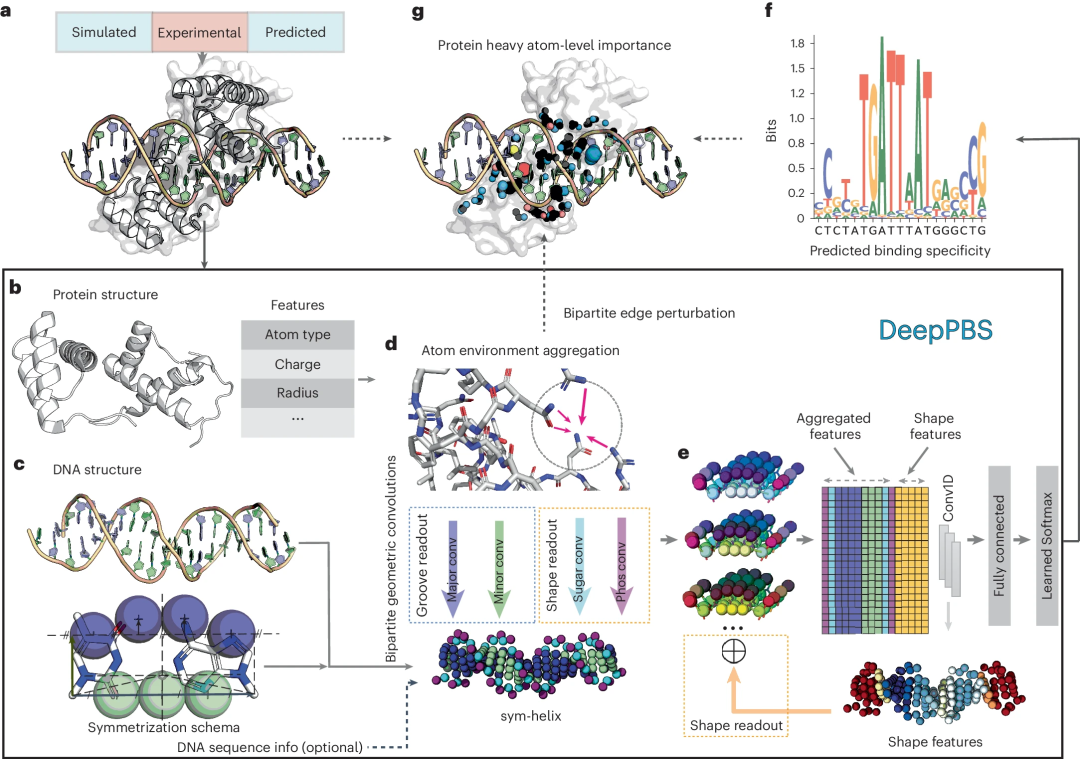
図: タンパク質ファミリー間の結合特異性を予測するための DeepPBS のパフォーマンス。 (出典: 論文)
DeepPBS への入力は実験構造に限定されません。 AlphaFold、OpenFold、RoseTTAFold などのタンパク質構造予測手法や、RoseTTAFoldNA (RFNA)、RoseTTAFold All-Atom、MELD-DNA、AlphaFold3 などのタンパク質-DNA 複合モデラーの急速な開発により、構造データが利用できるようになりました。分析 その数は指数関数的に増加しています。 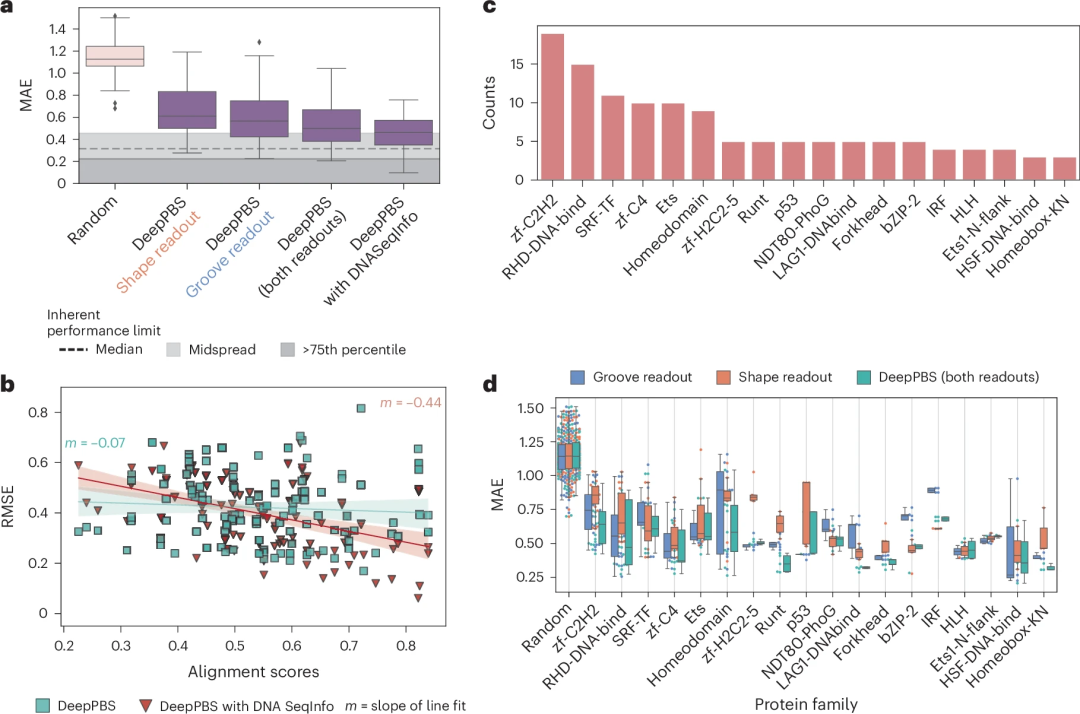
図: タンパク質-DNA 複合体の構造予測における DeepPBS の応用。 (出典: 論文)
解釈可能性の観点からは、DNA と相互作用するタンパク質内のさまざまな重原子の「相対重要度」(RI) スコアを DeepPBS から抽出できます。
がんの発生に重要なタンパク質のケーススタディとして、研究者らはこれらの RI スコアを通じて p53-DNA インターフェースを分析し、検証のために既存の文献にリンクしました。
そして、DeepPBS スコアは既存の知識とよく一致しており、アラニン スキャニング突然変異誘発実験と合理的な一致をもたらすように集計できます。
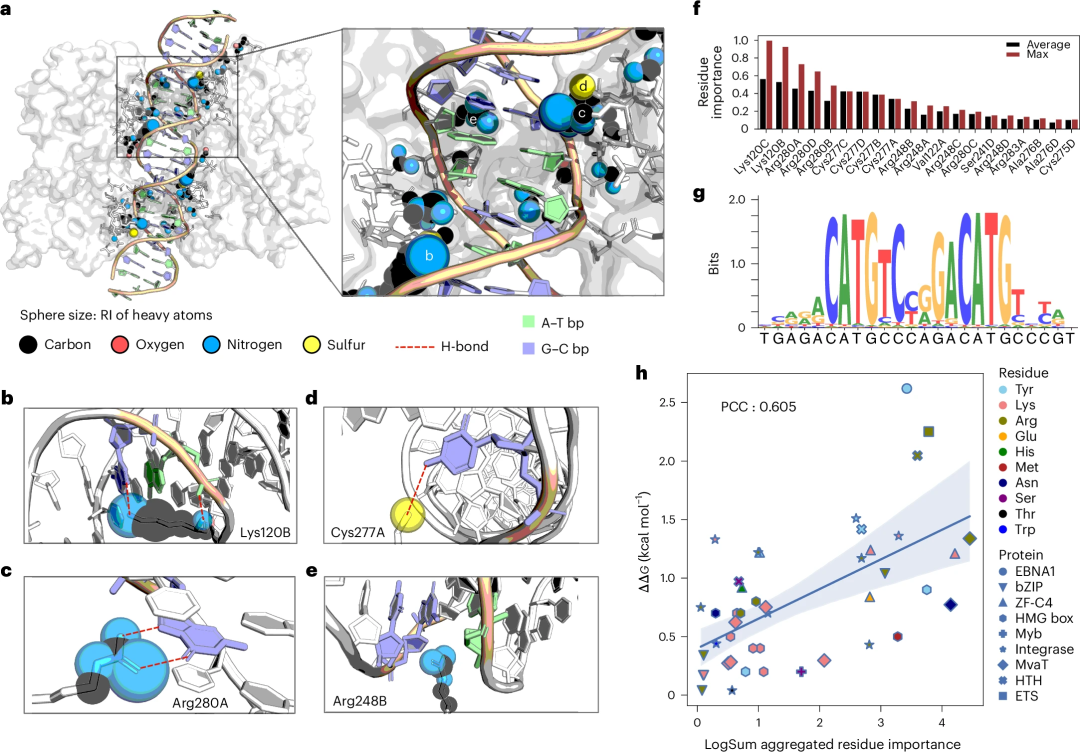
図: 研究および実験的検証を行うための例として、p53-DNA インターフェースの DeepPBS 重要度スコアの視覚化を取り上げます。 (出典: 論文)
追加の原理実証研究では、研究者らは、構造設計と DNA 突然変異誘発研究を組み合わせた最近の実験から得られた、特定の DNA 配列をターゲットとするインシリコ設計のタンパク質-DNA 複合体に DeepPBS を適用しました。 DeepPBS は、分子シミュレーションの軌跡の分析にも使用できます。
「研究者にとって、よく研究されているタンパク質ファミリーに限定されず、すべてのタンパク質に有効な方法を見つけることが重要です。この方法により、新しいタンパク質を設計することも可能になります。」と Rohs 氏は言いました。
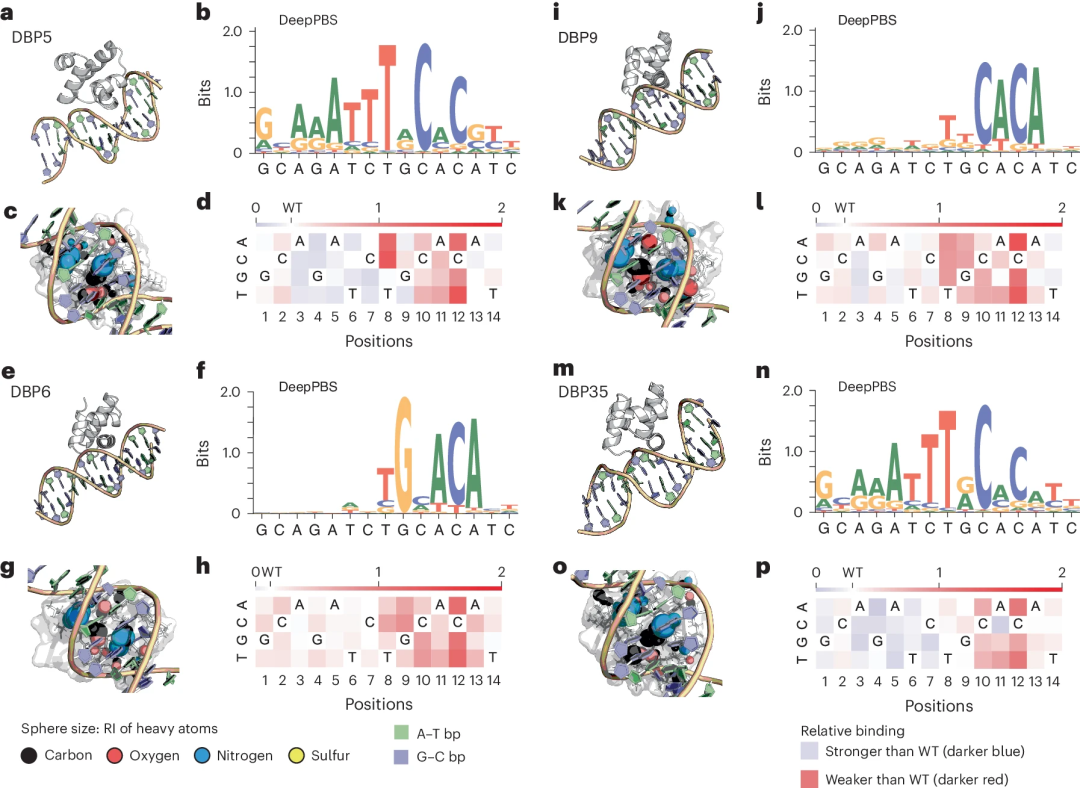
図: 特定の DNA 配列をターゲットとするコンピュータで設計された HTH スキャフォールドに DeepPBS を適用する。 (出典: 論文)
DeepPBS の現在のバージョンには固有の制限があります。二本鎖 DNA 用に調整されていますが、一本鎖 DNA、RNA、または化学修飾された塩基にはまだ機能しません。
ただし、このモデルは、これらのさまざまなシナリオや他のポリマー間相互作用、さらには機構的な突然変異にも対応できるように拡張される可能性があります。 DeepPBS アーキテクチャは、アプリケーションとエンジニアリングの強化に関して最適化および拡張できます。
それにもかかわらず、Rohs 氏は、DeepPBS には幅広い用途があるだろうと述べました。この新しい研究アプローチは、がん細胞の特定の変異を標的とする新薬や治療法の設計を加速するとともに、合成生物学における新たな発見やRNA研究への応用につながる可能性があります。
DeepPBS: https://deeppbs.usc.edu
以上がタンパク質と DNA の結合特異性を予測するために、USC チームが新しい幾何学的ディープラーニング手法を開発の詳細内容です。詳細については、PHP 中国語 Web サイトの他の関連記事を参照してください。

