 編集者 | KX
編集者 | KX
AIテクノロジーは抗体設計の支援において大きな進歩を遂げました。しかし、抗体設計は依然として血清からの抗原特異的抗体の単離に大きく依存しており、これはリソースを大量に消費し、時間のかかるプロセスです。
この問題を解決するために、Tencent AI Lab、北京大学深セン大学院、西京消化器病病院の研究チームは、必要な抗原結合特異性を実現する独自の人工抗体 CDRH3 は、天然抗体への依存を軽減します。
さらに、高精度の抗原抗体結合予測モデル A2binder は、抗原エピトープ配列と抗体配列を照合して結合特異性と親和性を予測するように設計されました。
要約すると、この研究は抗体生成と評価のための人工知能フレームワークを確立し、抗体医薬品の開発を大幅に加速する可能性があります。
関連研究「事前学習済み生成大規模言語モデルによるSARS-CoV-2抗体CDRH3のDe novo生成」が、8月10日付けの「Nature Communications」に掲載されました。
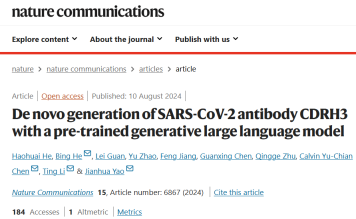
モノクローナル抗体としても知られる抗体医薬品は、生物学的療法の効果において重要な役割を果たします。これらの薬剤は、免疫系の作用を模倣することで、ウイルスやがん細胞などの病気の原因物質を選択的に標的にすることができます。抗体医薬は、従来の治療法よりも特異的かつ効果的なアプローチです。抗体医薬はさまざまな病気の治療において良い結果を示しています。
抗体医薬品の開発は、動物源からの抗体の単離、ヒト化、その親和性の最適化を含む複雑なプロセスです。しかし、抗体医薬品の開発は依然として天然抗体に大きく依存しています。
タンパク質の配列データは言語として見ることができるため、自然言語処理 (NLP) の分野における大規模な事前トレーニング済みモデルがタンパク質の表現パターンを学習するために使用されてきました。さまざまなタンパク質言語モデルが開発されています。しかし、抗体の多様性が高く、入手可能な抗原抗体対データが不足しているため、特定のエピトープに対して高い親和性を持つ抗体を生成することは依然として困難な課題です。
上記の課題に対処するために、Tencent AI Lab チームは、重要な役割を果たす重鎖相補性決定領域 3 (CDRH3) を最適化して生成するための、事前トレーニング済み抗体生成大規模言語モデル PALM-H3 を提案しました。抗体の特異性と多様性において重要な役割を果たします。
PALM-H3 によって生成された抗体の抗原に対する親和性を評価するために、研究者らは抗原抗体ドッキングと AI ベースの方法を組み合わせて使用しました。
研究者らは、抗体抗原親和性を評価するための A2binder も開発しました。 A2binder は、未知の抗原であっても、正確かつ一般化可能な親和性予測を可能にします。
PALM-H3とA2Binderのフレームワーク
PALM-H3とA2binderのワークフローとモデルフレームワークを下図に示します。
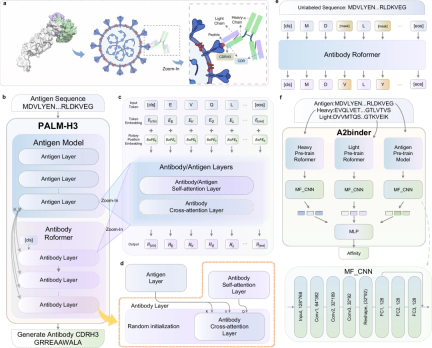
PALM-H3 は、抗体で新たに CDRH3 配列を生成するように設計されています。 CDRH3 領域は、特定の抗原配列に対する抗体の結合特異性を決定する際に最も重要な役割を果たします。 PALM-H3 は、ESM2 ベースの抗原モデルをエンコーダーとして、抗体 Roformer をデコーダーとして使用するトランスフォーマーのようなモデルです。この研究では、人工的に生成された抗体の結合親和性を予測するための A2binder も構築されました。
PALM-H3 と A2binder の構築には 3 つのステップが含まれていました: まず、研究者らは、不対抗体重鎖配列と軽鎖配列でそれぞれ 2 つの Roformer モデルを事前トレーニングしました。次に、事前トレーニング済みの ESM2、抗体重鎖 Roformer、および抗体軽鎖 Roformer に基づいて A2binder を構築し、ペアのアフィニティー データを使用してトレーニングしました。最後に、事前トレーニングされた ESM2 と抗体重鎖 Roformer を使用して PALM-H3 を構築し、対の抗原 CDRH3 データでトレーニングして CDRH3 を新たに生成しました。
A2binder は抗原抗体結合確率、親和性を正確に予測できます
A2binder のパフォーマンスは、親和性を予測する能力をいくつかのベースライン方法と比較することによって評価されました。
A2binder は、抗体配列の事前トレーニングにより、アフィニティー データセットで良好なパフォーマンスを発揮します。これにより、A2binder は、これらの配列に存在する固有のパターンを学習できるようになります。
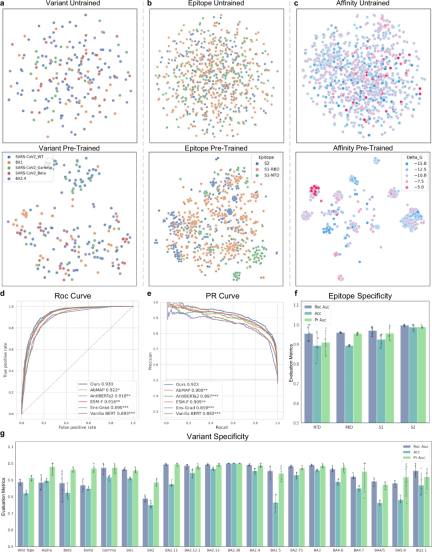
Les résultats montrent qu'A2binder fonctionne mieux que le modèle de base ESM-F sur tous les ensembles de données de prédiction d'affinité antigène-anticorps (ce dernier a le même cadre, mais le modèle pré-entraîné est la substitution ESM2), ce qui suggère que le pré-entraînement avec des séquences d'anticorps peut être bénéfique pour les tâches connexes en aval.
Pour évaluer les performances du modèle dans la prédiction des valeurs d'affinité, les chercheurs ont également utilisé deux ensembles de données, 14H et 14L, qui contiennent des étiquettes de valeurs d'affinité.
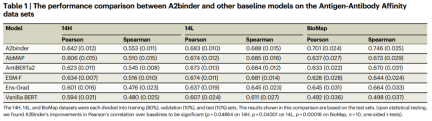
A2binder surpasse tous les modèles de base sur les mesures de corrélation de Pearson et de corrélation de Spearman. A2binder atteint une corrélation de Pearson de 0,642 sur l'ensemble de données 14H (une amélioration de 3 %) et de 0,683 sur l'ensemble de données 14L (une amélioration de 1 %).
Cependant, les performances d'A2binder et d'autres modèles de base ont légèrement diminué sur les ensembles de données 14H et 14L par rapport aux autres ensembles de données. Cette observation est cohérente avec les études précédentes.
PALM-H3 excelle dans la génération d'anticorps à forte probabilité de liaison
Les chercheurs ont exploré la différence entre les anticorps produits par PALM-H3 et les anticorps naturels anticorps. différence entre. Leurs séquences différaient de manière significative, mais les probabilités de liaison des anticorps produits n’étaient pas significativement affectées par ces différences. Dans le même temps, leurs différences structurelles conduisent à une diminution de l’affinité de liaison. Ces résultats sont cohérents avec des études antérieures sur l’analyse de réseau de bibliothèques d’anticorps et la génération de séquences protéiques fonctionnelles.

Dans l'ensemble, les résultats montrent que PALM-H3 est capable de générer une gamme diversifiée de séquences d'anticorps avec des affinités de liaison élevées, bien que contrairement aux anticorps naturels.
De plus, les chercheurs ont vérifié les performances du PALM-H3 via ClusPro et SnugDock. PALM-H3 est capable de générer des anticorps contre la séquence CDRH3 du peptide stabilisant la région HR2 du SRAS-CoV-2. Il a généré une nouvelle séquence CDRH3 et a validé que la séquence générée GRREAAWALA avait amélioré le ciblage des peptides stabilisant l'antigène par rapport à la séquence CDHR3 native GKAAGTFDS.
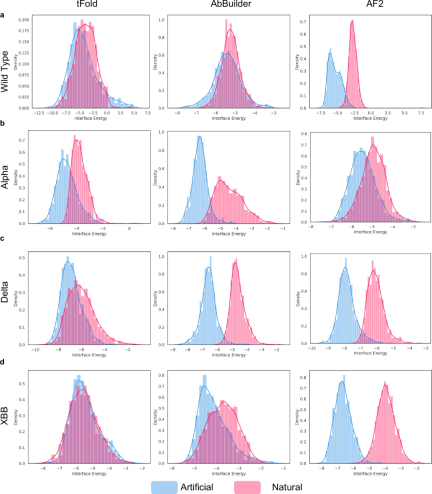
De plus, PALM-H3 est capable de générer des anticorps avec une affinité plus élevée contre la séquence émergente XBB CDRH3 du variant SARS-CoV-2. La séquence résultante AKDSRTSPLRLDYS a une affinité plus forte pour XBB que sa source, ASEVLDNLRDGYNF.
De plus, PALM-H3 surmonte non seulement les pièges optimaux locaux auxquels sont confrontées les stratégies traditionnelles de mutation séquentielle, mais il génère également des anticorps avec une affinité de liaison à l'antigène plus élevée que l'approche E-EVO. Ceci met en valeur les avantages de PALM-H3 dans la conception d’anticorps, permettant une exploration plus efficace de l’espace des séquences et la génération de liants de haute affinité ciblant des épitopes spécifiques.
Expériences in vitro
En outre, les chercheurs ont également mené des expériences in vitro, notamment le Western blot, l'analyse par résonance plasmonique de surface et la neutralisation des pseudovirus. L’expérience a fourni une vérification clé de l’efficacité de l’anticorps conçu par PALM-H3.
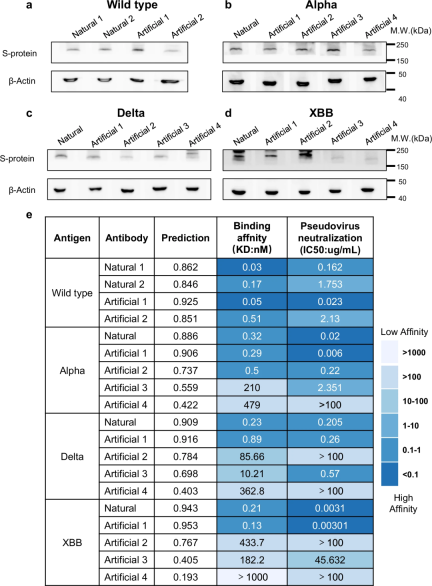
PALM-H3 Deux anticorps générés contre les protéines de pointe des variantes de type sauvage du SRAS-CoV-2, Alpha, Delta et XBB, ont atteint des niveaux plus élevés que les niveaux natifs dans ces essais. Une affinité de liaison plus élevée et pouvoir neutralisant des anticorps. Les résultats empiriques robustes de ces expériences en laboratoire humide complètent les prédictions et analyses informatiques, validant la capacité de PALM-H3 et A2binder à générer et à sélectionner des anticorps puissants avec une spécificité et une affinité élevées pour les antigènes connus et nouveaux.
En résumé, le PALM-H3 proposé intègre la capacité de pré-entraînement d'anticorps à grande échelle et l'efficacité de la fusion globale de caractéristiques, ce qui se traduit par d'excellentes performances de prédiction d'affinité et la capacité de concevoir des anticorps de haute affinité . De plus, la génération directe de séquences et la visualisation interprétable du poids en font un outil efficace et interprétable pour concevoir des anticorps de haute affinité.
以上がテンセントと北京大学のチームは抗体をゼロから設計し、大規模な言語モデルを事前トレーニングし、Nature サブジャーナルに発表しました。の詳細内容です。詳細については、PHP 中国語 Web サイトの他の関連記事を参照してください。
 メタの新しいAIアシスタント:生産性ブースターまたはタイムシンク?May 01, 2025 am 11:18 AM
メタの新しいAIアシスタント:生産性ブースターまたはタイムシンク?May 01, 2025 am 11:18 AMMetaは、NVIDIA、IBM、Dellなどのパートナーと協力して、Llama Stackのエンタープライズレベルの展開統合を拡大しました。セキュリティの観点から、MetaはLlama Guard 4、Llamafirewall、Cyberseceval 4などの新しいツールを立ち上げ、AIセキュリティを強化するためにLlama Defendersプログラムを開始しました。さらに、METAは、公共サービス、ヘルスケア、教育の改善に取り組んでいる新興企業を含む、Llama Impact Grantsの150万ドルを10のグローバル機関に分配しています。 Llama 4を搭載した新しいメタAIアプリケーションは、メタAIとして考案されました
 Gen Zersの80%はAI:研究と結婚しますMay 01, 2025 am 11:17 AM
Gen Zersの80%はAI:研究と結婚しますMay 01, 2025 am 11:17 AM人間との相互作用の先駆者であるJoi Aiは、これらの進化する関係を説明するために「AI-lationships」という用語を導入しました。 Joi Aiの関係療法士であるJaime Bronsteinは、これらが人間cを置き換えることを意図していないことを明確にしています
 AIはインターネットのボット問題を悪化させています。この20億ドルのスタートアップは最前線にありますMay 01, 2025 am 11:16 AM
AIはインターネットのボット問題を悪化させています。この20億ドルのスタートアップは最前線にありますMay 01, 2025 am 11:16 AMオンライン詐欺とボット攻撃は、企業にとって大きな課題をもたらします。 小売業者は、ボットの買いだめ製品、銀行の戦闘口座の買収、ソーシャルメディアプラットフォームと戦い、なりすまし者と闘っています。 AIの台頭は、この問題を悪化させます
 ロボットへの販売:あなたのビジネスを作ったり壊したりするマーケティング革命May 01, 2025 am 11:15 AM
ロボットへの販売:あなたのビジネスを作ったり壊したりするマーケティング革命May 01, 2025 am 11:15 AMAIエージェントは、マーケティングに革命をもたらす態勢が整っており、以前の技術的変化の影響を上回る可能性があります。 これらのエージェントは、生成AIの大幅な進歩を表し、ChatGPTのような情報を処理するだけでなく、Actioも取る
 コンピュータービジョンテクノロジーがどのようにNBAプレーオフを司会しているかMay 01, 2025 am 11:14 AM
コンピュータービジョンテクノロジーがどのようにNBAプレーオフを司会しているかMay 01, 2025 am 11:14 AM重要なNBAゲーム4の決定に対するAIの影響 2つの重要なゲーム4 NBAマッチアップは、司会におけるAIのゲームを変える役割を紹介しました。 最初に、デンバーのニコラ・ジョキッチの逃した3ポインターは、アーロン・ゴードンの最後の2秒の路地につながりました。 ソニーのホー
 AIがどのように再生医療の未来を加速しているかMay 01, 2025 am 11:13 AM
AIがどのように再生医療の未来を加速しているかMay 01, 2025 am 11:13 AM伝統的に、再生医療の専門知識を拡大すると、世界的に大規模な旅行、実践的なトレーニング、長年のメンターシップが必要でした。 現在、AIはこの風景を変えており、地理的な制限を克服し、ENを通じて進歩を加速しています
 Intel Foundry Direct Connect 2025からのキーテイクアウトMay 01, 2025 am 11:12 AM
Intel Foundry Direct Connect 2025からのキーテイクアウトMay 01, 2025 am 11:12 AMIntelは、製造プロセスを主要な位置に戻すように取り組んでいますが、Fab Semiconductorの顧客を引き付けてFabでチップを作成しようとしています。この目的のために、Intelは、そのプロセスの競争力を証明するだけでなく、パートナーが馴染みのある成熟したワークフローでチップを製造できることを実証するために、業界へのより多くの信頼を築かなければなりません。今日私が聞いたことはすべて、インテルがこの目標に向かっていると信じています。 新しいCEOのタンリバイの基調講演がその日をキックオフしました。タンリバイは簡単で簡潔です。彼は、IntelのFoundry Servicesにおけるいくつかの課題と、これらの課題に対処し、将来のIntelのFoundry Servicesの成功したルートを計画するために企業が行った対策を概説しています。 Tan Libaiは、IntelのOEMサービスが顧客をより多くするために実装されているプロセスについて話しました
 AIが間違っていましたか?今、そのための保険がありますMay 01, 2025 am 11:11 AM
AIが間違っていましたか?今、そのための保険がありますMay 01, 2025 am 11:11 AMAIのリスクを取り巻く増大する懸念に対処するために、グローバルな専門家保険会社であるChaucer GroupとArmilla AIは、新しいサードパーティの責任(TPL)保険商品を導入するために力を合わせました。 このポリシーは、企業を守ります


ホットAIツール

Undresser.AI Undress
リアルなヌード写真を作成する AI 搭載アプリ

AI Clothes Remover
写真から衣服を削除するオンライン AI ツール。

Undress AI Tool
脱衣画像を無料で

Clothoff.io
AI衣類リムーバー

Video Face Swap
完全無料の AI 顔交換ツールを使用して、あらゆるビデオの顔を簡単に交換できます。

人気の記事

ホットツール

SublimeText3 英語版
推奨: Win バージョン、コードプロンプトをサポート!

ZendStudio 13.5.1 Mac
強力な PHP 統合開発環境

MantisBT
Mantis は、製品の欠陥追跡を支援するために設計された、導入が簡単な Web ベースの欠陥追跡ツールです。 PHP、MySQL、Web サーバーが必要です。デモおよびホスティング サービスをチェックしてください。

ドリームウィーバー CS6
ビジュアル Web 開発ツール

mPDF
mPDF は、UTF-8 でエンコードされた HTML から PDF ファイルを生成できる PHP ライブラリです。オリジナルの作者である Ian Back は、Web サイトから「オンザフライ」で PDF ファイルを出力し、さまざまな言語を処理するために mPDF を作成しました。 HTML2FPDF などのオリジナルのスクリプトよりも遅く、Unicode フォントを使用すると生成されるファイルが大きくなりますが、CSS スタイルなどをサポートし、多くの機能強化が施されています。 RTL (アラビア語とヘブライ語) や CJK (中国語、日本語、韓国語) を含むほぼすべての言語をサポートします。ネストされたブロックレベル要素 (P、DIV など) をサポートします。

ホットトピック
 7885
7885 15
15 1649
1649 14
14 1410
1410 52
52 1301
1301 25
25 1246
1246 29
29



