Nature サブジャーナル、北京大学チームの一般的な AI フレームワークは、実験と計算の間のギャップを埋める、タンパク質間ドッキングの包括的な構造予測を実施
編集者 | 大根の皮
タンパク質複合体構造の予測は、医薬品開発、抗体設計、その他の応用において重要な役割を果たしますが、予測精度が限られているため、予測結果が実験結果と一致しないことがよくあります。
北京大学、昌平研究所、ハーバード大学の研究チームは、深層学習構造予測モデルを採用し、さらなる大規模な再トレーニングや微調整を行わずに、さまざまな形式やソースの実験的制約を統合する一般的なフレームワークである ColabDock を提案しました。
ColabDock は、AlphaFold2 を構造予測モデルとして使用し、シミュレートされた残基と表面制約による複雑な構造予測だけでなく、この方法での NMR 化学シフト摂動や共有結合標識による構造予測でも、HADDOCK や ClusPro よりも優れています。
また、界面スキャンの制限をシミュレートすることで、抗体-抗原界面の予測にも役立ちます。
この研究は「ColabDockを使用した実験的拘束によるタンパク質間ドッキングの統合構造予測」と題され、2024年8月5日に「Nature Machine Intelligence」に掲載されました。

タンパク質のドッキングは、生物学的メカニズムを理解するための重要な構造情報を提供します。タンパク質構造予測ではディープモデルが急速に発展していますが、ほとんどのモデルはフリードッキング方式で予測を実行するため、実験上の制約と予測された構造の間に不一致が生じる可能性があります。この問題を解決するために、北京大学、昌平研究所、その他の機関の研究チームは、制約付き複雑構造予測のための一般的なフレームワークである ColabDock を提案しました。これは、まばらな実験的制約フレームによってガイドされる一般的なタンパク質間ドッキングです。
この方法は、勾配逆伝播を通じて、実験的に制約された事前分布とデータ駆動型タンパク質構造予測モデルのエネルギーランドスケープを効果的に統合し、制約の矛盾や曖昧さを許容しながら両方を満たす立体構造を自動的に検索します。
ColabDock は、さらに広範な再トレーニングや微調整を行わずに、実験上の制約のさまざまな形式やソースを活用できます。
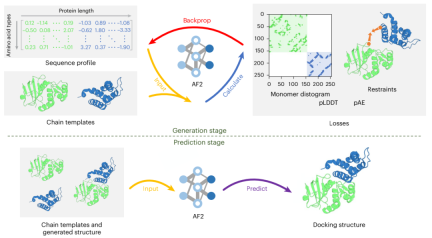 イラスト: ColabDock のワークフロー。 (出典: 論文)
イラスト: ColabDock のワークフロー。 (出典: 論文)
生成段階では、ColabDock は AlphaFold2 に基づいて開発されたタンパク質設計フレームワークである ColabDesign を使用します。入力配列プロファイルはロジット空間で最適化され、pLDDT および pAE 測定を最大化しながら、特定の実験的制約とテンプレートに基づいて複雑な構造を生成する構造予測モデルを導きます。
予測フェーズでは、生成された複雑な構造と指定されたテンプレートに基づいて構造が予測されます。 ColabDock はターゲットごとに複数の実行を実行し、異なる立体構造を生成します。最終的な立体構造は、ランク付けされたサポート ベクター マシン (SVM) アルゴリズムによって選択されました。
堅牢なパフォーマンス概念実証として、研究者らは、ColabDock の構造予測モデルとして AlphaFold2 を採用しました。もちろん、RoseTTAFold2 や AF-Multimer など、他のデータ駆動型深層学習モデルもここで使用できます。
研究者らは、合成データセットと、NMR 化学シフト摂動 (CSP)、共有結合標識 (CL)、シミュレートされた深部変異スキャン (DMS) などの数種類の実験制約で ColabDock をテストしました。
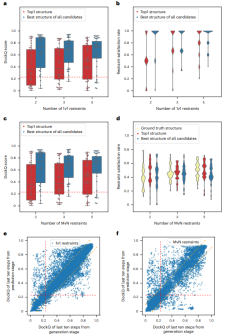 イラスト: 検証セットでの ColabDock のパフォーマンス。 (出典: 論文)
イラスト: 検証セットでの ColabDock のパフォーマンス。 (出典: 論文)
ColabDock は、1v1 制約と MvN 制約という 2 種類の制約を評価します。前者は残基間レベルであり、例には XL-MS からの制約が含まれます。後者は界面レベルにあり、NMR および CL 実験に関連します。
合成データセットでのテスト結果は、ColabDock が満足のいくパフォーマンスを達成していることを示しています。さらに、予想通り、制約の数が増えると ColabDock のパフォーマンスが向上します。
制約がほとんどない場合でも、ColabDock はベンチマーク データセットと同じフレーム設定で AF-Multimer よりも優れたパフォーマンスを示し、より多くの制約が提供されるとより少ないコンフォメーションに収束し、追加情報の効果的な適用を実証します。
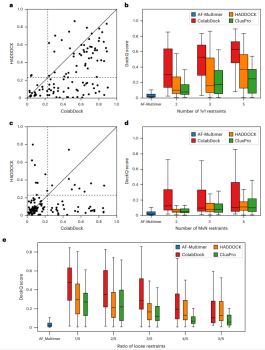 図: ColabDock、HADDOCK、ClusPro をベンチマーク セットで比較。 (出典: 論文)
図: ColabDock、HADDOCK、ClusPro をベンチマーク セットで比較。 (出典: 論文)
HADDOCK 및 ClusPro와 비교하여 ColabDock은 제약 조건 품질이 높을 때 더 나은 성능을 발휘합니다. 두 실험 데이터 세트 모두에서 ColabDock은 제공된 제약 조건의 수와 품질에 관계없이 여전히 HADDOCK과 ClusPro보다 성능이 뛰어납니다.
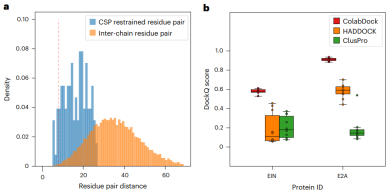
그림: CSP 세트에서 ColabDock의 성능 및 제약 분석. (출처: 논문)
마지막으로 연구원들은 항체-항원 데이터 세트에 대한 다양한 도킹 방법의 성능을 평가했습니다. ColabDock은 HADDOCK 및 ClusPro보다 중간 품질 이상의 구조 비율이 훨씬 더 높다고 예측했습니다.
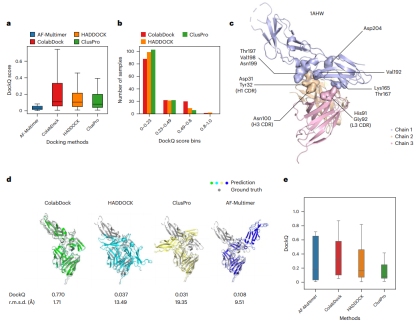
그림: 항체-항원 벤치마크 세트에 대한 ColabDock, HADDOCK 및 ClusPro 비교. (출처: 논문)
이는 ColabDock이 항체 디자인 분야에서 잠재적인 활용 가치를 가지고 있음을 보여줍니다. 또한 ColabDock은 새로 출시된 편견 없는 데이터 세트에서 AF-Multimer와 비슷하거나 훨씬 더 나은 성능을 보여줍니다.
제한 사항 및 결론
ColabDock에도 몇 가지 제한 사항이 있습니다. 현재 ColabDock은 AlphaFold2 거리 맵의 상한에 따라 결정되는 22Å보다 작은 거리만 수용할 수 있습니다. 이러한 제한으로 인해 모델은 XL-MS 시약의 작은 하위 집합에만 적용 가능합니다.
조각 기반 최적화 없이 ColabDock은 제한된 메모리로 인해 NVIDIA A100 그래픽 처리 장치(GPU)에서 잔여물이 1,200개 미만인 컴플렉스만 처리할 수 있습니다.
또한 이 방법은 특히 대규모 단백질 복합체의 경우 시간이 많이 걸릴 수 있습니다. AlphaFold2의 bfloat16 부동 소수점 형식 버전을 사용하면 메모리를 절약하고 계산 속도를 높이는 데 도움이 될 것으로 예상됩니다.
앞으로는 연구자들이 이를 반복적으로 최적화한 후 통합 프레임워크로서 ColabDock이 실험 단백질 과학과 계산 단백질 과학 사이의 격차를 해소하는 데 도움이 될 것이라고 믿습니다.
논문 링크:https://www.nature.com/articles/s42256-024-00873-z
以上がNature サブジャーナル、北京大学チームの一般的な AI フレームワークは、実験と計算の間のギャップを埋める、タンパク質間ドッキングの包括的な構造予測を実施の詳細内容です。詳細については、PHP 中国語 Web サイトの他の関連記事を参照してください。
 踊りましょう:私たちの人間のニューラルネットを微調整するための構造化された動きApr 27, 2025 am 11:09 AM
踊りましょう:私たちの人間のニューラルネットを微調整するための構造化された動きApr 27, 2025 am 11:09 AM科学者は、彼らの機能を理解するために、人間とより単純なニューラルネットワーク(C. elegansのものと同様)を広く研究してきました。 ただし、重要な疑問が生じます。新しいAIと一緒に効果的に作業するために独自のニューラルネットワークをどのように適応させるのか
 新しいGoogleリークは、Gemini AIのサブスクリプションの変更を明らかにしますApr 27, 2025 am 11:08 AM
新しいGoogleリークは、Gemini AIのサブスクリプションの変更を明らかにしますApr 27, 2025 am 11:08 AMGoogleのGemini Advanced:Horizonの新しいサブスクリプションティア 現在、Gemini Advancedにアクセスするには、1か月あたり19.99ドルのGoogle One AIプレミアムプランが必要です。 ただし、Android Authorityのレポートは、今後の変更を示唆しています。 最新のGoogle p
 データ分析の加速がAIの隠されたボトルネックをどのように解決しているかApr 27, 2025 am 11:07 AM
データ分析の加速がAIの隠されたボトルネックをどのように解決しているかApr 27, 2025 am 11:07 AM高度なAI機能を取り巻く誇大宣伝にもかかわらず、エンタープライズAIの展開内に大きな課題が潜んでいます:データ処理ボトルネック。 CEOがAIの進歩を祝う間、エンジニアはクエリの遅い時間、過負荷のパイプライン、
 MarkitDown MCPは、任意のドキュメントをマークダウンに変換できます!Apr 27, 2025 am 09:47 AM
MarkitDown MCPは、任意のドキュメントをマークダウンに変換できます!Apr 27, 2025 am 09:47 AMドキュメントの取り扱いは、AIプロジェクトでファイルを開くだけでなく、カオスを明確に変えることです。 PDF、PowerPoint、Wordなどのドキュメントは、あらゆる形状とサイズでワークフローをフラッシュします。構造化された取得
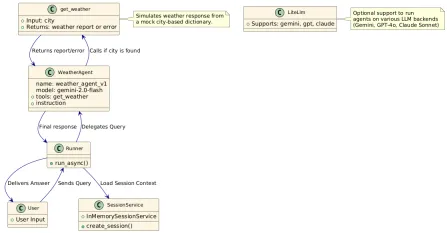 建物のエージェントにGoogle ADKを使用する方法は? - 分析VidhyaApr 27, 2025 am 09:42 AM
建物のエージェントにGoogle ADKを使用する方法は? - 分析VidhyaApr 27, 2025 am 09:42 AMGoogleのエージェント開発キット(ADK)のパワーを活用して、実際の機能を備えたインテリジェントエージェントを作成します。このチュートリアルは、ADKを使用して会話エージェントを構築し、GeminiやGPTなどのさまざまな言語モデルをサポートすることをガイドします。 w
 効果的な問題解決のためにLLMを介したSLMの使用 - 分析VidhyaApr 27, 2025 am 09:27 AM
効果的な問題解決のためにLLMを介したSLMの使用 - 分析VidhyaApr 27, 2025 am 09:27 AMまとめ: Small Language Model(SLM)は、効率のために設計されています。それらは、リソース不足、リアルタイム、プライバシーに敏感な環境の大手言語モデル(LLM)よりも優れています。 特にドメインの特異性、制御可能性、解釈可能性が一般的な知識や創造性よりも重要である場合、フォーカスベースのタスクに最適です。 SLMはLLMSの代替品ではありませんが、精度、速度、費用対効果が重要な場合に理想的です。 テクノロジーは、より少ないリソースでより多くを達成するのに役立ちます。それは常にドライバーではなく、プロモーターでした。蒸気エンジンの時代からインターネットバブル時代まで、テクノロジーの力は、問題の解決に役立つ範囲にあります。人工知能(AI)および最近では生成AIも例外ではありません
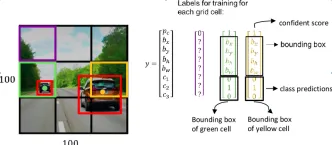 コンピュータービジョンタスクにGoogle Geminiモデルを使用する方法は? - 分析VidhyaApr 27, 2025 am 09:26 AM
コンピュータービジョンタスクにGoogle Geminiモデルを使用する方法は? - 分析VidhyaApr 27, 2025 am 09:26 AMコンピュータービジョンのためのGoogleGeminiの力を活用:包括的なガイド 大手AIチャットボットであるGoogle Geminiは、その機能を会話を超えて拡張して、強力なコンピュータービジョン機能を網羅しています。 このガイドの利用方法については、
 Gemini 2.0 Flash vs O4-Mini:GoogleはOpenaiよりもうまくやることができますか?Apr 27, 2025 am 09:20 AM
Gemini 2.0 Flash vs O4-Mini:GoogleはOpenaiよりもうまくやることができますか?Apr 27, 2025 am 09:20 AM2025年のAIランドスケープは、GoogleのGemini 2.0 FlashとOpenaiのO4-Miniの到着とともに感動的です。 数週間離れたこれらの最先端のモデルは、同等の高度な機能と印象的なベンチマークスコアを誇っています。この詳細な比較


ホットAIツール

Undresser.AI Undress
リアルなヌード写真を作成する AI 搭載アプリ

AI Clothes Remover
写真から衣服を削除するオンライン AI ツール。

Undress AI Tool
脱衣画像を無料で

Clothoff.io
AI衣類リムーバー

Video Face Swap
完全無料の AI 顔交換ツールを使用して、あらゆるビデオの顔を簡単に交換できます。

人気の記事

ホットツール

Dreamweaver Mac版
ビジュアル Web 開発ツール

SublimeText3 Mac版
神レベルのコード編集ソフト(SublimeText3)

SAP NetWeaver Server Adapter for Eclipse
Eclipse を SAP NetWeaver アプリケーション サーバーと統合します。

MinGW - Minimalist GNU for Windows
このプロジェクトは osdn.net/projects/mingw に移行中です。引き続きそこでフォローしていただけます。 MinGW: GNU Compiler Collection (GCC) のネイティブ Windows ポートであり、ネイティブ Windows アプリケーションを構築するための自由に配布可能なインポート ライブラリとヘッダー ファイルであり、C99 機能をサポートする MSVC ランタイムの拡張機能が含まれています。すべての MinGW ソフトウェアは 64 ビット Windows プラットフォームで実行できます。

PhpStorm Mac バージョン
最新(2018.2.1)のプロフェッショナル向けPHP統合開発ツール

ホットトピック
 7753
7753 15
15 1643
1643 14
14 1398
1398 52
52 1293
1293 25
25 1234
1234 29
29


