ホームページ >テクノロジー周辺機器 >AI >わずか数秒でタンパク質の動態情報を正確に推定できる山東大学、北京工業大学などのAIモデルRMSF-netがNatureサブジャーナルに掲載されました。
わずか数秒でタンパク質の動態情報を正確に推定できる山東大学、北京工業大学などのAIモデルRMSF-netがNatureサブジャーナルに掲載されました。
- 王林オリジナル
- 2024-07-10 14:55:00840ブラウズ

タンパク質のダイナミクスは、そのメカニズムを理解する上で非常に重要です。ただし、タンパク質の動態情報をコンピューターで予測することは困難です。
ここで、山東大学、BioMap、北京工業大学、湖北医科大学、寧夏医科大学、キングアブドラ科学技術大学 (KAUST) の研究チームが、以前の手法を上回るニューラル ネットワーク モデル RMSF -net を提案しました。大規模なタンパク質ダイナミクス データセットで最良の結果を生成するモデルは、タンパク質のダイナミクス情報を数秒で正確に推測できます。
実験タンパク質構造データとクライオ EM データの統合から効果的に学習することにより、このメソッドは、クライオ EM 画像と PDB モデルの間のインタラクティブな双方向制約と監視を正確に特定し、ダイナミクス予測の効率を最大化することができます。
RMSF-net は、タンパク質動態研究において重要な役割を果たす無料で使用できるツールです。
この研究は「複雑な原子構造とクライオEM密度情報を統合した深層学習によるタンパク質構造の柔軟性の正確な予測」と題され、7月2日に「Nature Communications」に掲載されました。

- https://www.nature.com/articles/s41467-024-49858-x
RMSF-net GitHub アドレス:
- https://github. com/XintSong/RMSF-net
タンパク質のダイナミクス
タンパク質のダイナミクスは、そのメカニズムを理解する上で非常に重要です。クライオ電子顕微鏡 (クライオ EM) テクノロジーは、ほとんどのタンパク質を分解でき、高分子構造は 3D 密度マップで表されます。
クライオ電子顕微鏡の限界
元の 2D 粒子画像の解像度と S/N 比が低いため、クライオ電子顕微鏡分析では再構成中の小さな構造変化を解決できません。
クライオ電子顕微鏡におけるディープラーニングの応用
ディープラーニング手法は、クライオ電子顕微鏡画像の自動解析に広く使用されています。高解像度のクライオ EM マップを使用すると、クライオ EM マップからタンパク質データ バンク (PDB) モデルを構築できます。
RMSF-net の概要
RMSF-net は、極低温電子顕微鏡密度マップ用のニューラル ネットワーク モデルです。クライオ EM 密度と PDB モデル情報を活用して、タンパク質の動的情報を数秒で正確に推測します。
RMSF
RMSF は、分子動力学 (MD) 解析において分子構造の柔軟性を評価するために広く使用されている測定方法です。その主な目的は、タンパク質内の局所構造 (残基、原子) の RMSF を予測することです。
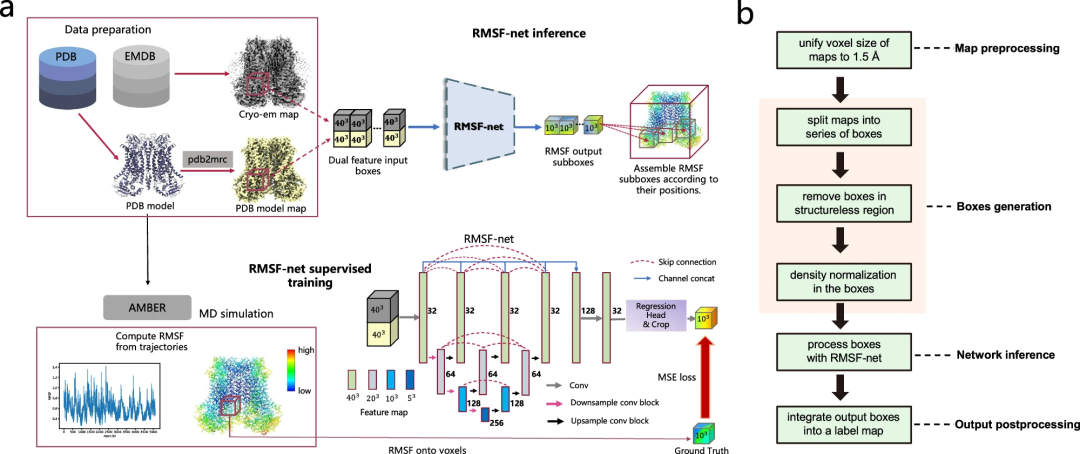
クライオ EM 画像に加えて、RMSF-net は追加入力として PDB モデルを利用して、MD シミュレーション結果に非常に近い RMSF 予測を生成します。
RMSF-net は、相互接続された 2 つのモジュールを含む 3 次元畳み込みニューラル ネットワークです。メイン モジュールは、Unet+ (L3) アーキテクチャを使用して、入力密度ボックスの特徴をエンコードおよびデコードします。別のモジュールは、1x1 畳み込みを利用して、Unet+バックボーンによって生成された特徴マップのチャネルを回帰します。次に、中心クリッピングが回帰モジュール出力に適用されて、中心の RMSF サブボックスが取得されます。ここで、ボクセル値は、その中に含まれる原子の RMSF に対応します。最後に、RMSF サブボックスは、結合アルゴリズムを使用して RMSF マップに空間的に結合されます。
さらに、研究者らは、RMSF-net のトレーニングと検証のための大規模なタンパク質ダイナミクス データセットも構築しました。このデータセットでは、適合した PDB モデルを持つ 335 個のクライオ EM 構造エントリが選択され、対応する MD シミュレーションが実行されました。包括的な実験結果は、RMSF-net の効率と有効性を実証しています。
表: データセットに対するさまざまな RMSF 予測手法のパフォーマンス。 (出典: 論文)
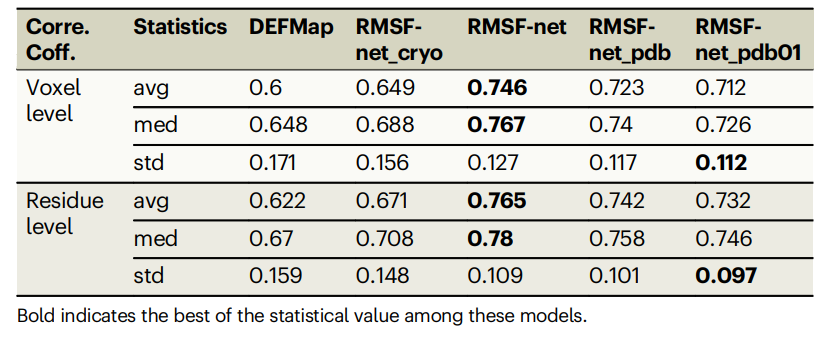
RMSF-netは、厳密な5分割相互検証で良好なパフォーマンスを示し、MDシミュレーション結果との相関係数は0.746±0.127でした。 RMSF-net の相関係数は、DEFMap と比較して 15%、ベースライン手法と比較して 10% 改善されました。
ダイナミクス予測の解釈可能性
研究者らは、比較実験を通じてRMSFネットダイナミクス予測の解釈可能性を強化しました。彼らは、RMSF 予測プロセスを 2 つのステップに分けています:
- 構造情報抽出 (Occ2RMSF-net)
- 抽出された構造情報に基づく運動学予測
研究によると、低温電子顕微鏡スペクトル (DEFMap や RMSF-net_cryo など) に基づくモデルの動力学予測は主にタンパク質の解釈 構造の実装。これは、構造と機能の関係の第一原理と一致する、タンパク質のトポロジーとダイナミクスの関係を強調しています。
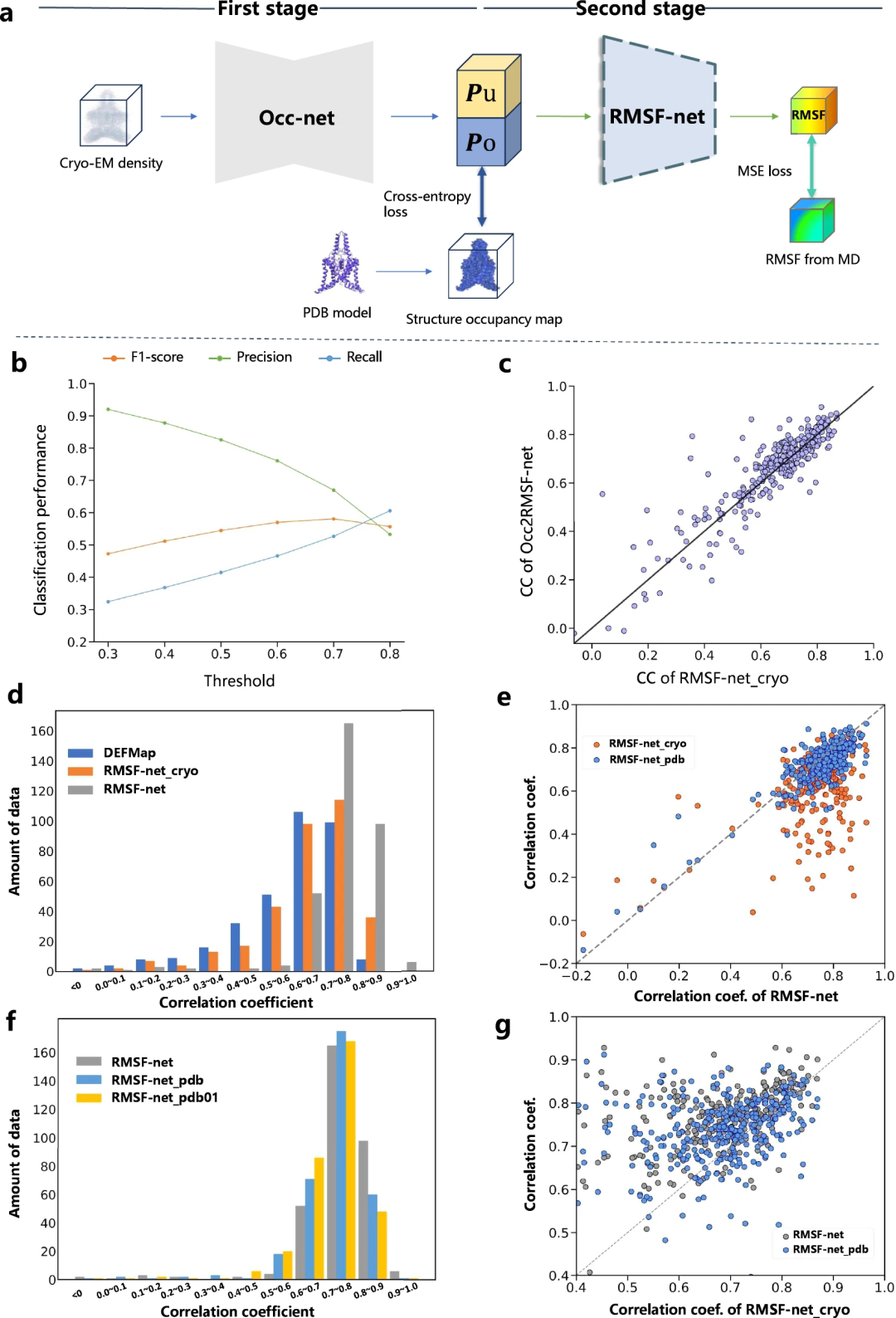
さらに、RMSF-net_cryo、RMSF-net_pdb、および最終的な二重結合 RMSF-net の包括的な比較により、次のことが証明されます: 一方で、PDB モデルからの構造情報が再生されます。 RMSF-netの役割における主要な役割。ディープモデルはMDシミュレーションから構造トポロジーと柔軟性の間のパターンを学習し、その一方で、モデルは極低温電子顕微鏡の不均一な密度分布に含まれる速度論的情報によってさらに強化されます。地図。これらの結果は、RMSF-net におけるタンパク質動態予測におけるクライオ EM マップと PDB モデルからの情報の相補的な役割を検証します。
限界と今後の方向性
RMSF-net が主に溶液中の純粋なタンパク質とその複合体の柔軟性を予測することに限定されていることは否定できません。この方法は、小分子リガンドに結合したときまたは膜環境におけるタンパク質の動的特性に関して、特定の局所領域で不正確さを示す可能性があります。
RMSF-net の優れたパフォーマンスは、この方向でのさらなる研究の実現可能性を明らかにしています。この研究は、核酸およびタンパク質-核酸複合体には拡張されていません。多重構造予測や遷移解析を含む高分子動力学のあらゆる側面の包括的な特性評価には、今後さらに広範かつ詳細な研究が必要です。
それにもかかわらず、RMSF-net は、その優れたパフォーマンスと超高速処理速度により、タンパク質のダイナミクスを予測するツールとして、タンパク質の構造とダイナミクスの研究において依然として大きな応用の可能性を秘めています。
注: 表紙はインターネットから取得したものです
以上がわずか数秒でタンパク質の動態情報を正確に推定できる山東大学、北京工業大学などのAIモデルRMSF-netがNatureサブジャーナルに掲載されました。の詳細内容です。詳細については、PHP 中国語 Web サイトの他の関連記事を参照してください。

