
編集者 | 大根の皮
タンパク質は、病原体と戦うための確立されたツールであり、実験的テストのために潜在的な治療法を絞り込むために使用されます。高品質のタンパク質構造が必要であり、タンパク質は完全または部分的に硬いとみなされることがよくあります。
ベルリン自由大学の研究者らは、タンパク質-リガンド複合体の完全に柔軟な全原子構造を配列情報から直接予測できる人工知能システムを開発しました。
古典的なドッキング方法は依然として優れていますが、これは標的タンパク質の結晶構造にも依存します。柔軟な全原子構造の予測に加えて、予測信頼度メトリック (plDDT) を使用して、正確な予測を選択し、強い結合剤と弱い結合剤を区別することができます。
この研究は「Umolによる配列情報からのタンパク質-リガンド複合体の構造予測」と題され、2024年5月28日に「Nature Communications」に掲載されました。

タンパク質とタンパク質の標的接触は、新薬の評価や既知の物質の再配置において重要な問題です。既存の接触方法には限界があります。高品質のタンパク質構造が必要であること、正確な接触姿勢を決定することが難しいこと、構造安定性などの他の要素を反映することが難しい結合能力 (親和性) 評価に基づいていることがほとんどです。しかし、既存の接触方法は、高品質のタンパク質構造、正確な接触ポーズ、およびマルチベースの親和性評価の必要性によって制限されています。したがって、新しいリガンドの探索は、タンパク質の構築と構造評価を組み合わせたアプローチによって制限されます。
この分野では機械学習が適用されていますが、既知のターゲット領域でのパフォーマンスはスコアリング関数に基づく古典的な手法をまだ超えていません。さらに、予測されたタンパク質構造は、リガンドドッキングに直接使用するのには適さないことがよくあります。
さらに、評価セット内の構造が類似性ではなく放出時間に基づいて分割された場合、バイアスが導入され、特にトレーニングでは見られない受容体構造に直面した場合、パフォーマンスは半減します。
タンパク質の柔軟性は、結合状態に到達し、ドッキングを成功させるために重要です。RoseTTAFold All-Atom はタンパク質を予測する際にリガンドに結合できますが、PoseBusters テスト セットでの成功率はわずか 42% であり、目に見えないタンパク質の場合は非常に困難です。タンパク質の挙動は不明であり、タンパク質-リガンド複合体構造予測の課題がまだ完全に解決されていないことを示しています。
ベルリン自由大学のチームは、AlphaFold2 の EvoFormer を拡張することで、配列情報に基づいてタンパク質-リガンド複合体の構造を予測できる AI 手法を開発しました。このネットワークは RFAA に似ていますが、3D 軌跡が含まれておらず、テンプレート構造または追加の結晶学的リガンド データが入力としてまたはトレーニング中に使用される点が異なります。
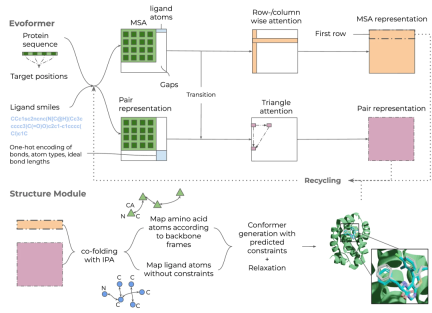
イラスト:ウモルの概要。 (出典: 論文)
タンパク質配列から開始して、代替タンパク質ターゲット (ポケット) とリガンドを SMILES が作成し、多重配列アラインメント (MSA) と結合マトリックスを作成します。これから、ネットワーク内にフィーチャが生成され、3D 構造が生成されます。最終的なタンパク質-リガンド複合体構造を生成するために構造情報は必要ないため、タンパク質やリガンドの柔軟性に制限はありません。
Umol は、PoseBusters テスト セットにポケット情報を含めた場合、それぞれ 45%、42% と、最も近い RoseTTAFold All-Atom および NeuralPlexer1 の 24% と比較して、より高い成功率 (SR、リガンド RMSD ≤ 2 Å) を達成します。これは、タンパク質-リガンド構造予測において最もパフォーマンスの高い方法です。

イラスト: 予測精度。 (出典:論文)
Umol からポケット情報を削除し、RFAA からテンプレート情報を削除すると、SR はそれぞれ 18% と 8% に低下します。 AF 予測で DiffDock を使用する場合、精度は 21% ですが、高精度のインターフェイス予測 (ポケット RMSD
2 Å 成功閾値をわずかに超える多くのリガンドポーズは同等である可能性があり、より柔軟なスコアリングシステムが必要である可能性があることを示唆しています。 Umol の成功率は、2.35 Å のしきい値で AutoDock Vina を上回ります。天然のタンパク質構造がスコアリングに使用されない場合、たとえ小さなアライメントエラーであっても問題になる可能性があります。
共フォールディングされたタンパク質-リガンド複合体は、薬物の再配置を促進する可能性があります。特に、研究者らは、リガンドの予測された IDDT (plDDT) を使用して正確なドッキングポーズを選択できる一方、タンパク質ポケットの pIDDT は正確な界面を選択するのに適していることを発見しました。

イラスト: 信頼性の指標と精度。 (出典: 論文)
リガンド plDDT も高親和性リガンドを低親和性リガンドから分離し、Umol および Umol ポケットの不確実性の予測の一部が弱い結合剤である可能性があることを示唆しています。これは、Umol の能力をさらに実証し、タンパク質とリガンドの相互作用の重要な側面が理解されているようであることを強調しています。

イラスト: BindingDB の予測。 (出典: 論文)
ポケット情報なしで 18% の精度にもかかわらず、ネットワークは依然として強いバインダーと弱いバインダーをある程度区別できます。これは、未知の複合体に注釈を付けるのに特に役立ち、チームは 336 個のタンパク質 - リガンド構造を非常に高い信頼性で提示しました (リガンド plDDT > 85)。これらの構造は合理的であるように見え、L-plDDT スコアは高いですが、依然として実験的に検証する必要があることに注意してください。
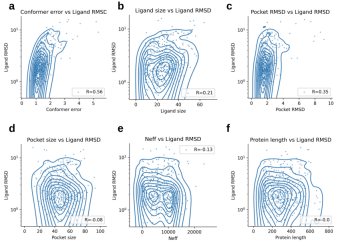
図: Umol-pocket を使用して、PoseBusters テスト セット (n=428) の予測されたさまざまな特徴とリガンド RMSD (LRMSD) の間の関係を分析します。 (出典: 論文)
研究者らは、モデルの予測性能と「同じタンパク質またはリガンドに関連する異なる特徴」との間に明確な関係を発見できませんでした。

イラスト: 最も難しい 5 つの構造。 (出典: 論文)
しかし、Umol-pocket は、他の方法では予測が困難な 5 ケース中 3 ケースで正確でした。訓練されたネットワークを反転することにより、新しいリガンド結合タンパク質またはタンパク質結合リガンドを設計できます。もう 1 つのオプションは、転移学習を使用して、同じ目的の生成拡散モデルを作成することです。この場合、リガンドまたはタンパク質 plDDT を最大化して、高親和性バインダーを作成することができます。
PDBbind の現在のバージョンには、2019 年に PDB から処理されたデータが含まれています。それ以来、追加のタンパク質-リガンド複合体が提出されており、より高い精度が達成できる可能性があることが示唆されています。
しかし、意味のあるタンパク質-リガンドドッキング結果を得るためにどの程度の精度が必要かは現時点では不明です。タンパク質構造予測の高精度は、低分子や RNA などの他の分子が関与するタスクでは達成できません。
タンパク質の共進化情報がないと、構造予測の精度が急速に低下します。小分子や RNA については同様の情報源がないため、原子の表現に頼らざるを得ません。
表: PDBBind 2020 バージョンの PoseBuster ベンチマーク セットの成功率 (RMSD≤2Å のリガンドの割合) を配列同一性 (seqid) で割ったもの。 (出典: 論文)

研究者らは、ポケット情報は非常に効果的であり、ポケット情報がないと深層学習手法は過学習になりやすいと考えています。この発見は、PoseBusters テスト セット内の多くの分子にはトレーニング データ セット内の非常に類似した類似体が含まれているものの、この類似性はモデルの成功とは相関しないという観察をさらに裏付けます。

イラスト: いくつかのテスト。 (出典: 論文)
Vina や Gold などの構造ベースのドッキング手法では、同程度の過学習は観察されません。それらは原子スコア関数に基づいており、したがってタンパク質の相同性に同程度に依存していないため、これは予想されます。
ディープ ラーニング手法はトレーニング セットで大幅に高いパフォーマンスを示し、タンパク質の相同性がタンパク質とリガンドのドッキングにおいて重要な役割を果たしていることが示されています。テスト セットでの RFAA のパフォーマンスはトレーニング セットでのパフォーマンスよりも高く、これはトレーニング セットとテスト セットの間でデータ漏洩の可能性があることを示しています。
結論として、タンパク質とリガンドの相互作用の複雑さを完全に理解するにはまだ長い道のりがありますが、深層学習を使用して複合体全体の構造を予測することで、科学者は解決策に近づくことができる可能性があります。
Umol: https://github.com/patrickbryant1/Umol
紙のリンク: https://www.nature.com/articles/s41467-024-48837-6
以上が配列情報を使用してタンパク質-リガンド複合体の構造を直接予測することで、RoseTTAFold シリーズの成功率を上回ります。の詳細内容です。詳細については、PHP 中国語 Web サイトの他の関連記事を参照してください。
 カリフォルニアは、AIをタップして、速い追跡の山火事回復許可を促進しますMay 04, 2025 am 11:10 AM
カリフォルニアは、AIをタップして、速い追跡の山火事回復許可を促進しますMay 04, 2025 am 11:10 AMAIは、野火の回復許可を合理化します オーストラリアのハイテク企業ArchistarのAIソフトウェアは、機械学習とコンピュータービジョンを利用して、地域の規制に準拠するための建築計画の評価を自動化します。この前検証は重要です
 米国がエストニアのAI駆動型デジタル政府から学ぶことができることMay 04, 2025 am 11:09 AM
米国がエストニアのAI駆動型デジタル政府から学ぶことができることMay 04, 2025 am 11:09 AMエストニアのデジタル政府:米国のモデル? 米国は官僚的な非効率性と闘っていますが、エストニアは説得力のある代替品を提供しています。 この小さな国は、AIを搭載した、ほぼ100%デジタル化された市民中心の政府を誇っています。 これはそうではありません
 生成AIによる結婚式の計画May 04, 2025 am 11:08 AM
生成AIによる結婚式の計画May 04, 2025 am 11:08 AM結婚式を計画することは記念碑的な仕事であり、しばしば最も組織化されたカップルでさえ圧倒されます。 この記事は、AIの影響に関する進行中のフォーブスシリーズの一部(こちらのリンクを参照)で、生成AIが結婚式の計画にどのように革命をもたらすことができるかを調べます。 結婚式のpl
 デジタル防衛AIエージェントとは何ですか?May 04, 2025 am 11:07 AM
デジタル防衛AIエージェントとは何ですか?May 04, 2025 am 11:07 AM政府は、さまざまな確立されたタスクにそれらを利用している一方で、企業はAIエージェントを販売のためにますます活用しています。 ただし、消費者の支持者は、個人がターゲットのターゲットに対する防御として自分のAIエージェントを所有する必要性を強調しています
 生成エンジン最適化に関するビジネスリーダーのガイド(GEO)May 03, 2025 am 11:14 AM
生成エンジン最適化に関するビジネスリーダーのガイド(GEO)May 03, 2025 am 11:14 AMGoogleはこのシフトをリードしています。その「AIの概要」機能はすでに10億人以上のユーザーにサービスを提供しており、誰もがリンクをクリックする前に完全な回答を提供しています。[^2] 他のプレイヤーも速く地位を獲得しています。 ChatGpt、Microsoft Copilot、およびPE
 このスタートアップは、AIエージェントを使用して悪意のある広告となりすましアカウントと戦っていますMay 03, 2025 am 11:13 AM
このスタートアップは、AIエージェントを使用して悪意のある広告となりすましアカウントと戦っていますMay 03, 2025 am 11:13 AM2022年、彼はソーシャルエンジニアリング防衛のスタートアップDoppelを設立してまさにそれを行いました。そして、サイバー犯罪者が攻撃をターボチャージするためのより高度なAIモデルをハーネスするにつれて、DoppelのAIシステムは、企業が大規模に戦うのに役立ちました。
 世界モデルがどのように生成AIとLLMの未来を根本的に再形成しているかMay 03, 2025 am 11:12 AM
世界モデルがどのように生成AIとLLMの未来を根本的に再形成しているかMay 03, 2025 am 11:12 AM出来上がりは、適切な世界モデルとの対話を介して、生成AIとLLMを実質的に後押しすることができます。 それについて話しましょう。 革新的なAIブレークスルーのこの分析は、最新のAIで進行中のForbes列のカバレッジの一部であり、
 2050年5月:私たちは祝うために何を残しましたか?May 03, 2025 am 11:11 AM
2050年5月:私たちは祝うために何を残しましたか?May 03, 2025 am 11:11 AM労働者2050年。全国の公園は、ノスタルジックなパレードが街の通りを通り抜ける一方で、伝統的なバーベキューを楽しんでいる家族でいっぱいです。しかし、お祝いは現在、博物館のような品質を持っています。


ホットAIツール

Undresser.AI Undress
リアルなヌード写真を作成する AI 搭載アプリ

AI Clothes Remover
写真から衣服を削除するオンライン AI ツール。

Undress AI Tool
脱衣画像を無料で

Clothoff.io
AI衣類リムーバー

Video Face Swap
完全無料の AI 顔交換ツールを使用して、あらゆるビデオの顔を簡単に交換できます。

人気の記事

ホットツール

MinGW - Minimalist GNU for Windows
このプロジェクトは osdn.net/projects/mingw に移行中です。引き続きそこでフォローしていただけます。 MinGW: GNU Compiler Collection (GCC) のネイティブ Windows ポートであり、ネイティブ Windows アプリケーションを構築するための自由に配布可能なインポート ライブラリとヘッダー ファイルであり、C99 機能をサポートする MSVC ランタイムの拡張機能が含まれています。すべての MinGW ソフトウェアは 64 ビット Windows プラットフォームで実行できます。

メモ帳++7.3.1
使いやすく無料のコードエディター

Dreamweaver Mac版
ビジュアル Web 開発ツール

SublimeText3 Mac版
神レベルのコード編集ソフト(SublimeText3)

SublimeText3 英語版
推奨: Win バージョン、コードプロンプトをサポート!






