Maison >Périphériques technologiques >IA >NVIDIA, Mila et Caltech lancent conjointement un modèle multimodal de structure moléculaire-texte combinant LLM et découverte de médicaments
NVIDIA, Mila et Caltech lancent conjointement un modèle multimodal de structure moléculaire-texte combinant LLM et découverte de médicaments
- WBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBavant
- 2024-01-14 20:00:05891parcourir

Auteur | Liu Shengchao
Éditeur | Kaixia
À partir de 2021, la combinaison du grand langage et de la multimodalité a balayé la communauté de recherche sur l'apprentissage automatique.
Avec le développement de grands modèles et d'applications multimodales, pouvons-nous appliquer ces techniques à la découverte de médicaments ? Et ces descriptions textuelles en langage naturel peuvent-elles apporter de nouvelles perspectives à ce problème difficile ? La réponse est oui, et nous en sommes optimistes
Récemment, des équipes de recherche de l'Institut des algorithmes d'apprentissage de Montréal (Mila) au Canada, de NVIDIA Research, de l'Université de l'Illinois à Urbana-Champaign (UIUC), de l'Université de Princeton et de l'Université de Californie Institute of Technology, un modèle multimodal de structure moléculaire-texte MoleculeSTM est proposé en apprenant conjointement la structure chimique et la description textuelle des molécules grâce à des stratégies d'apprentissage contrastées.
Cette recherche s'intitule « Structure moléculaire multimodale – modèle de texte pour la récupération et l'édition textuelles » et a été publiée dans « Nature Machine Intelligence » le 18 décembre 2023.

Lien papier : https://www.nature.com/articles/s42256-023-00759-6 doit être réécrit
Le Dr Liu Shengchao est le premier auteur, et le professeur Anima Anandkumar de NVIDIA Research est. l'auteur correspondant. Nie Weili, Wang Chengpeng, Lu Jiarui, Qiao Zhuoran, Liu Ling, Tang Jian et Xiao Chaowei sont co-auteurs.
Ce projet a été réalisé par le Dr Liu Shengchao après avoir rejoint NVIDIA Research en mars 2022, sous la direction des professeurs Nie Weili, professeur Tang Jian, professeur Xiao Chaowei et professeur Anima Anandkumar.
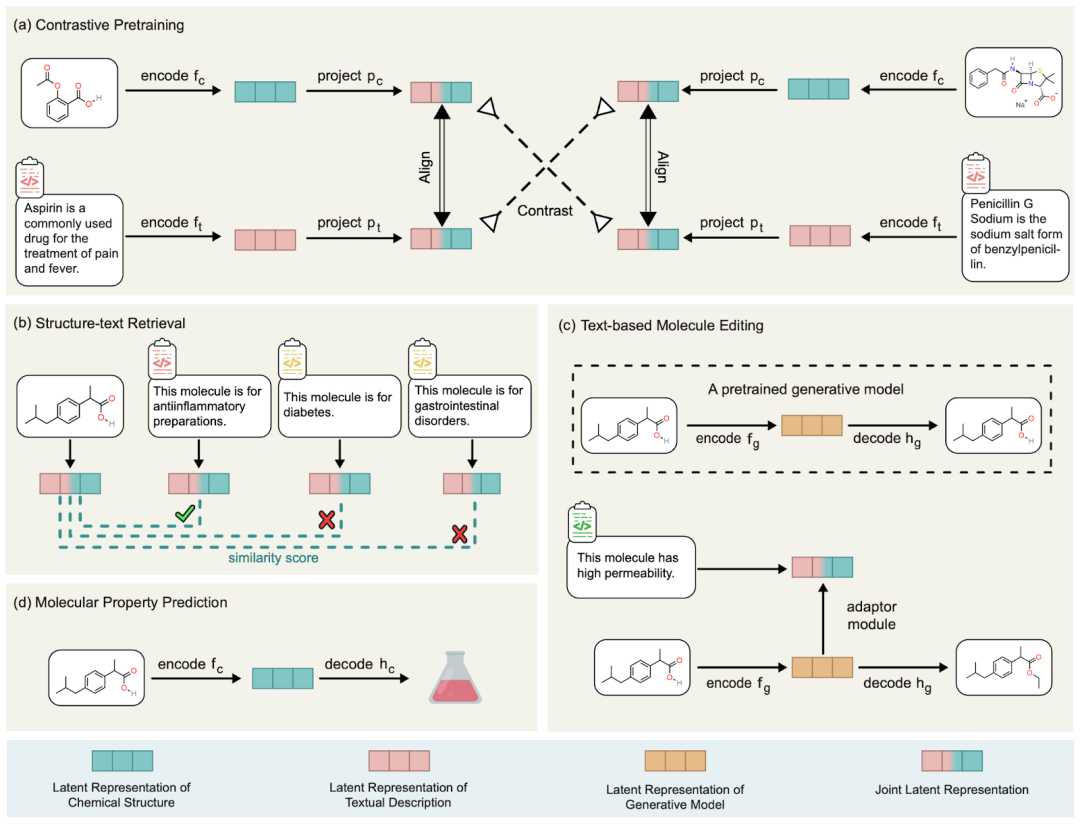
Le Dr Liu Shengchao a déclaré : "Notre motivation était de mener une exploration préliminaire du LLM et de la découverte de médicaments, et a finalement proposé MoleculeSTM." est très simple et directe, c'est-à-dire que la description des molécules peut être divisée en deux catégories : la structure chimique interne et la description fonctionnelle externe. Ici, nous utilisons une méthode contrastive de pré-formation pour aligner et connecter ces deux types d'informations. Le diagramme spécifique est présenté dans la figure ci-dessous
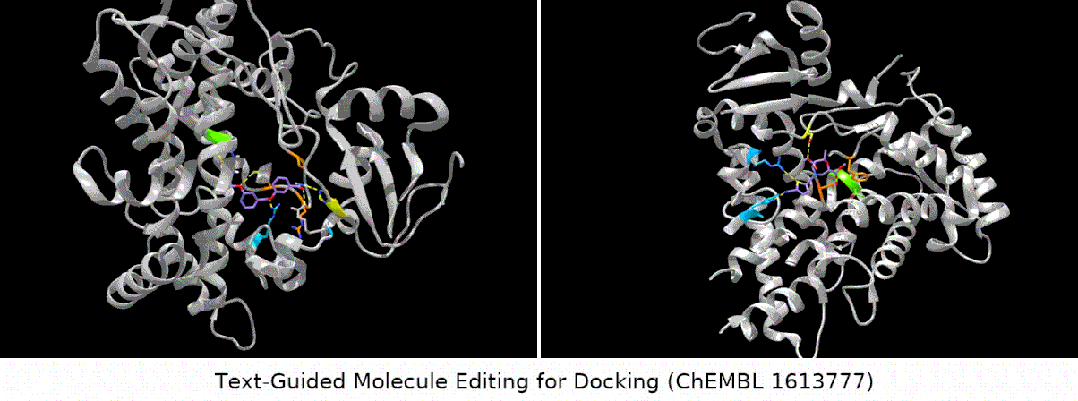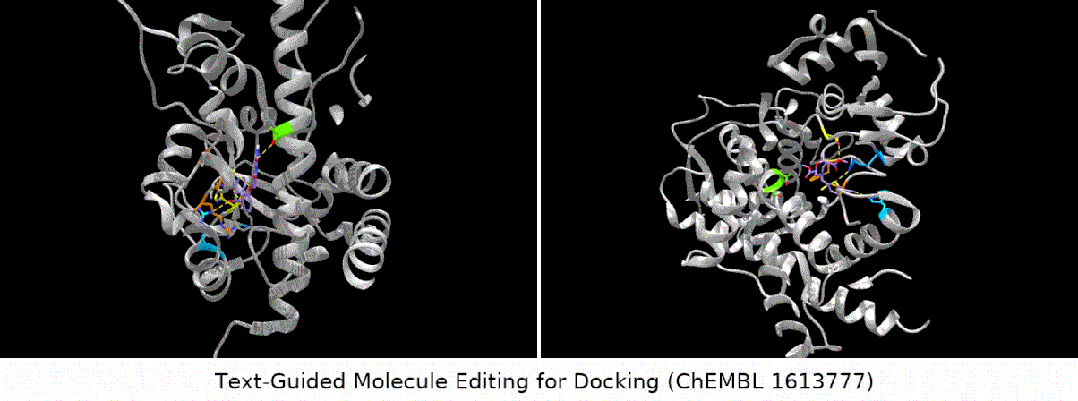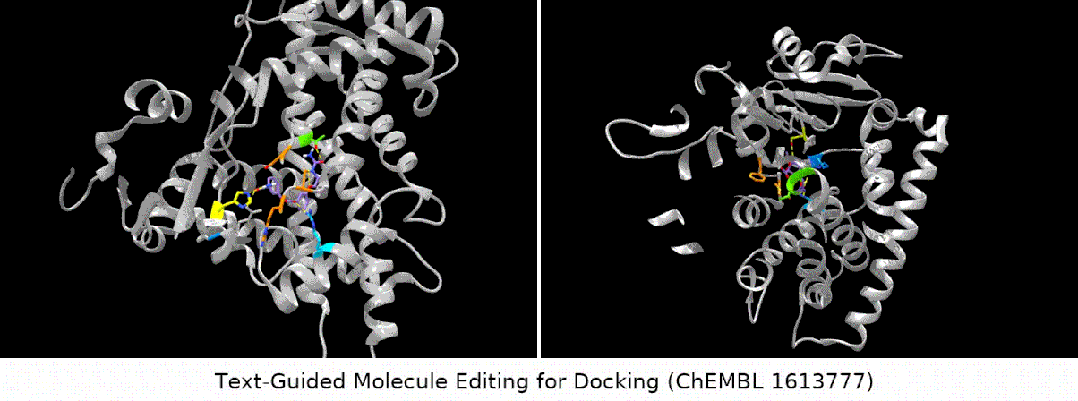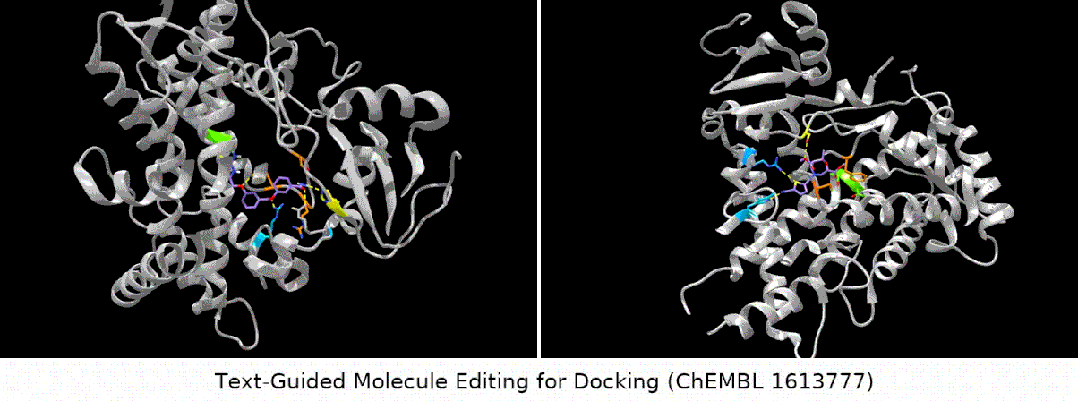
Illustration : organigramme MoleculeSTM.
Et cet alignement de MoleculeSTM a une très bonne propriété : lorsqu'il y a des tâches difficiles à résoudre dans l'espace chimique, nous pouvons les transférer dans l'espace du langage naturel. Et les tâches en langage naturel seront relativement plus faciles à résoudre en raison de ses caractéristiques. Sur cette base, nous avons conçu une grande variété de tâches en aval pour vérifier son efficacité. Ci-dessous, nous discutons en détail de plusieurs idées.
 Caractéristiques du langage naturel et des grands modèles de langage
Caractéristiques du langage naturel et des grands modèles de langage
Le vocabulaire ouvert signifie que nous pouvons exprimer toutes les connaissances humaines actuelles en langage naturel, de sorte que les nouvelles connaissances qui apparaîtront dans le futur peuvent également être résumées et résumées en utilisant le langage existant. Résumer. Par exemple, si une nouvelle protéine apparaît, nous espérons décrire sa fonction en langage naturel. La compositionnalité signifie qu'en langage naturel, un concept complexe peut être exprimé conjointement par plusieurs concepts simples. Ceci est très utile pour des tâches telles que l'édition multi-attributs : il est très difficile d'éditer des molécules pour qu'elles répondent à plusieurs propriétés en même temps dans l'espace chimique, mais nous pouvons exprimer plusieurs propriétés très simplement en langage naturel.
Dans notre récent travail ChatDrug (https://arxiv.org/abs/2305.18090), nous avons exploré les caractéristiques du dialogue entre le langage naturel et les grands modèles de langage. Les amis qui sont intéressés par cela peuvent le vérifier- Conception de tâches. induit par les fonctionnalités fait référence à la conception de la planification et de l'organisation des tâches en fonction des caractéristiques du produit ou du système
- La première tâche que nous envisageons est d'être capable d'effectuer des simulations informatiques et d'obtenir des résultats. À l’avenir, les résultats de vérification en laboratoire humide seront pris en compte, mais cela n’entre pas dans le cadre des travaux actuels.
- Deuxièmement, nous ne considérons que les problèmes dont les résultats sont ambigus. Des exemples spécifiques incluent le fait de rendre une certaine molécule plus soluble ou pénétrable dans l’eau. Certains problèmes ont des résultats clairs, comme l'ajout d'un certain groupe fonctionnel à une certaine position dans une molécule. Nous pensons que de telles tâches sont plus simples et plus directes pour les experts en drogue et en chimie. Elle peut donc être utilisée comme tâche de validation de principe à l'avenir, mais elle ne deviendra pas l'objectif principal de la tâche.
Nous avons ainsi conçu trois grandes catégories de tâches :
- Récupération de texte de structure sans tir ;
- Édition de molécules basée sur du texte sans tir ;
- Prédiction des propriétés moléculaires.
Nous nous concentrerons sur la deuxième tâche dans la section suivante
Les résultats qualitatifs de l'édition de molécules sont reformulés comme suit :
Cette tâche consiste à saisir une molécule et une description en langage naturel (comme des attributs supplémentaires) au en même temps, puis il est souhaitable de pouvoir produire des descriptions textuelles en langage complexe de nouvelles molécules. Il s’agit d’une optimisation des leads guidée par texte.
La méthode spécifique consiste à utiliser le modèle de génération de molécules déjà entraîné et notre MoleculeSTM pré-entraîné pour apprendre l'alignement de leurs espaces latents afin d'effectuer une interpolation de l'espace latent, puis générer les molécules cibles par décodage. Le diagramme de processus est le suivant.
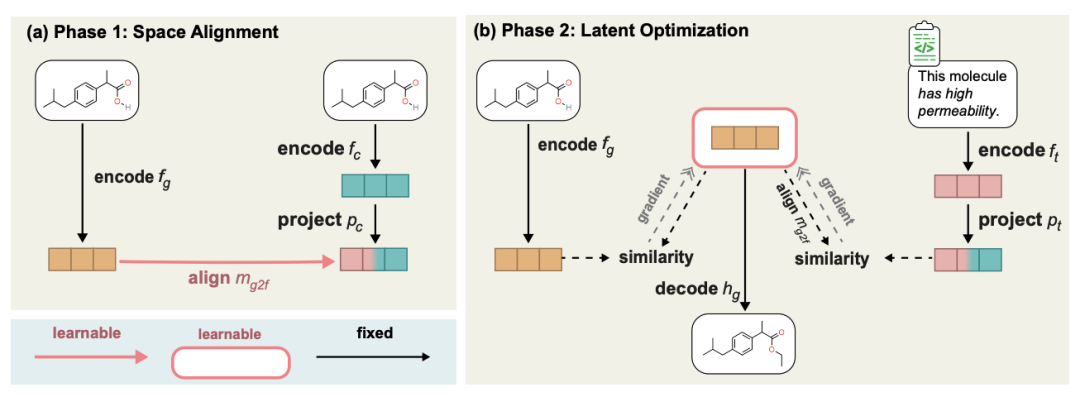
Le contenu qui doit être réécrit est : un diagramme de processus en deux étapes d'édition moléculaire guidée par texte à échantillon nul
Nous montrons ici les résultats qualitatifs de plusieurs groupes d'édition moléculaire, reformulés comme suit : (Le les détails des résultats des tâches restantes en aval peuvent être consultés (voir le document original). Nous considérons principalement quatre types de tâches d'édition moléculaire :
- Édition d'un seul attribut : édition d'un seul attribut, tel que la solubilité dans l'eau, la pénétrabilité et le nombre de donneurs et d'accepteurs de liaisons hydrogène.
- Modification des attributs composites : modifiez plusieurs attributs en même temps, tels que la solubilité dans l'eau et le nombre de donneurs de liaisons hydrogène.
- Édition de similarité de médicament : (Annexe D.5) consiste à rapprocher la molécule d'entrée et la molécule cible du médicament.
- Recherche voisine de médicaments brevetés : pour les médicaments brevetés, les médicaments intermédiaires sont souvent déclarés ensemble. Ce que nous faisons ici, c'est combiner le médicament intermédiaire avec la description en langage naturel pour voir s'il peut générer le médicament cible final.
- éditeur d'affinité de liaison : nous avons sélectionné plusieurs tests ChEMBL comme cibles, dans le but d'avoir une affinité de liaison plus élevée entre les molécules d'entrée et les cibles.
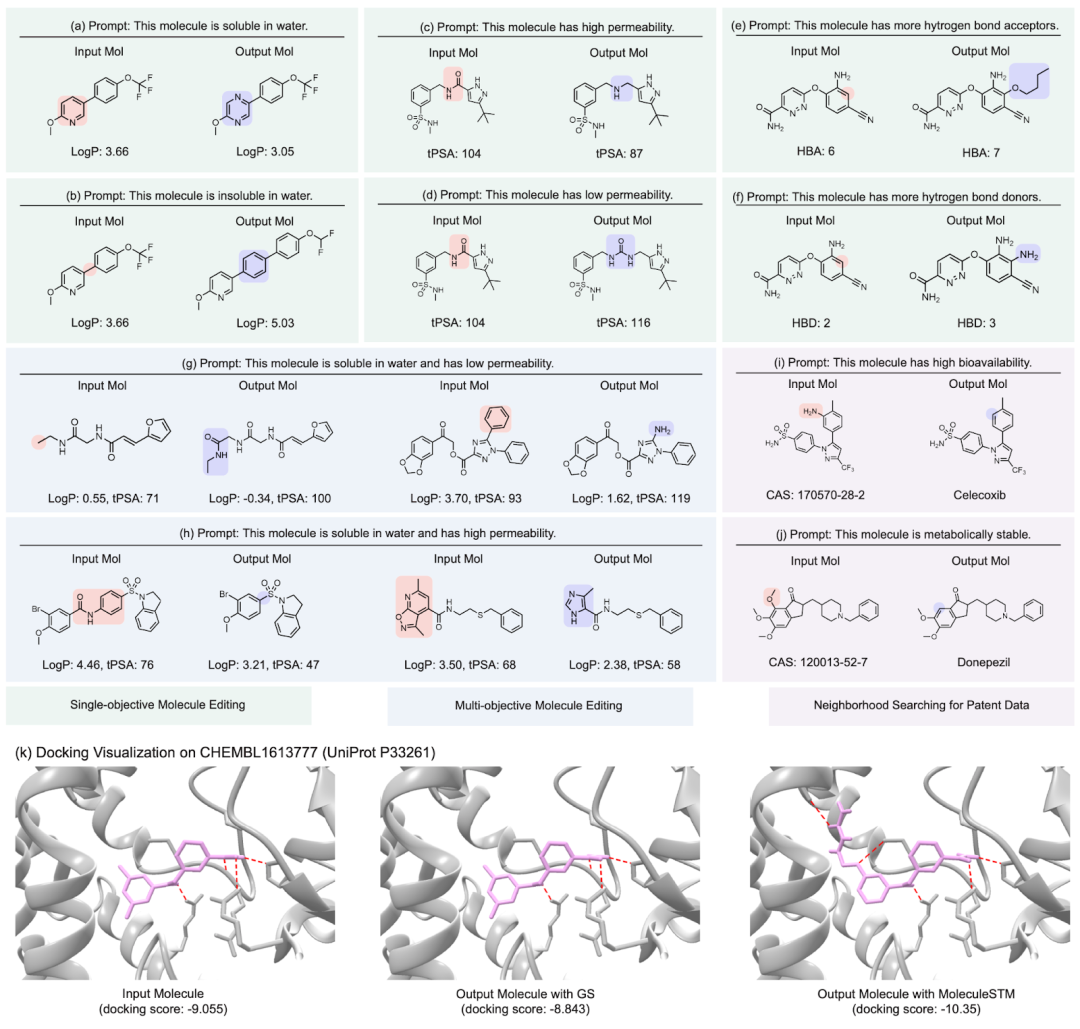
Affichage des résultats : édition de molécules guidée par texte sans échantillon. (Remarque : il s'agit d'une traduction directe de la phrase originale en chinois.)
Ce qui est plus intéressant est le dernier type de tâche. Nous avons constaté que MoleculeSTM peut effectivement effectuer une correspondance de ligand sur la base de la description textuelle du composé principal. optimisation. (Remarque : les informations sur la structure des protéines ici ne seront connues qu'après évaluation.)
Ce qui précède est le contenu détaillé de. pour plus d'informations, suivez d'autres articles connexes sur le site Web de PHP en chinois!

