Maison >Périphériques technologiques >IA >La nouvelle méthode d'analyse et de génération de données multimodales de l'équipe UW-chinoise JAMIE améliore considérablement les capacités de prédiction du type de cellule et de sa fonction
La nouvelle méthode d'analyse et de génération de données multimodales de l'équipe UW-chinoise JAMIE améliore considérablement les capacités de prédiction du type de cellule et de sa fonction
- WBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBavant
- 2023-06-10 14:43:291499parcourir
Ces dernières années, avec le développement rapide de la technologie unicellulaire, nous avons pu mesurer diverses caractéristiques de cellules uniques pour obtenir des données multimodales unicellulaires (telles que scRNA-seq, scATAC-seq, Patch-seq ).
Ces données nous aident à mieux comprendre les fonctions cellulaires et les mécanismes moléculaires. Par exemple, les chercheurs ont récemment utilisé des méthodes d’apprentissage automatique pour analyser la relation entre les données multimodales unicellulaires afin de comprendre les mécanismes biologiques impliqués dans les types de cellules et les maladies.
Cependant, l'acquisition de données multimodales unicellulaires est souvent coûteuse et une perte modale se produit souvent. Les méthodes d'apprentissage automatique existantes nécessitent généralement des données multimodales entièrement adaptées pour le remplissage et l'intégration des données, et ne conviennent pas aux situations dans lesquelles les modalités manquent.
Afin de résoudre ce problème, le laboratoire de Wang Daifeng de l'Université du Wisconsin-Madison a développé une méthode d'apprentissage automatique open source basée sur des auto-encodeurs variationnels conjoints - Joint Variational Autoencoders for Multimodal Imputation and Embedding (JAMIE).
JAMIE peut être utilisé pour l'analyse intégrée de données multimodales unicellulaires, telles que l'alignement des données, l'intégration et le remplissage des données manquantes afin de mieux prédire les types et les fonctions des cellules.
Ce travail a été récemment publié dans "Nature Machine Intelligence".

Adresse papier : https://www.nature.com/articles/s42256-023-00663-z
Adresse du projet : https://github.com/daifengwanglab /JAMIE
Introduction à la méthode JAMIE
JAMIE entraîne un modèle d'auto-encodeur variationnel conjoint réutilisable pour améliorer la modalité unique en projetant les données multimodales disponibles séparément dans des espaces latents similaires. Capacité à déduire des modèles d'état.
Comme le montre la figure 1, pour effectuer une imputation multimodale, JAMIE alimente les données dans un encodeur puis traite les résultats de l'espace latent via le décodeur opposé.
JAMIE combine la génération d'espace latent réutilisable et flexible d'auto-encodeurs avec l'estimation automatique de la correspondance des méthodes d'alignement, permettant le traitement de données multimodales avec une correspondance incomplète.
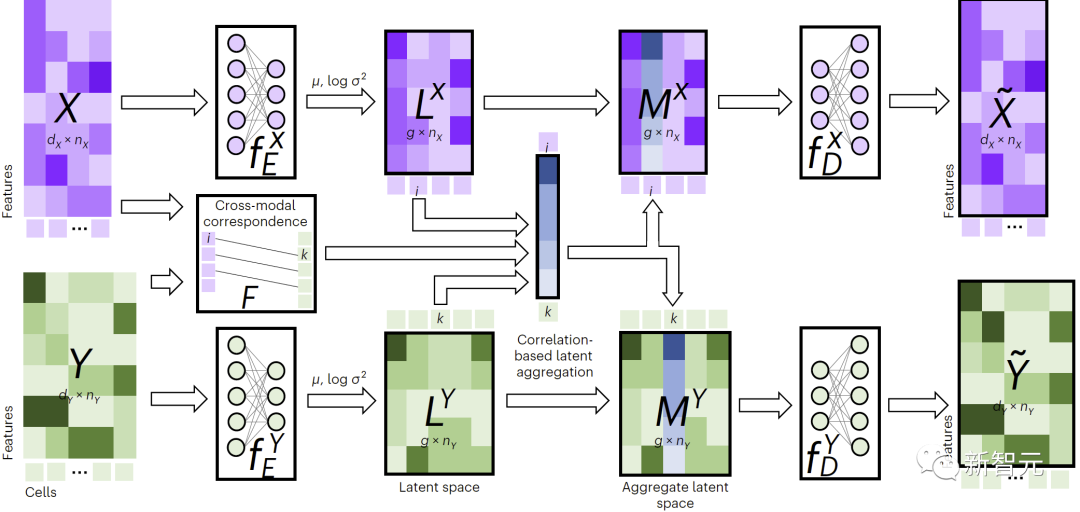
Figure 1. Présentation de la méthode JAMIE
Plus précisément, JAMIE peut être divisé en deux étapes suivantes :
- Prétraitement des données d'entrée. En prenant le mode bimodal comme exemple, supposons que les matrices de données correspondant aux modes sont et respectivement. Notez que les dimensions et la somme des caractéristiques peuvent être différentes, ainsi que le nombre d'échantillons. Le prétraitement normalise chaque ligne de chaque matrice pour avoir une moyenne de 0 et une variance de 1. S'il existe des données correspondantes, l'utilisateur peut fournir une matrice de corrélation modale pour améliorer les performances, ce qui signifie que le ème échantillon du modal correspond complètement au ème échantillon du modal, signifie qu'il n'y a pas de correspondance connue et signifie qu'il y a une partie. correspondance.
- Utilisez l'auto-encodeur variationnel conjoint pour connaître l'espace latent de similarité de chaque modalité : et , où (par défaut, réglable par l'utilisateur) est la dimension de l'espace latent. Pendant le processus de formation, JAMIE minimise la fonction de perte suivante :
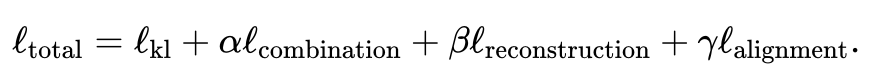
La fonction de perte totale contient quatre éléments.
Le premier élément calcule la divergence Kullback-Leibler (KL) entre la distribution déduite par l'auto-encodeur variationnel et la distribution normale standard multivariée, ce qui aide à maintenir la continuité de l'espace latent ; les échantillons correspondants ; le troisième terme est la somme des erreurs quadratiques moyennes entre la matrice de données reconstruite et la matrice de données d'origine ; le quatrième terme utilise la correspondance intermodale déduite pour ajuster l'espace latent généré ;
Pour les expressions spécifiques de chaque élément, veuillez consulter le texte original de l'article. Les poids des deuxième, troisième et quatrième éléments par rapport au premier élément peuvent être ajustés par l'utilisateur. JAMIE fournit également des poids par défaut adaptés aux situations courantes.
Le tableau suivant montre la comparaison du modèle et de la portée applicable de JAMIE avec les méthodes de pointe actuelles. JAMIE unifie les fonctionnalités de plusieurs méthodes d'intégration et d'interpolation différentes dans une seule architecture, permettant ainsi l'interpolation des modalités manquantes, permettant la compatibilité des données non omiques et la capacité de gérer des données multimodales avec seulement des avantages de correspondance partielle.
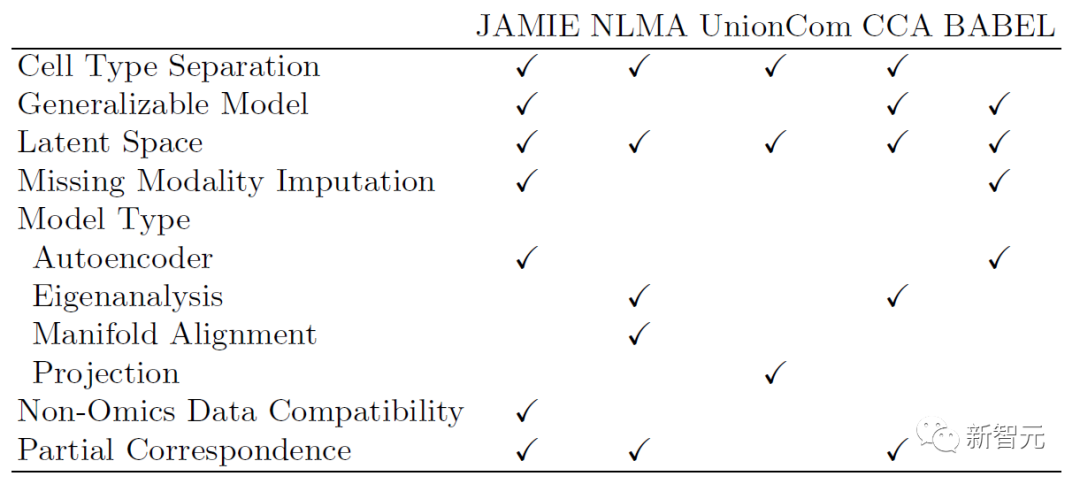
Tableau 1. Comparaison des différentes méthodes d'intégration multimodale et de remplissage modal manquant. Grâce à une architecture unique, JAMIE intègre des fonctionnalités de plusieurs méthodes d'intégration et d'interpolation différentes. NLMA : Nonlinear Manifold Alignment [15], UnionCom [7], CCA : Canonical Correlation Analysis [15, 16], BABEL [5].
Principales applications de JAMIE
Intégration et prédiction phénotypique de données multimodales
L'intégration de données multimodales peut améliorer les performances de classification, améliorer la connaissance phénotypique et la compréhension de mécanismes biologiques complexes.
Étant donné deux ensembles de données et les relations correspondantes, JAMIE peut générer des données spatiales latentes, basées sur l'encodeur formé et, et effectuer un clustering ou une classification basée sur .
Le clustering basé sur des données d'espace latent présente plusieurs avantages, tels que l'intégration des deux modalités dans la génération de fonctionnalités. JAMIE peut alors prédire les correspondances d'échantillons, telles que la prédiction du type de cellule.
Pour les ensembles de données partiellement étiquetés, les cellules du même cluster doivent avoir des types similaires.
JAMIE sépare les caractéristiques des différents types de données dans le processus de génération de données spatiales latentes, de sorte que des algorithmes de clustering ou de classification complexes ne sont généralement pas nécessaires pour obtenir de meilleurs résultats.
Pour les données de grande dimension, JAMIE utilise UMAP [32] pour la visualisation du regroupement de types de cellules.
Imputation de données multimodales
De nombreuses méthodes d'imputation multimodales actuelles ne peuvent pas démontrer qu'elles ont appris les mécanismes biologiques sous-jacents à des fins d'imputation.
Par rapport aux réseaux de rétroaction ou aux méthodes de régression linéaire, JAMIE peut mieux apprendre les mécanismes biologiques sous-jacents pour prédire les données manquantes sur la base de fondements mathématiques plus rigoureux.
La figure 2 montre le processus de JAMIE pour le remplissage de données intermodales. JAMIE entraîne d'abord les modèles d'encodage et de décodage sur les données d'entraînement.
Pour les nouvelles données, JAMIE utilise d'abord l'encodeur appris à partir des données pour les projeter dans l'espace latent pour obtenir, puis les obtient en agrégeant les caractéristiques de l'espace latent, et enfin les décode en données de motif manquantes via le décodeur correspondant.
JAMIE utilise l'espace latent pour prédire la correspondance entre les cellules, ce qui peut aider à comprendre la relation entre les caractéristiques des données et les phénotypes.
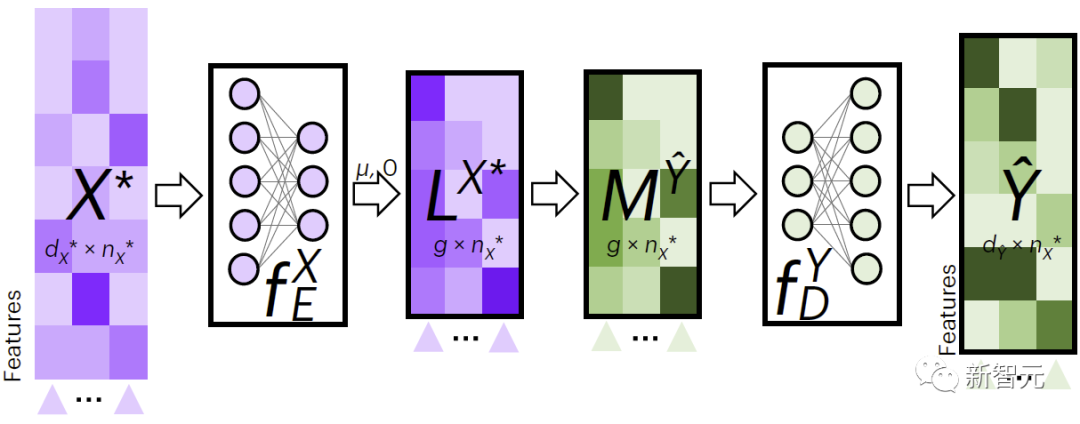
Figure 2. Interpolation multimodale JAMIE
Explication des caractéristiques de l'espace latent et des caractéristiques de remplissage
Pour expliquer le modèle formé , JAMIE adopte SHAP (SHapley Explications additives)[18].
SHAP évalue l'importance des caractéristiques d'entrée individuelles en modulant par échantillonnage les prédictions individuelles générées par le modèle. Cela peut être utilisé pour une variété d’applications intéressantes.
Si la variable cible peut être facilement séparée par phénotype, SHAP peut identifier les caractéristiques pertinentes pour une étude plus approfondie. De plus, si nous effectuons une imputation, SHAP peut révéler les connexions intermodales apprises par le modèle.
À partir d'un modèle et d'un échantillon, apprenez la valeur SHAP telle que où se trouve le vecteur de caractéristique d'arrière-plan.
Si , alors la somme des valeurs SHAP et de la sortie en arrière-plan sera égale à , chacune étant proportionnelle à l'impact sur la sortie du modèle.
Une autre technique utile consiste à sélectionner une métrique clé pour la classification (par exemple, LTA [7, 19]) ou l'imputation (par exemple, correspondance entre les caractéristiques imputées et les caractéristiques mesurées) et à l'utiliser une par une dans le modèle. évalué en supprimant (en remplaçant) chaque fonctionnalité par une valeur d'arrière-plan.
Ensuite, si la métrique clé s'aggrave, cela indique que les fonctionnalités supprimées sont plus importantes pour les résultats du modèle.
Résultats expérimentaux
JAMIE a utilisé quatre ensembles de données multimodales unicellulaires couramment utilisés pour la vérification.
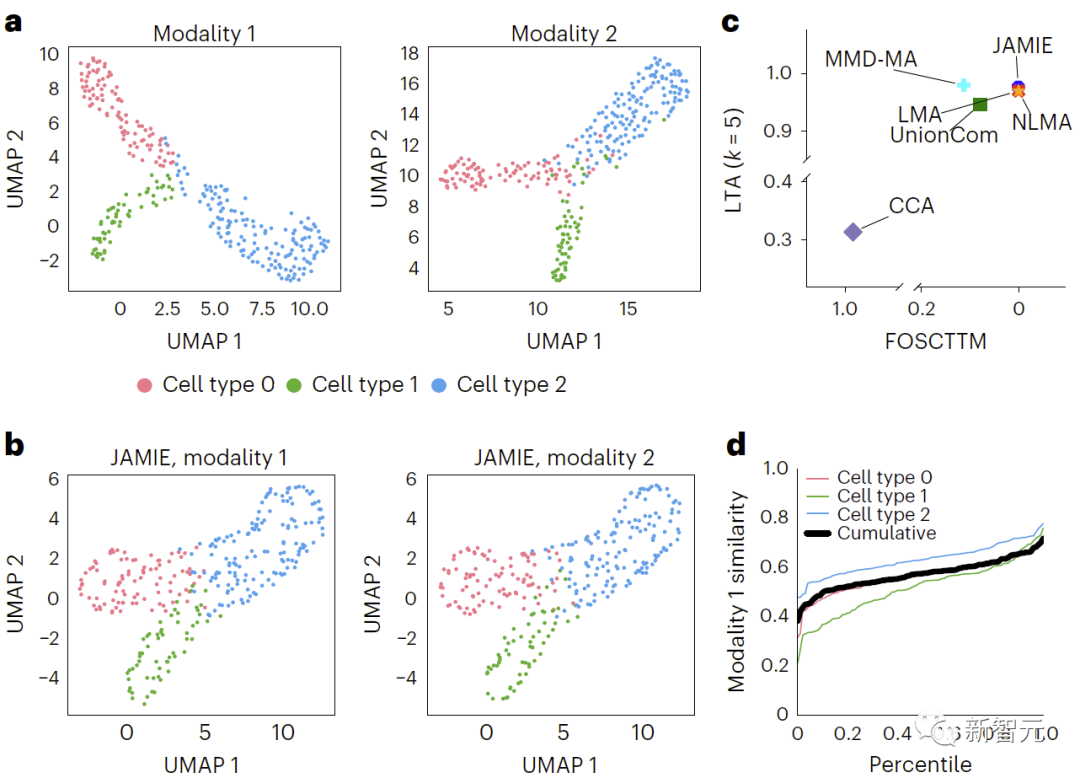
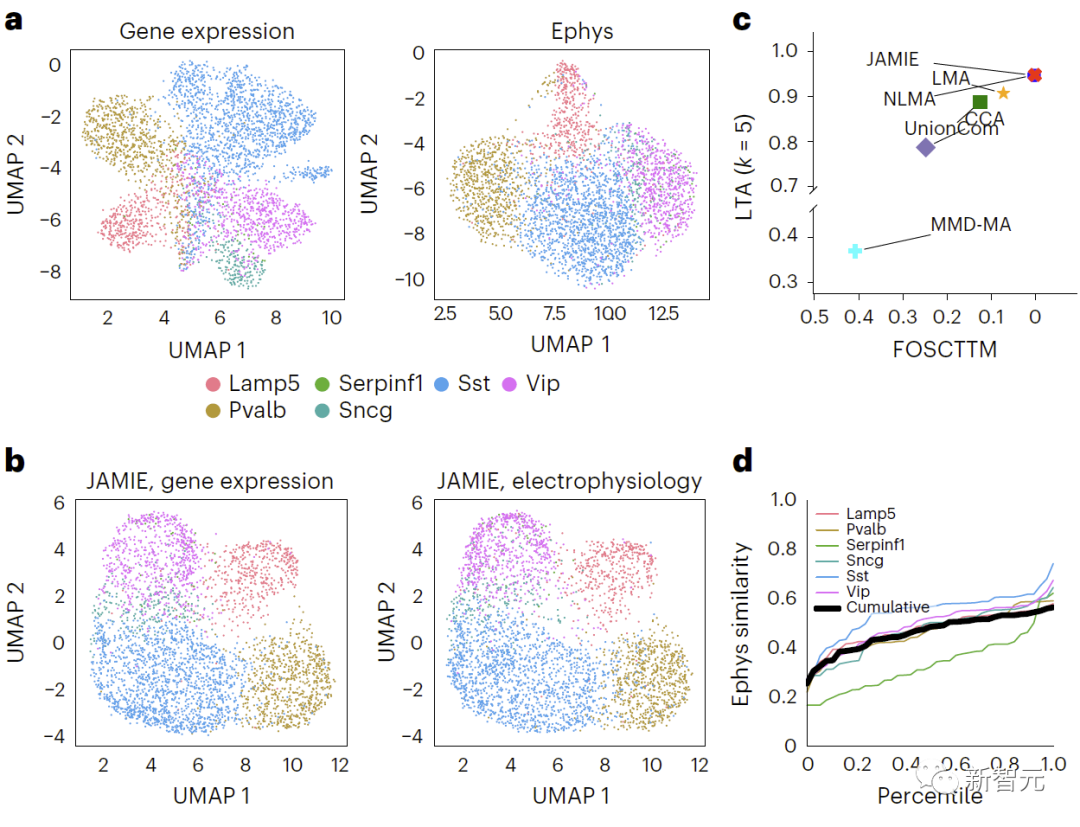
(1) Données multimodales simulées (300 échantillons, 3 types de cellules) générées par échantillonnage de distribution gaussienne de variétés ramifiées de MMD-MA ; (2) à partir du cortex visuel de souris (expression du gène Patch-seq et électrophysiologique) données de caractérisation de cellules neuronales uniques dans le cortex moteur de souris (1 208 échantillons, 9 types de cellules) et le cortex moteur de souris (1 208 échantillons, 9 types de cellules
(3) à partir de 10 fois l'expression de gènes multi-omiques unicellulaires et la chromatine ; données d'accessibilité pour 8 981 échantillons dans le cerveau humain en développement (21 semaines de gestation, couvrant 7 types de cellules majeurs du cortex cérébral humain
(4) expression du gène scRNA-seq et données d'accessibilité de la chromatine scATAC-seq de 4 301 cellules de la lignée cellulaire d'adénocarcinome du côlon COLO-320DM.
L'évaluation a révélé que JAMIE est nettement meilleur que les autres méthodes (comparaison des résultats des données de simulation de collecteurs de branches de MMD-MA dans la figure 3 et comparaison des résultats des données du cortex visuel de la souris dans la figure 4) et donne la priorité au multimodal. remplissage de fonctionnalités importantes tout en fournissant potentiellement de nouvelles informations mécanistiques à résolution cellulaire.
Résumé
En résumé, JAMIE est un nouveau modèle de réseau neuronal profond pour la prédiction intégrée de données multimodales unicellulaires.Il convient aux données multimodales complexes, mixtes ou partiellement correspondantes, mises en œuvre via une nouvelle méthode d'agrégation d'intégration latente qui repose sur une structure d'auto-encodeur variationnel conjoint (VAE). En plus des performances supérieures mentionnées ci-dessus, JAMIE dispose également de capacités informatiques efficaces et d'une faible utilisation de la mémoire. De plus, les modèles pré-entraînés et les intégrations latentes multimodales apprises peuvent être réutilisés dans les analyses en aval.
Bien sûr, pour des ensembles de données plus volumineux, la formation des auto-encodeurs variationnels (VAE) prend beaucoup de temps. Par conséquent, les méthodes de sélection de fonctionnalités précédentes, telles que la PCA automatique dans JAMIE, contribuent à réduire les délais. Étant donné que la VAE utilise la perte de reconstruction, le prétraitement des données est également crucial pour éviter que des fonctionnalités volumineuses ou répétées n'affectent de manière disproportionnée les fonctionnalités intégrées de faible dimension. Pour une imputation multimodale spécifique, la diversité de l'ensemble de données de formation doit être soigneusement prise en compte pour éviter de biaiser le modèle final et d'avoir un impact négatif sur sa capacité de généralisation. JAMIE peut également potentiellement être étendu pour aligner des ensembles de données provenant de différentes sources plutôt que de différentes modalités, telles que les données d'expression génique dans différentes conditions.
Introduction aux auteurs
Les auteurs de l'article sont Noah Cohen Kalafut (étudiant au doctorat au Département d'informatique), Huang Xiang (chercheur principal) et Wang Daifeng (PI) sont affiliés au Département de biostatistique et d'informatique médicale, Département d'informatique, Université du Wisconsin-Madison et Weisman Research Center. L'auteur correspondant est le professeur Wang Daifeng.
Fondé en 1973, le Centre Weisman fait progresser la recherche sur le développement humain, les troubles neurodéveloppementaux et les maladies neurodégénératives depuis un demi-siècle.
Ce qui précède est le contenu détaillé de. pour plus d'informations, suivez d'autres articles connexes sur le site Web de PHP en chinois!
Articles Liés
Voir plus- Tendances technologiques à surveiller en 2023
- Comment l'intelligence artificielle apporte un nouveau travail quotidien aux équipes des centres de données
- L'intelligence artificielle ou l'automatisation peuvent-elles résoudre le problème de la faible efficacité énergétique des bâtiments ?
- Co-fondateur d'OpenAI interviewé par Huang Renxun : les capacités de raisonnement de GPT-4 n'ont pas encore atteint les attentes
- Bing de Microsoft surpasse Google en termes de trafic de recherche grâce à la technologie OpenAI

