Maison >Périphériques technologiques >IA >Application des modèles DNN multimodaux aux tâches de prédiction des interactions médicamenteuses
Application des modèles DNN multimodaux aux tâches de prédiction des interactions médicamenteuses
- WBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBavant
- 2023-05-31 11:01:311246parcourir

1. Introduction au contexte
Tout d'abord, permettez-moi de partager avec vous le contexte pertinent de la découverte de médicaments.
1. Introduction générale
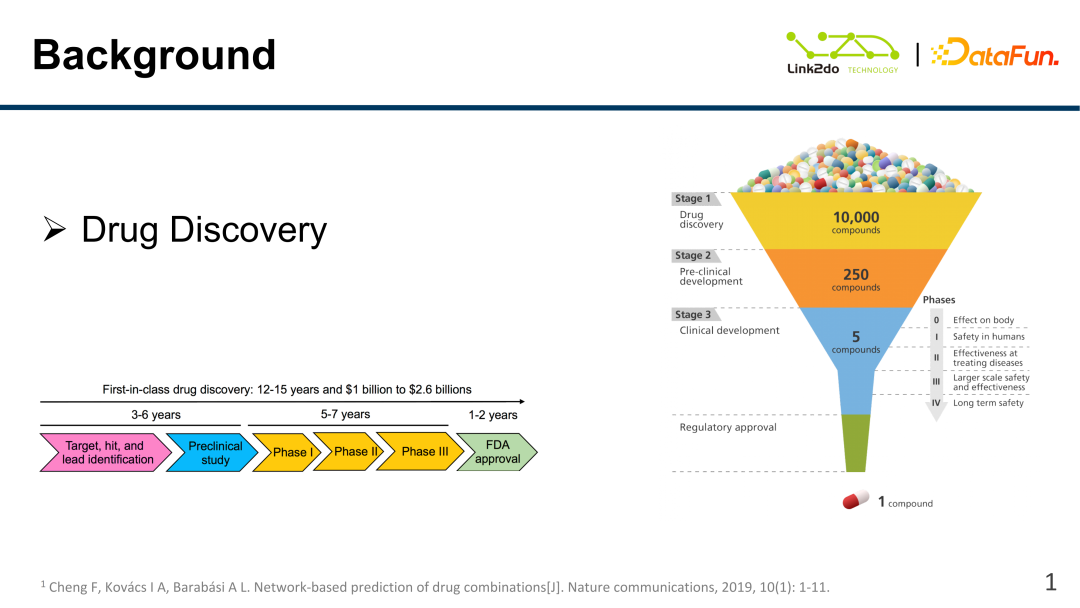
Le domaine de la découverte de médicaments a été très en vogue ces dernières années, en particulier l'utilisation de l'IA pour aider les sociétés pharmaceutiques dans leur travail de découverte de médicaments, y compris la recherche sur les médicaments. et le développement. Le processus de recherche et développement de médicaments a un cycle très long. Habituellement, le processus de recherche et développement du premier médicament pour un certain type de maladie clinique nécessite des milliards de fonds et plus de dix ans. Elle est principalement divisée selon les étapes suivantes :
(1) Recherche sur les cibles de la maladie et identification des protéines centrales de la maladie.
(2) Vérifier l'efficacité du médicament avant les essais cliniques : y compris la recherche sur la toxicité du médicament, son efficacité, les méthodes de prise, etc.
(3) Essais cliniques.
(4) Approbation et certification FDA.
Le cycle traditionnel du processus de développement de médicaments est donc très long. De plus, sur la reconnaissance initiale de plus de 10 000 médicaments, 5 médicaments sont entrés dans la phase d'essai clinique et finalement un seul médicament a été approuvé pour la commercialisation. Dans ce contexte, comment aider les sociétés pharmaceutiques à sélectionner plus rapidement des médicaments efficaces parmi les médicaments candidats, et comment pré-étudier rapidement l'impact, le rôle, l'efficacité, etc. des médicaments dans la phase d'essai clinique est devenu un sujet de recherche brûlant. , dans lequel l'IA, en particulier la technologie des réseaux neuronaux profonds, peut considérablement accélérer le processus de recherche et de développement de médicaments.
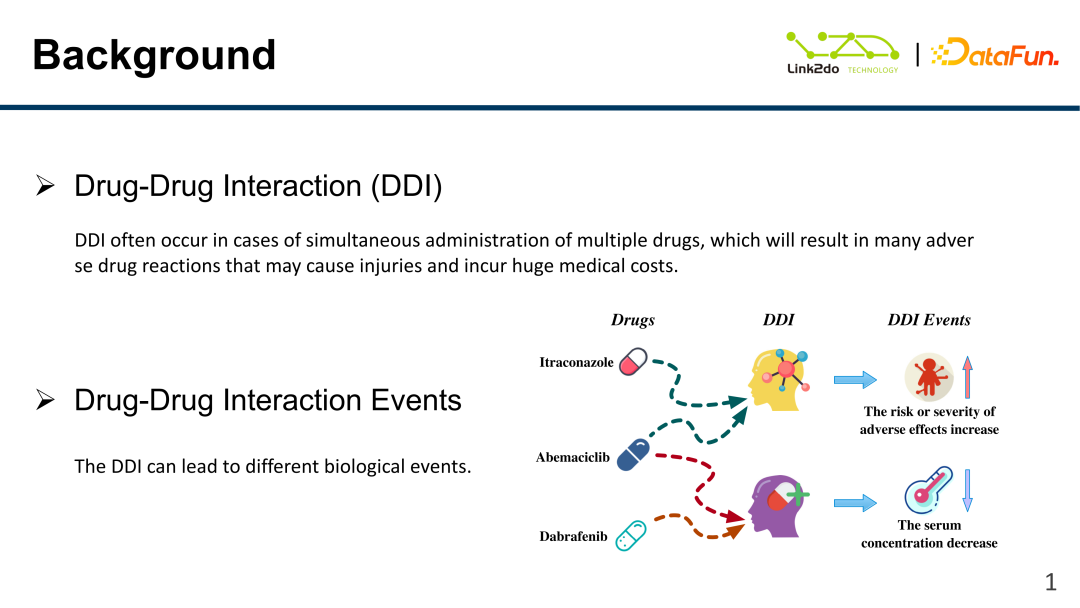
Le contenu partagé aujourd'hui ne concerne pas le dépistage des médicaments. Il se concentre principalement sur les effets secondaires et l'efficacité des médicaments candidats. L'objectif principal de la recherche est de réduire la toxicité des médicaments et d'améliorer leur efficacité.
Comme le montre l'image ci-dessus, le DDI (Drug-Drug Interaction) fait référence à l'interaction entre les médicaments. Réaliser des analyses croisées entre les médicaments de recherche et les médicaments existants pour découvrir les effets secondaires des médicaments de recherche, tels que leurs effets sur l'organisme, etc., et les découvrir et les classer à l'avance grâce à des expériences. Pour un exemple simple, « le médicament est divisé en trois parties par poison ». Où se reflète principalement la toxicité du médicament ? Dans de nombreux cas, cela se produit lorsqu’un médicament est associé à d’autres médicaments, une interaction chimique entre deux ou plusieurs médicaments. L'image dans le coin inférieur droit montre trois médicaments. L'itraconazole est un médicament lié aux tumeurs. S'il est mélangé à l'abémaciclib, il provoquera des effets secondaires graves, tels qu'une insuffisance hépatique, une insuffisance hépatique et rénale. les patients peuvent avoir des conséquences graves. Si l'abéciclib et le dabrafenib sont mélangés, cela entraînera une diminution de la concentration sérique et provoquera d'autres maladies. Par conséquent, un grand nombre de tests sont nécessaires dans le processus de développement de nouveaux médicaments, mais il est impossible d'utiliser de vraies personnes pour tester et ne peuvent être testés qu'avec des souris ou d'autres animaux.
Le contenu partagé aujourd'hui consiste à utiliser des réseaux neuronaux multimodaux pour prédire à l'avance le DDI d'un médicament en fonction des ingrédients médicamenteux existants (y compris en cours de développement et connus), des allergies, etc.
2. Question
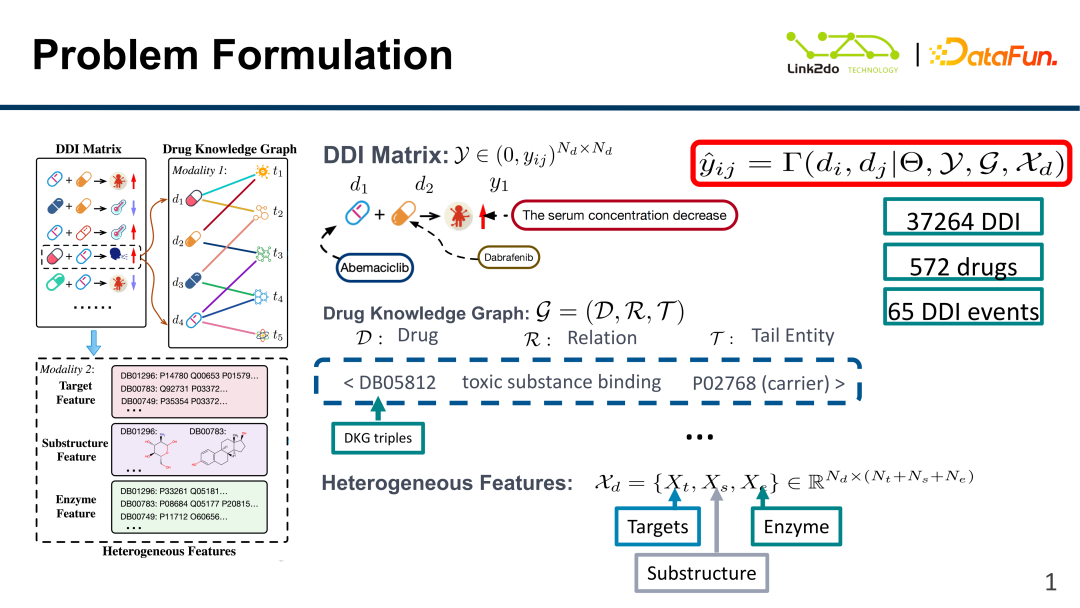
Comme le montre la figure ci-dessus, les interactions médicamenteuses peuvent être résumées sous la forme d'une matrice DDI. La matrice décrit les résultats des interactions médicamenteuses. Par exemple, les médicaments d1 (abexiclib) et d2 (dabrafenib) entraîneront y1 (diminution de la concentration sérique). ). Cette étude comprenait 37 264 données DDI, impliquant 572 médicaments (d) et 65 résultats de réactions (y, comme une diminution de la concentration sérique, etc.). Et sur la base de ces données, un Drug Knowledge Graph (DKG) a été construit : les nœuds sont les médicaments et les bords sont les relations entre les médicaments. Le triple DKG est {D : médicament, R : relation inter-médicaments, T : entité de queue}.
En plus des données ci-dessus, le modèle multimodal intègre également les caractéristiques hétérogènes du médicament (HF, caractéristiques hétérogènes) : {Cible : cible, sous-structure : composant/structure chimique, enzyme : enzyme}, chaque les dimensions de chaque caractéristique sont différentes, par exemple, l'information cible est une protéine. Enfin, les matrices DDI, DKG et HF sont fusionnées sur la même distribution de probabilité pour la modélisation.
2. Introduction au modèle MDNN
Ensuite, nous présenterons le cadre du modèle MDNN multimodal hétérogène.
1. Cadre global MDNN
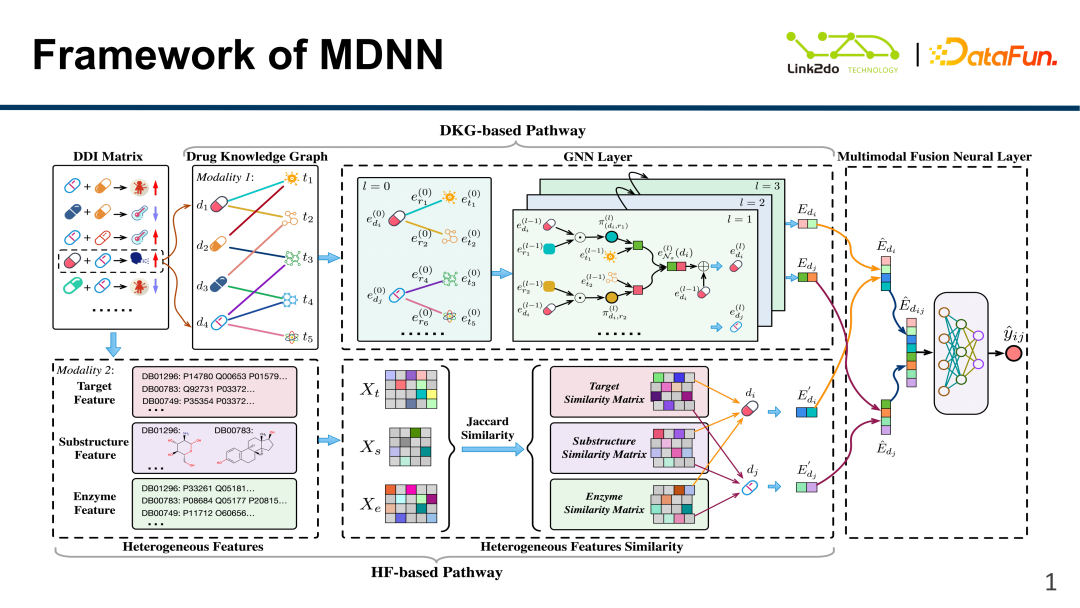
Ce modèle est appelé MDNN Les données de base sont principalement divisées en deux parties : la matrice DDI et les données hétérogènes. les trois parties suivantes :
(1) Basé sur la partie DKG : Exprimer principalement des informations sur les ingrédients du médicament lui-même (principes actifs, ingrédients toxiques), les relations entre les médicaments, etc. en construisant un médicament graphe de connaissances.
(2) Basé sur la partie HF : Décrire les informations caractéristiques de base du médicament lui-même en intégrant des données caractéristiques hétérogènes telles que des cibles, des enzymes et des structures moléculaires.
(3) Réseau neuronal de fusion multimodale : Fusionnez efficacement les deux parties des données de caractéristiques, DKG et HF, et effectuez une modélisation unifiée des données fusionnées.
2. Construction du module DKG
Ce qui suit présentera le processus de construction basé sur DKG.
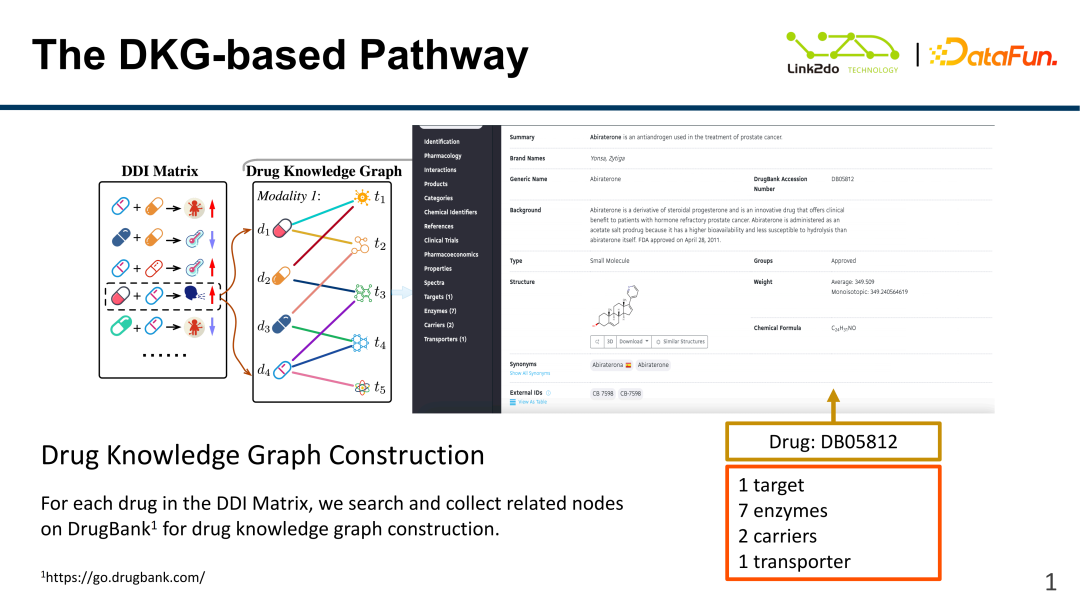
L'image ci-dessus montre le contenu principal du graphique de connaissances sur les médicaments. L'image de gauche est la matrice DDI. La matrice contient près de 600 médicaments. Les ingrédients et les informations sur les actions de ces médicaments sont stockés. la base de données (DrugBank, c'est-à-dire « Drug Bank »). L'image de droite montre un exemple d'informations sur les médicaments dans la « Banque de médicaments », telles que les enzymes, les supports, les cibles et d'autres caractéristiques de base hétérogènes. En prenant comme exemple le médicament DB05812, en plus de la cible. , En plus des enzymes et des structures moléculaires, il existe également des porteurs et des transporteurs. Cependant, ces deux types de données sont relativement rares et n'ont pas autant de dimensions que les autres caractéristiques. La distinction dans l'ensemble de données actuel n'est donc pas grande. ces deux données ne sont pas utilisées pour le moment. Les principales données utilisées sont les cibles, les enzymes et les structures moléculaires.
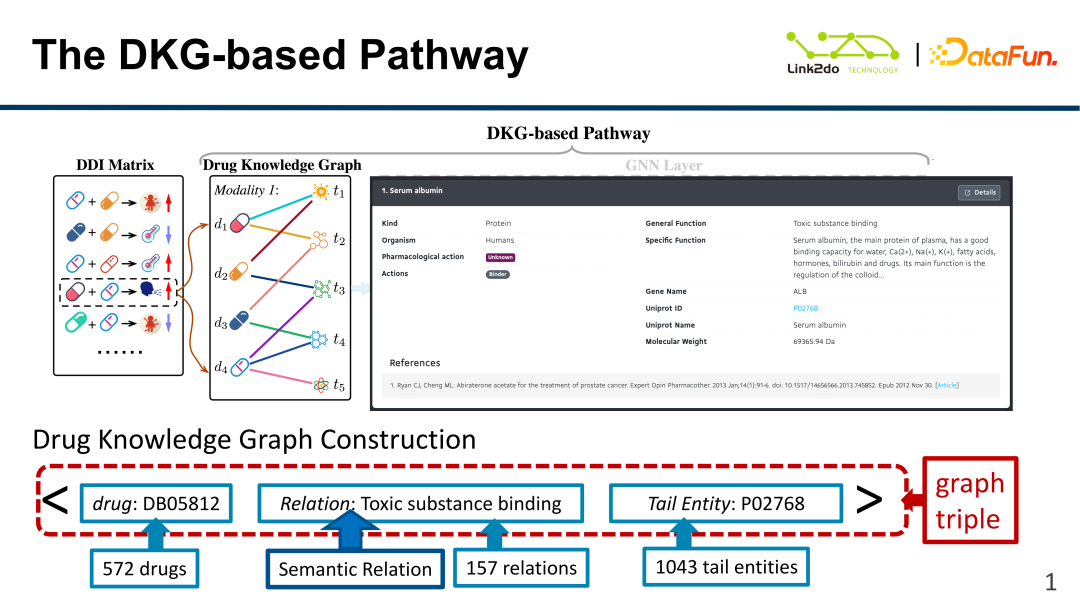
Comme le montre la figure ci-dessus, le graphe de connaissances est principalement composé de nœuds et de bords, où les nœuds sont des médicaments et des ingrédients, et les bords sont des relations. La relation affichée par le triplet dans l'exemple est la relation entre les composants toxiques, c'est-à-dire qu'il existe une relation entre les composants toxiques entre le médicament nodal « DB05812 » et le composant nodulaire « P02768 ». Sur la base des triplets de relations entre ingrédients médicamenteux obtenus auprès de la « Banque de médicaments », le graphe de connaissances DKG est formé, qui contient 572 types de médicaments. Les bords (relations) des triplets sont appelés relations sémantiques. Il existe au total 157 types de relations. Ingrédients Il existe 1043 types de nœuds d'entité de queue. Chaque DKG peut extraire les informations correspondantes de la « Banque de médicaments » en fonction des exigences de la tâche et les construire, de sorte que le DKG est équivalent à un sous-graphique dans le graphique de connaissances « Banque de médicaments ».
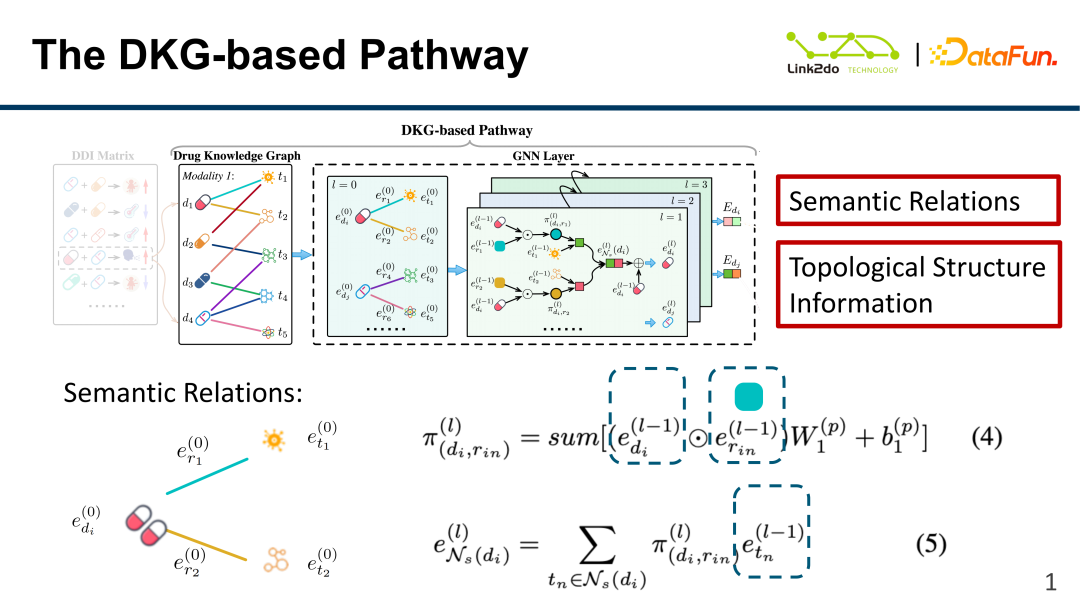
Sur la base du DKG, deux types d'informations sont résumés. La figure ci-dessus montre la construction du modèle d'information sur la relation sémantique. Sur la base des composants toxiques, calculez d'abord le produit interne du médicament (d) et la relation (r) dans la couche précédente, et résumez-le via le poids actuel de la couche (W1) pour obtenir la fonction π, c'est-à-dire le les informations de bord et de nœud du médicament sont additionnées via la fonction π, puis effectuent une sommation pondérée de la fonction π et du composant de couche précédent (t) pour obtenir e, c'est-à-dire que les informations de bord sont obtenues.
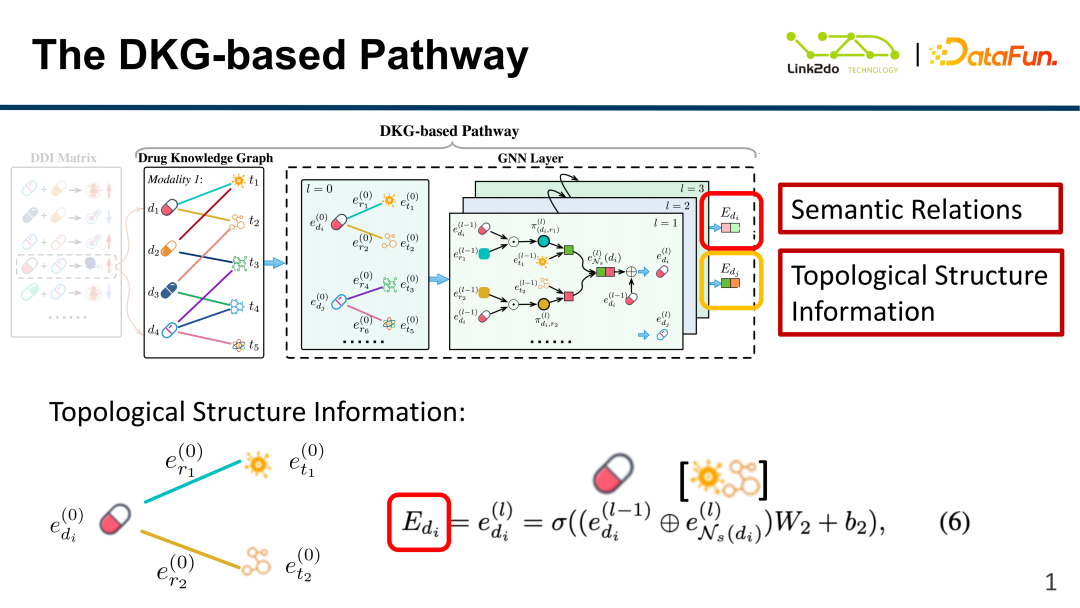
De même, la figure ci-dessus montre la construction du modèle d'informations de structure topologique du graphe. En plus des composants toxiques, les médicaments peuvent également contenir plusieurs autres relations de composants (bords, c'est-à-dire e) du même médicament et leurs poids correspondants W2 peuvent être connectés pour finalement obtenir le E correspondant à chaque médicament. Grâce à la méthode ci-dessus, les arêtes DKG et les informations de structure topologique sont efficacement fusionnées et représentées.
3. Construction du module HF
Comme indiqué ci-dessous, en plus des informations secondaires et des informations DDI mentionnées ci-dessus, les médicaments disposent également d'informations multimodales très riches : un même médicament peut cibler plusieurs cibles. Différents médicaments ont également des structures moléculaires différentes, qui représentent leurs propriétés moléculaires correspondantes ; les médicaments se lieront à différentes cibles sous l'action de différentes enzymes. Ces trois types d'informations sont vectorisées, puis la similarité entre médicaments est mesurée par simple similarité Jaccard pour obtenir la matrice de similarité correspondante.
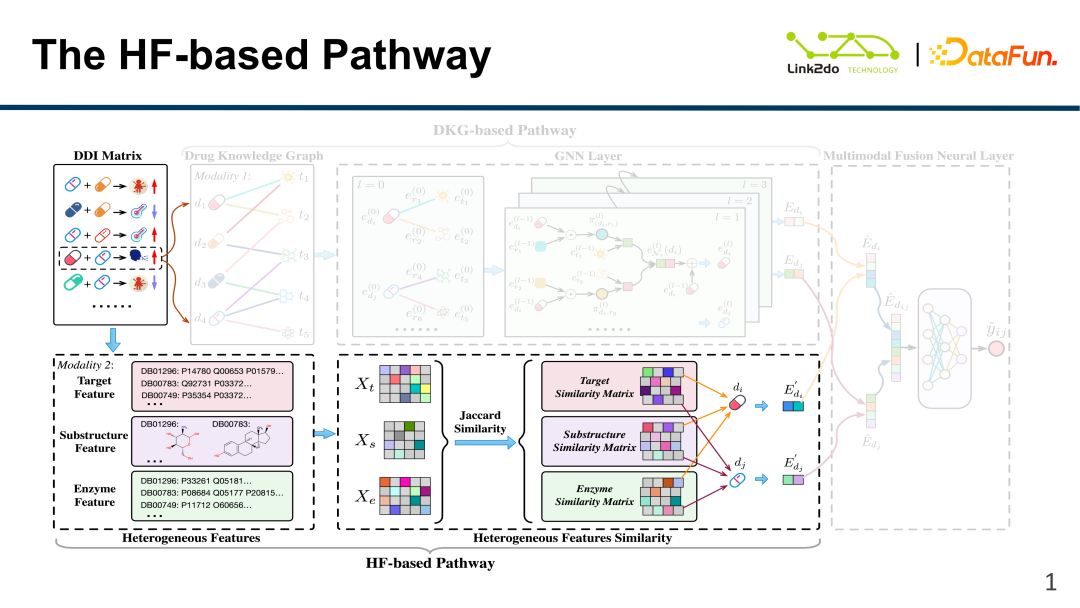
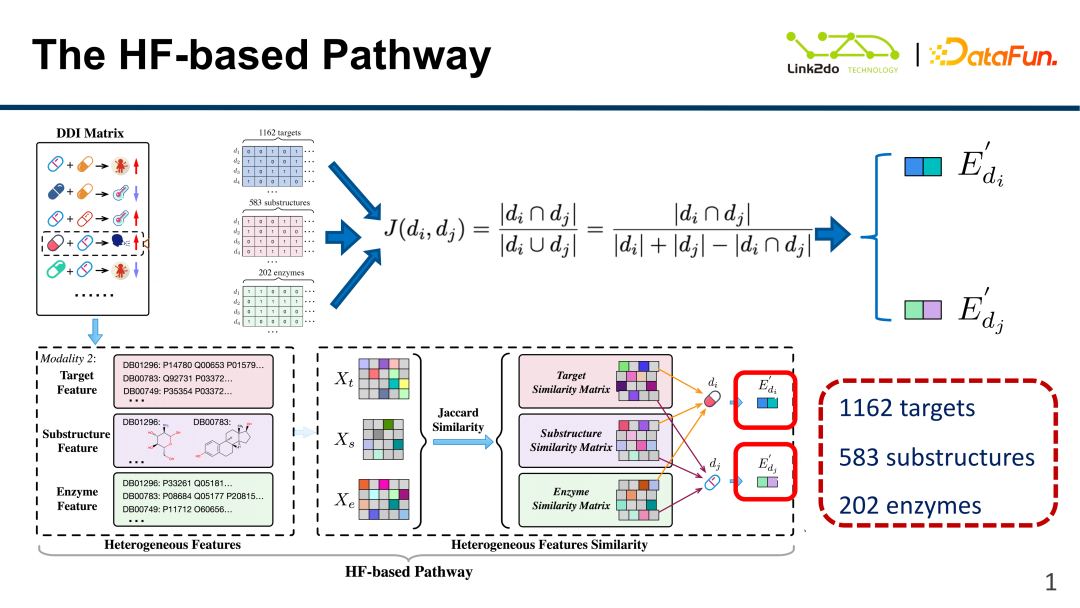
Enfin, les trois matrices de similarité sont fusionnées pour obtenir le E' correspondant à chaque médicament, c'est-à-dire que l'information basée sur les caractéristiques isomères du médicament est obtenue. La dimension de ce vecteur de caractéristiques est également petite, comprenant des informations sur 1 162 cibles, 583 structures et 202 enzymes.
4. Couche de fusion multimodèle
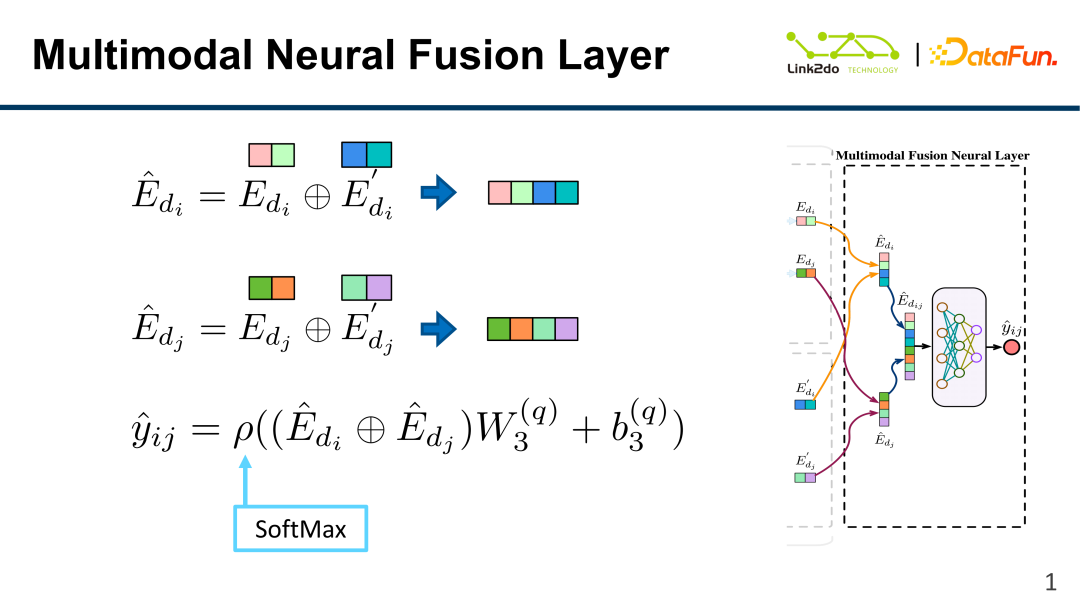
Comme le montre la figure ci-dessus, le résultat DKG E et le résultat HF E' de chaque médicament sont finalement épissés et fusionnés. la couche de fusion pour obtenir :
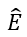
Récupérez ensuite la couche de sortie grâce à la fonction softmax :
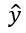
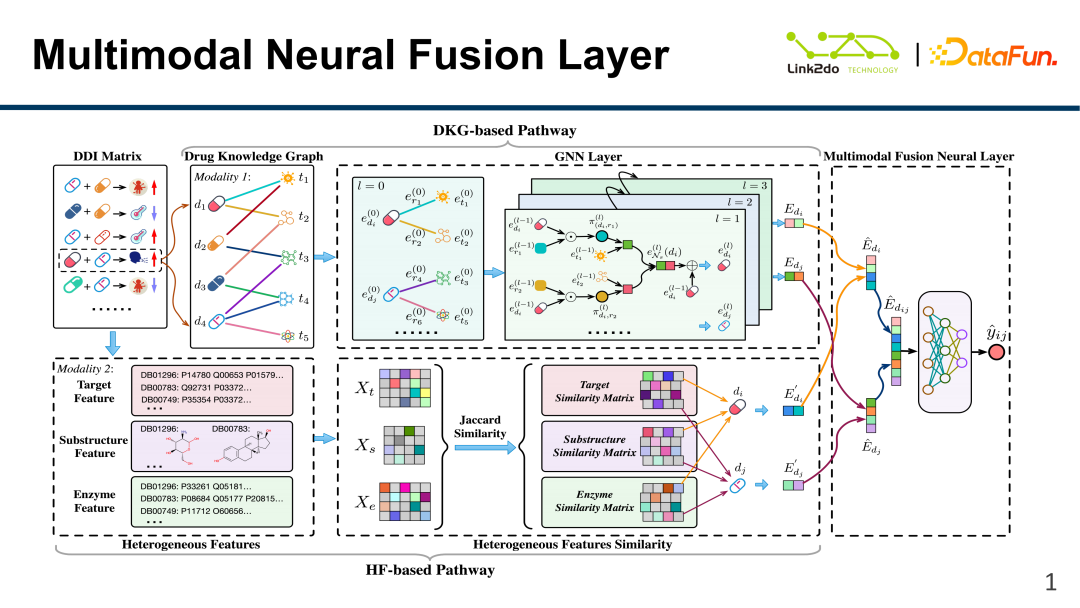
L'ensemble du cadre du modèle est tel qu'indiqué ci-dessus. La structure du cadre n'est pas compliquée, mais elle combine les informations sur les médicaments de manière relativement efficace.
3. Effet modèle MDNN
Ensuite, je partagerai l'effet modèle avec vous.
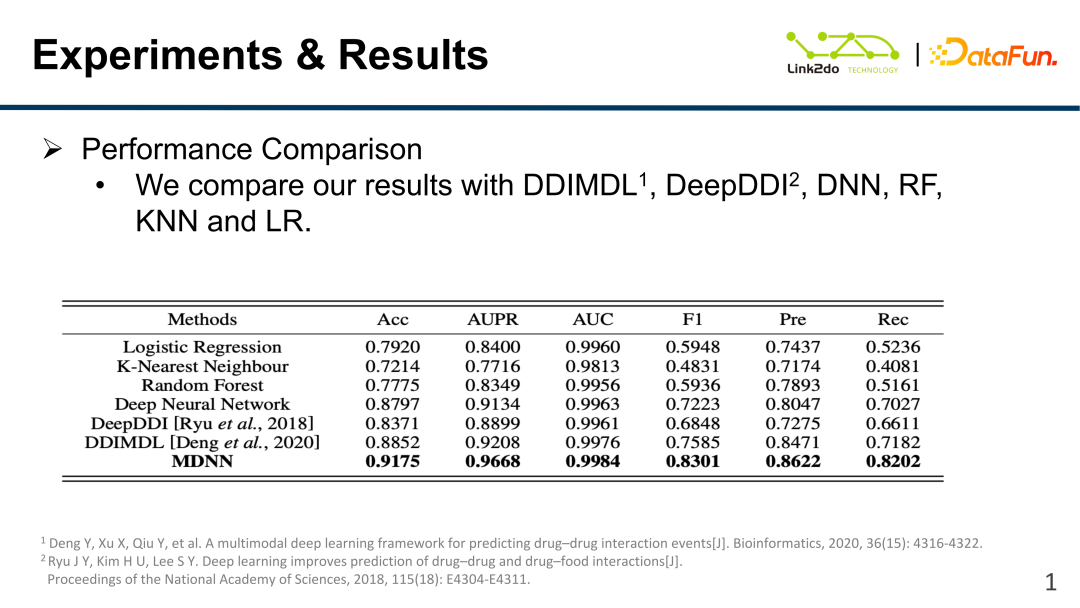
La figure ci-dessus montre les résultats de comparaison avec les algorithmes les plus couramment utilisés actuellement. L'algorithme MDNN a atteint l'état de l'Acc, de l'AUC, de la F1, de l'AUPR, de la précision, du rappel et d'autres indicateurs d'évaluation. Le résultat de l'art. (L'algorithme ci-dessus n'est pas incorporé dans l'algorithme GNN.)
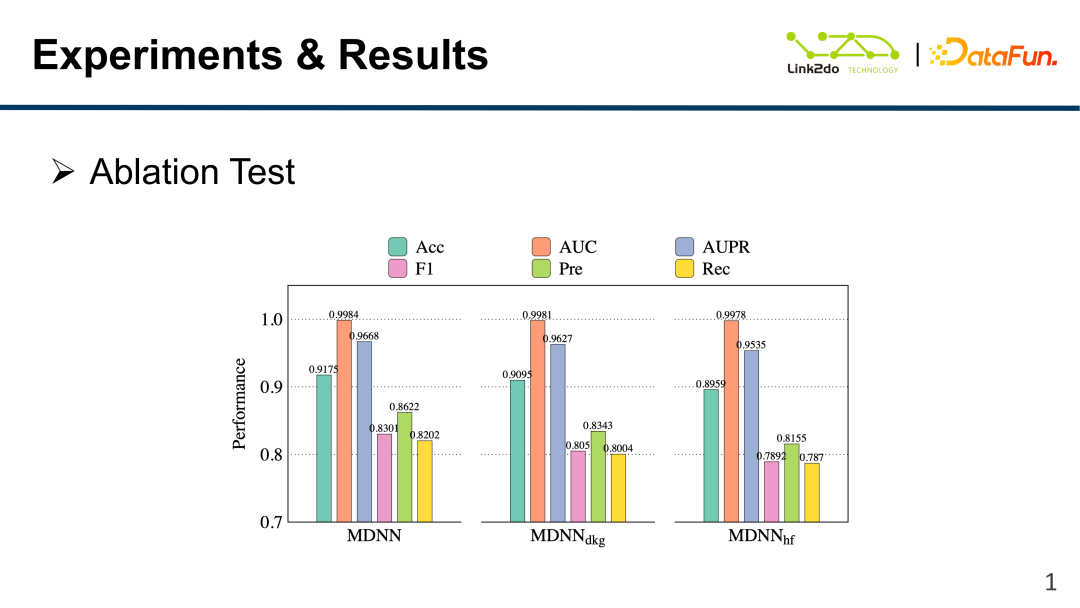
La figure ci-dessus montre la différence entre les effets du MDNN fusionné avec DKG, HK et sans fusion. Il est facile de voir que le résultat de la fusion est meilleur que l’utilisation de l’une ou l’autre méthode seule.
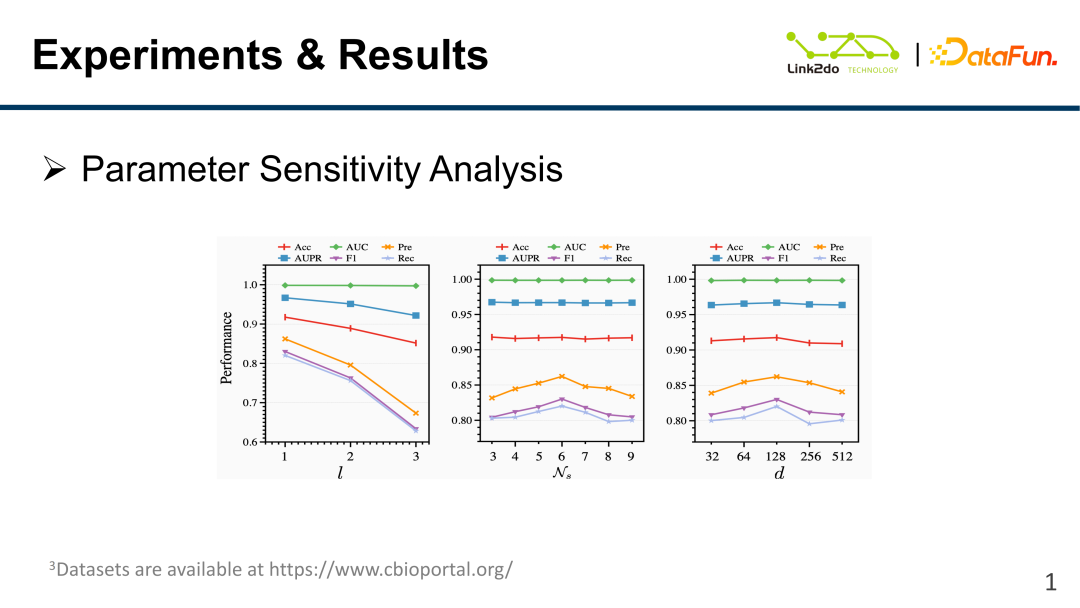
L'analyse de l'ajustement des paramètres multimodaux, c'est-à-dire la sensibilité des paramètres, est présentée dans la figure ci-dessus, qui montre le nombre de couches de réseau neuronal l et le nombre de nœuds N s Lorsque les paramètres changent, les fluctuations correspondantes de chaque indice d'évaluation se produiront.
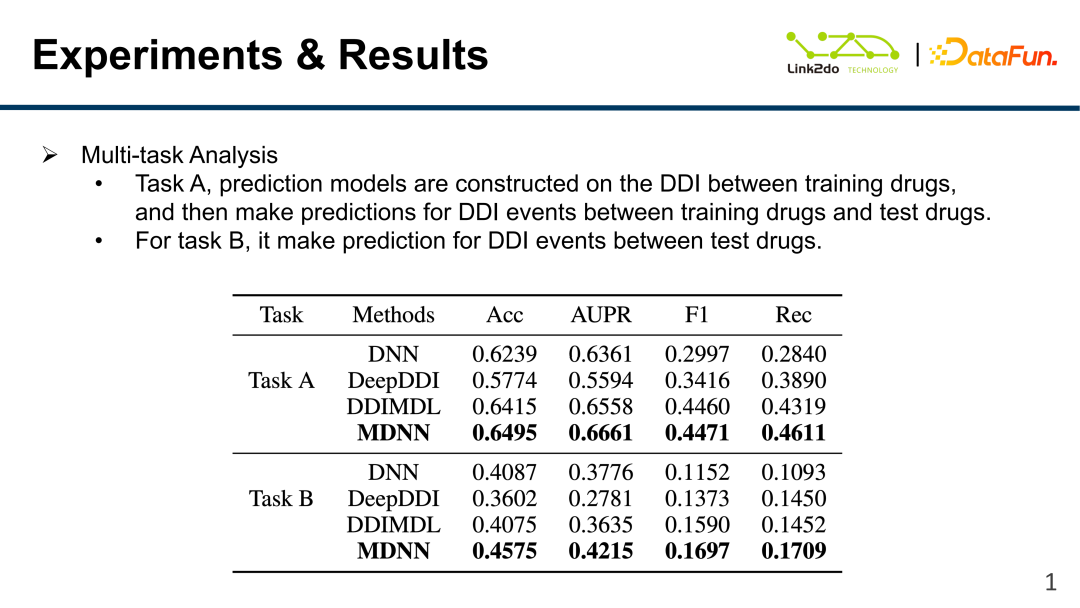
De plus, une analyse multitâche a été réalisée. Le modèle de prédiction de la tâche A a été construit à travers l'ensemble d'entraînement pour prédire le DDI entre les médicaments de l'ensemble d'entraînement et le médicament du test. le modèle de prédiction pour la tâche B a également été construit via l'ensemble d'entraînement, mais prédit le DDI entre les médicaments de l'ensemble de test. Si les médicaments de l’ensemble d’entraînement et de l’ensemble de test sont strictement séparés, l’effet de prédiction du modèle sera considérablement réduit.
Il reste encore de nombreux problèmes à résoudre dans le domaine de la recherche et du développement de médicaments : comment découvrir/cribler efficacement des médicaments au lieu de simplement étudier le DDI.
4. Résumé
Enfin, pour résumer le contenu partagé cette fois, l'algorithme MDNN en lui-même n'est pas compliqué. Le plus important est d'utiliser des données multimodales et des informations structurelles :
.

(1) Construire un graphe de connaissances sur les médicaments basé sur les effets des médicaments, construire des fonctionnalités de données multimodales basées sur les caractéristiques du médicament lui-même (cible, structure moléculaire, enzyme), et enfin intégrer ces deux fonctionnalités pour construire un Modèle MDNN.
(2) Amélioration du problème de prédiction du DDI
(3) Par rapport aux méthodes existantes, MDNN a le meilleur effet sur l'ensemble de données ;
Mais dans les applications pratiques, le modèle MDNN comporte encore de nombreux domaines qui nécessitent une optimisation et une amélioration supplémentaires, comme une meilleure méthodologie ou de meilleures données.
5. Séance de questions et réponses
Q1 : L'ensemble de données « Banque de médicaments » est-il un ensemble de données public ?
A1 : L'ensemble de données du graphique de connaissances de cette étude est public, et l'ensemble de données original « Banque de médicaments » est également un ensemble de données public. Cependant, en fonction des médicaments dans chaque domaine de recherche, les ensembles de données du graphique de connaissances construits seront également différents, et il n'existe pas de graphique de connaissances unifié et universel.
Q2 : Quelles sont les applications des méthodes de recherche partagées telles que le knowledge graph, la fusion multimodale, etc. en dehors du domaine pharmaceutique ? Comme les protéines, l’immunité, etc. ?
A2 : Outre les produits biopharmaceutiques, il existe de nombreuses applications dans le domaine du e-commerce. Par exemple, dans l'ensemble de données de la classe User-Item, l'utilisateur dispose de nombreuses informations multimodales, telles que la profession, l'âge, les étiquettes d'achat, etc. Les produits Item contiennent également de nombreuses informations et il existe de nombreuses relations entre eux. tels que les achats, l'évaluation, les favoris, les clics, etc. Comportement. Si les données du domaine sont conformes à la forme hétérogène, vous pouvez essayer d'utiliser ces méthodes d'analyse. La difficulté réside dans la construction d'un graphe de connaissances dans un domaine spécifique.
Ce qui précède est le contenu détaillé de. pour plus d'informations, suivez d'autres articles connexes sur le site Web de PHP en chinois!
Articles Liés
Voir plus- Tendances technologiques à surveiller en 2023
- Comment l'intelligence artificielle apporte un nouveau travail quotidien aux équipes des centres de données
- L'intelligence artificielle ou l'automatisation peuvent-elles résoudre le problème de la faible efficacité énergétique des bâtiments ?
- Co-fondateur d'OpenAI interviewé par Huang Renxun : les capacités de raisonnement de GPT-4 n'ont pas encore atteint les attentes
- Bing de Microsoft surpasse Google en termes de trafic de recherche grâce à la technologie OpenAI

