Maison >Périphériques technologiques >IA >Présentation d'ImageMol, le premier cadre de génération d'images moléculaires au monde basé sur l'apprentissage auto-supervisé
Présentation d'ImageMol, le premier cadre de génération d'images moléculaires au monde basé sur l'apprentissage auto-supervisé
- WBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBavant
- 2023-04-23 12:46:081717parcourir
Moléculaire est la plus petite unité qui maintient la stabilité chimique des substances. L’étude des molécules est un enjeu fondamental dans de nombreux domaines scientifiques comme la pharmacie, la science des matériaux, la biologie ou encore la chimie.
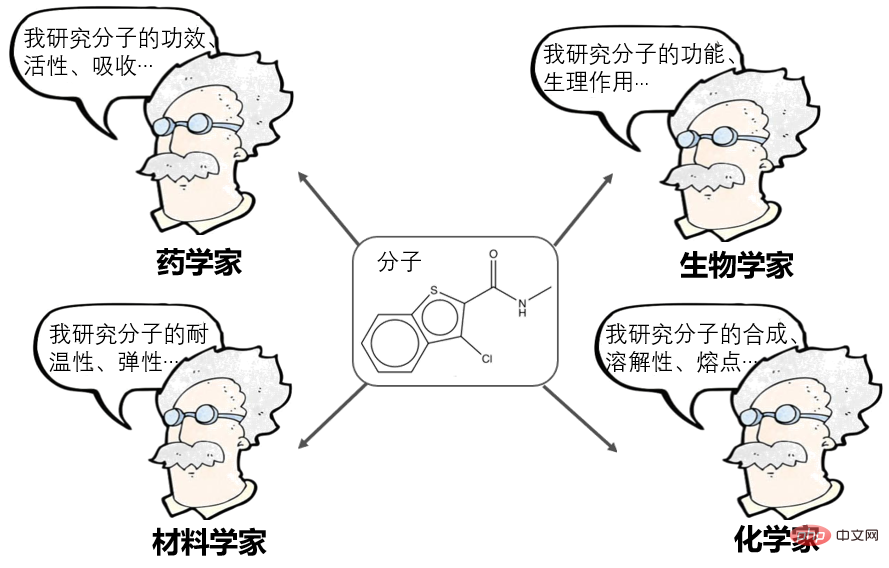
L'apprentissage de la représentation moléculaire a été une direction très populaire ces dernières années et peut actuellement être divisé en de nombreuses écoles :
- Les pharmaciens informatiques disent : les molécules peuvent être représentées comme une chaîne d'empreintes digitales, ou de descripteurs, comme AttentiveFP proposé par Shanghai Drug, sont des représentants remarquables à cet égard.
- NLPer a déclaré : Les molécules peuvent être exprimées sous forme de SMILES (séquences) puis traitées comme un langage naturel, comme le X-Mol de Baidu, qui est un représentant exceptionnel à cet égard.
- Les chercheurs en réseaux neuronaux graphiques disent : les molécules peuvent être représentées sous forme de graphique (Graph), qui est une matrice de contiguïté, puis traitées à l'aide de réseaux neuronaux graphiques, tels que GROVER de Tencent, DMPNN du MIT, MOLCLR de CMU et d'autres méthodes. représentant exceptionnel à cet égard.
Cependant, les méthodes de caractérisation actuelles présentent encore certaines limites. Par exemple, la représentation des séquences manque d'informations structurelles explicites sur les molécules, et la capacité d'expression des réseaux neuronaux graphiques existants présente encore de nombreuses limites (le professeur Shen Huawei de l'Institut de technologie informatique de l'Académie chinoise des sciences en a discuté, voir le rapport de M. Shen « The Capacité d'expression des réseaux de neurones graphiques").
Ce qui est intéressant, c'est que lorsque nous étudions les molécules en chimie au lycée, nous voyons des images de molécules. Lorsque les chimistes conçoivent des molécules, ils observent et réfléchissent également sur la base d'images moléculaires. Une idée naturelle surgit spontanément : "Pourquoi ne pas utiliser directement des images moléculaires pour représenter des molécules ?" Si vous pouvez utiliser directement des images pour représenter des molécules, alors tous les dix-huit arts martiaux en CV (vision par ordinateur) ne peuvent pas être utilisés. utilisé pour étudier des molécules ?
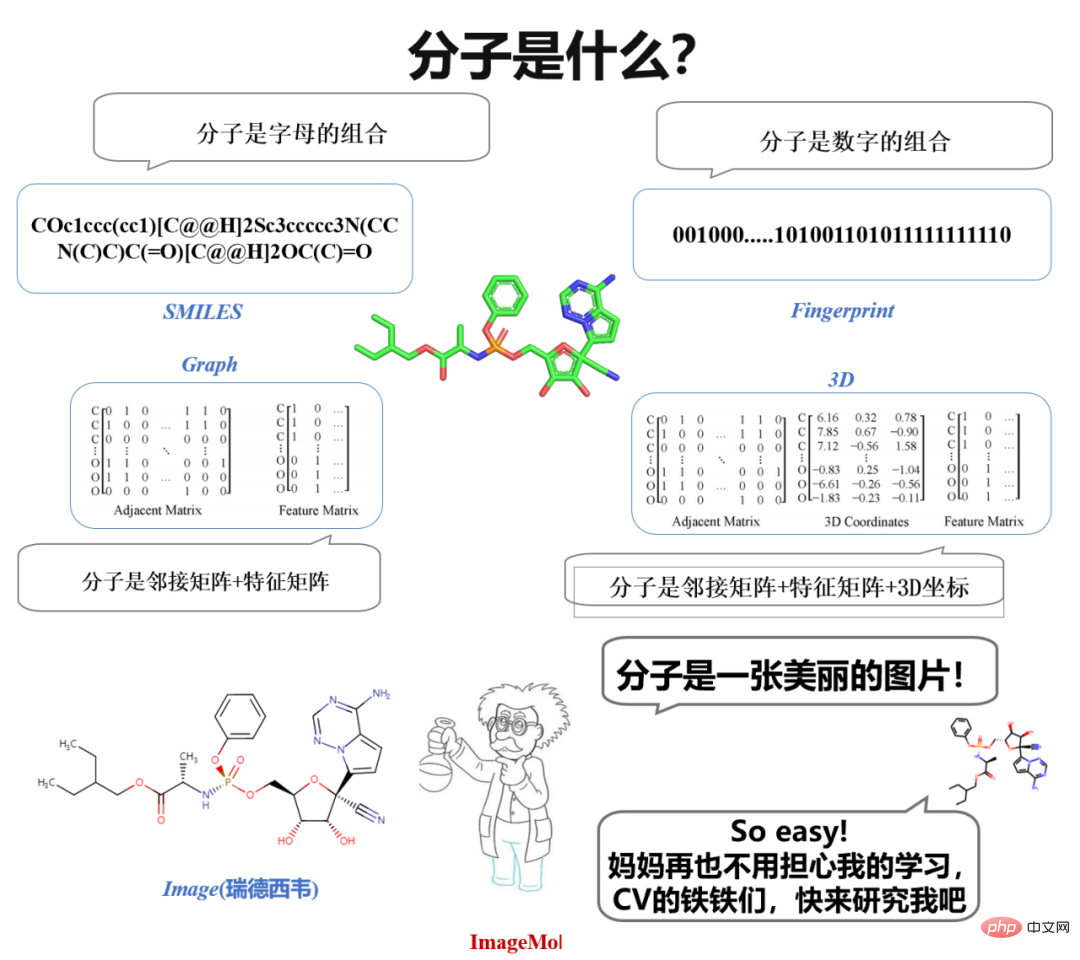
Faites-le, il y a tellement de modèles dans les CV, que diriez-vous de les utiliser pour apprendre des molécules ? Arrêtez, il y a un autre problème important : les données ! Des données particulièrement étiquetées ! Dans le domaine du CV, l’annotation des données ne semble pas difficile. Pour des problèmes classiques de CV et de PNL comme la reconnaissance d’images ou la classification d’émotions, une personne peut annoter en moyenne 800 données. Cependant, dans le domaine moléculaire, les propriétés moléculaires ne peuvent être évaluées que par des expériences humides et des expériences cliniques, de sorte que les données étiquetées sont très rares.
Sur cette base, des chercheurs de l'Université du Hunan ont proposé le premier cadre d'apprentissage non supervisé au monde pour les images moléculaires, ImageMol, qui utilise des données d'images moléculaires non étiquetées à grande échelle pour un pré-entraînement non supervisé afin de fournir des informations sur les propriétés moléculaires et les cibles des médicaments. fournit un nouveau paradigme et prouve que les images moléculaires ont un grand potentiel dans le domaine du développement de médicaments intelligents. Le résultat a été publié dans la revue internationale de premier plan « Nature Machine Intelligence » sous le titre « Prédiction précise des propriétés moléculaires et des cibles médicamenteuses à l'aide d'un cadre d'apprentissage de représentation d'image auto-supervisé ». Le succès obtenu à l'intersection de la vision par ordinateur et des domaines moléculaires démontre le grand potentiel de l'utilisation de la technologie de vision par ordinateur pour comprendre les propriétés moléculaires et les mécanismes cibles des médicaments, et offre de nouvelles opportunités de recherche dans le domaine moléculaire. L'architecture globale d'ImageMol est présentée dans la figure ci-dessous, divisée en trois parties au total :
(1) Concevoir un encodeur moléculaire ResNet18 (bleu clair), qui peut extraire des caractéristiques latentes d'environ 10 millions d'images moléculaires (a).
(2) Prenant en compte les connaissances chimiques et les informations structurelles dans les images moléculaires, cinq stratégies de pré-entraînement (MG3C, MRD, JPP, MCL, MIR) sont utilisées pour optimiser la représentation latente de l'encodeur moléculaire (b). Plus précisément :
① MG3C (Classification des clusters chimiques multi-granularité) : Le classificateur de structure (bleu foncé) est utilisé pour prédire les informations sur la structure chimique dans les images moléculaires
② MRD (Discrimination de rationalité moléculaire) : Classificateur de rationalité ( vert), qui est utilisé pour distinguer les molécules raisonnables et déraisonnables ;
③ JPP (Prédiction du puzzle) : Le classificateur Jigsaw (gris clair) est utilisé pour prédire l'arrangement raisonnable des molécules ;
④ MCL (MASK) ; -based contrastive learning basé sur MASK contrastive learning) : Le classificateur contrastif Classificateur contrastif (gris foncé) Utilisé pour maximiser la similarité entre l'image originale et l'image masque
⑤ MIR (Reconstruction d'image moléculaire) : Le générateur (jaune) ) est utilisé pour restaurer les caractéristiques latentes de l'image moléculaire, et le discriminateur est (violet) utilisé pour distinguer les images réelles des fausses images de molécules générées par le générateur.
(3) Affinez l'encodeur moléculaire prétraité dans les tâches en aval pour améliorer encore les performances du modèle (c).
Évaluation de référence
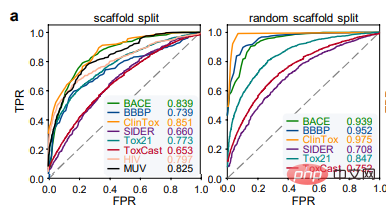
Les auteurs ont d'abord utilisé 8 ensembles de données de référence sur la découverte de médicaments pour évaluer les performances d'ImageMol, et ont utilisé les deux stratégies de fractionnement les plus populaires (répartition de l'échafaudage et répartition aléatoire de l'échafaudage) pour évaluer les performances d'ImageMol. Performances sur tous les ensembles de données de référence. Dans la tâche de classification, la courbe des caractéristiques de fonctionnement du récepteur (ROC) et la zone sous la courbe (AUC) sont utilisées pour évaluer. À partir des résultats expérimentaux, on peut voir qu'ImageMol peut obtenir des valeurs d'AUC plus élevées (Figure a).
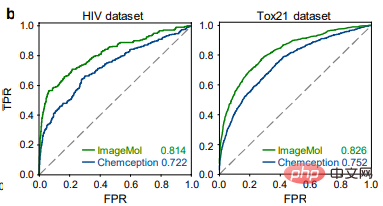
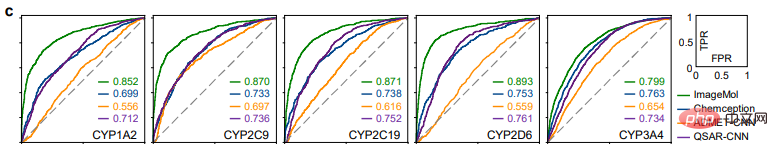
En comparant les résultats de détection du VIH et de Tox21 entre ImageMol et Chemception, un cadre de réseau neuronal convolutionnel classique pour prédire les images moléculaires (Figure b), ImageMol a une valeur d'ASC plus élevée. Cet article évalue en outre les performances d'ImageMol dans la prédiction du métabolisme des médicaments par cinq enzymes métabolisantes majeures : CYP1A2, CYP2C9, CYP2C19, CYP2D6 et CYP3A4. La figure c montre qu'ImageMol obtient de meilleurs résultats par rapport à trois modèles de représentation basés sur des images moléculaires de pointe (Chemception46, ADMET-CNN12 et QSAR-CNN47) dans la prédiction des inhibiteurs par rapport aux non-inhibiteurs de cinq enzymes majeures du métabolisme des médicaments. . a atteint des valeurs d'AUC plus élevées (allant de 0,799 à 0,893).
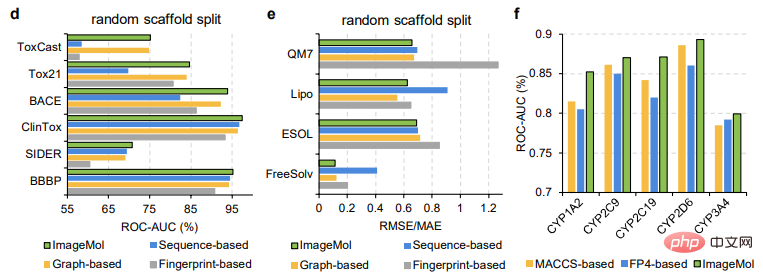
Cet article compare en outre les performances d'ImageMol avec trois modèles de représentation moléculaire de pointe, comme le montrent les figures d, e. ImageMol a de meilleures performances par rapport aux modèles basés sur les empreintes digitales (tels que AttentiveFP), aux modèles basés sur des séquences (tels que TF_Robust) et aux modèles basés sur des graphiques (tels que N-GRAM, GROVER et MPG) qui utilisent un partitionnement squelette aléatoire. De plus, ImageMol a atteint des valeurs d'ASC plus élevées sur les CYP1A2, CYP2C9, CYP2C19, CYP2D6 et CYP3A4 par rapport aux méthodes traditionnelles basées sur MACCS et aux méthodes basées sur FP4 (Figure f).
ImageMol Comparé aux modèles basés sur des séquences (y compris RNN_LR, TRFM_LR, RNN_MLP, TRFM_MLP, RNN_RF, TRFM_RF et CHEM-BERT) et aux modèles basés sur des graphiques (y compris MolCLRGIN, MolCLRGCN et GROVER), comme le montre la figure g, ImageMol De meilleures performances de l'ASC ont été obtenues pour les CYP1A2, CYP2C9, CYP2C19, CYP2D6 et CYP3A4.
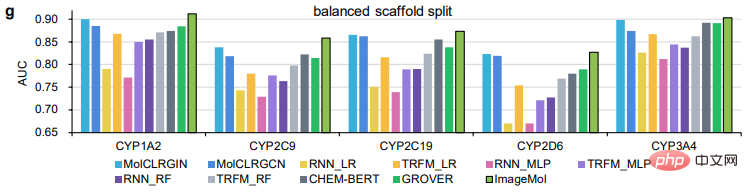
Dans la comparaison ci-dessus entre ImageMol et d'autres modèles avancés, nous pouvons voir la supériorité d'ImageMol.
Depuis l’apparition du COVID-19, nous devons élaborer de toute urgence des stratégies de traitement efficaces contre le COVID-19. Par conséquent, les auteurs ont évalué ImageMol en conséquence sous cet aspect.
Prédiction de 13 cibles du SRAS-CoV-2
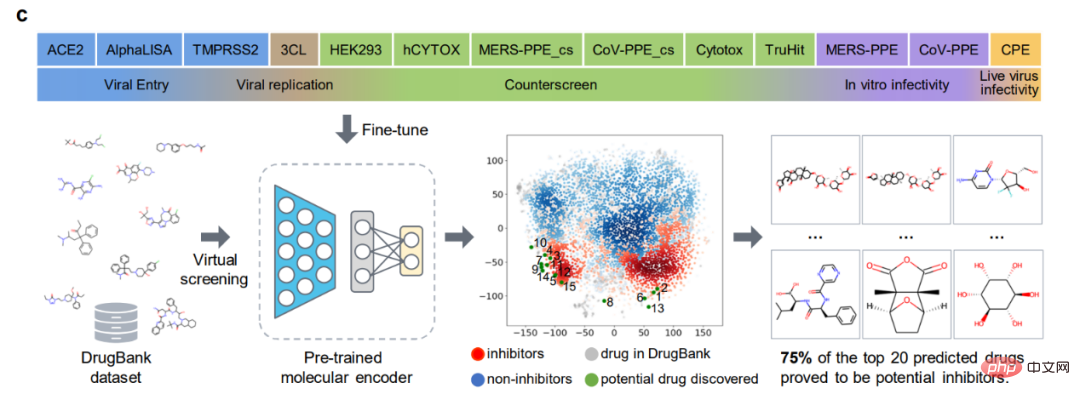
ImageMol a mené des expériences de prédiction sur le SRAS-CoV-2 qui est actuellement préoccupant dans 13 ensembles de données de tests biologiques sur le SRAS-CoV-2, ImageMol a atteint des valeurs d'ASC élevées. de 72,6% à 83,7%. Le panneau a révèle la signature potentielle identifiée par ImageMol, qui se regroupe bien sur 13 cibles ou points finaux anti-SARS-CoV-2 actifs et inactifs, avec des valeurs d'ASC supérieures aux autres. Le GNN du modèle Jure est plus de 12 % plus élevé, reflétant la grande précision et la forte généralisation du modèle.
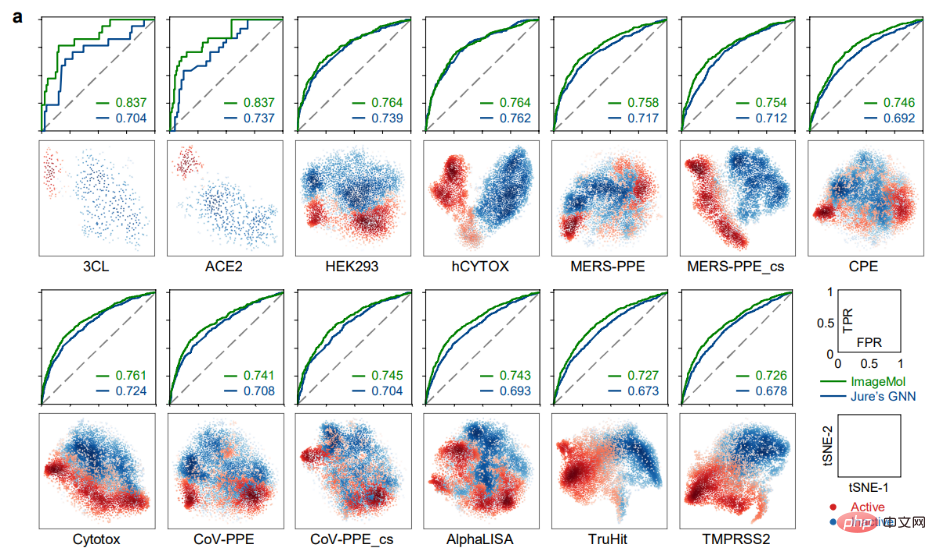
Identification des inhibiteurs anti-SARS-CoV-2
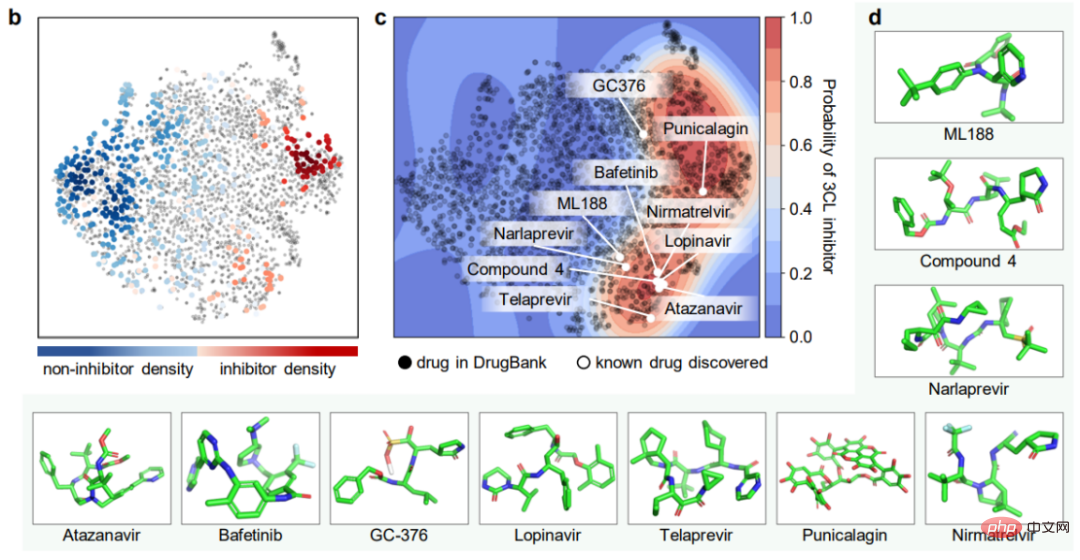
L'expérience la plus directe liée à la recherche de molécules médicamenteuses est ici, utilisez ImageMol pour identifier directement les molécules inhibitrices ! Grâce à la représentation par image moléculaire des inhibiteurs et des non-inhibiteurs de la protéase 3CL (qui s'est avérée être une cible de développement thérapeutique prometteuse pour le traitement du COVID-19) dans le cadre d'ImageMol, cette étude a révélé que les inhibiteurs et les non-inhibiteurs de la 3CL ont différences significatives dans les puits t séparés dans le tracé SNE, comme le montre la figure b ci-dessous.
De plus, ImageMol a identifié 10 des 16 inhibiteurs de protéase 3CL connus et a visualisé ces 10 médicaments dans l'espace d'intégration de la figure (taux de réussite de 62,5 %), indiquant qu'ils sont efficaces contre le SRAS-CoV-2. Forte généralisation capacité dans la découverte de médicaments. Lors de l’utilisation du test HEY293 pour prédire les médicaments réutilisés anti-SARS-CoV-2, ImageMol a prédit avec succès 42 médicaments sur 70 (taux de réussite de 60 %), ce qui indique qu’ImageMol est également efficace pour déduire des médicaments candidats potentiels dans le test HEY293. fort potentiel de promotion. La figure c ci-dessous montre la découverte par ImageMol de médicaments qui sont des inhibiteurs potentiels de la 3CL sur l'ensemble de données DrugBank. Le panneau d montre la structure moléculaire de l’inhibiteur 3CL découvert par ImageMol.
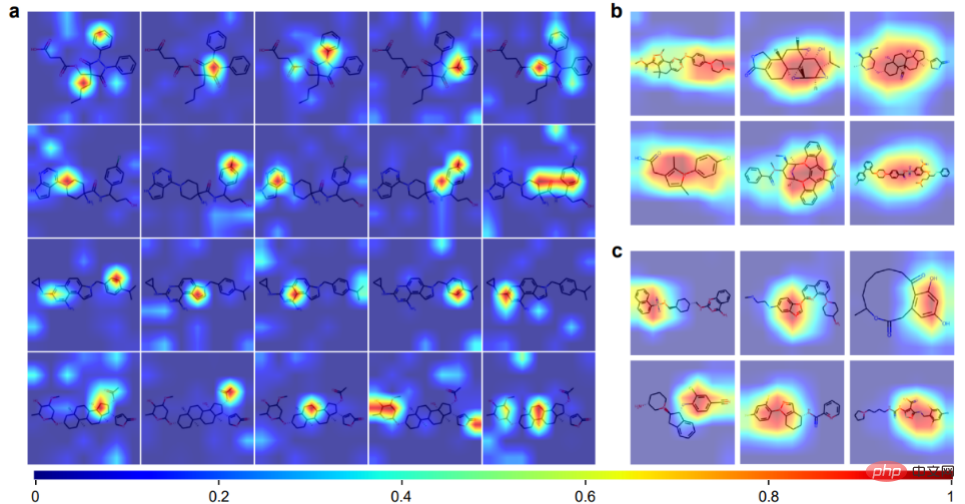
Attention Visualisation
ImageMol peut obtenir une connaissance préalable des informations chimiques à partir de représentations d'images moléculaires, notamment les liaisons = O, les liaisons -OH, les liaisons -NH3 et les cycles benzéniques. Les panneaux b et c montrent 12 exemples de molécules visualisées par Grad-CAM d'ImageMol. Cela signifie qu'ImageMol capte simultanément avec précision l'attention sur les informations structurelles globales (b) et locales (c). Ces résultats permettent aux chercheurs de comprendre visuellement comment la structure moléculaire affecte les propriétés et les cibles.
Ce qui précède est le contenu détaillé de. pour plus d'informations, suivez d'autres articles connexes sur le site Web de PHP en chinois!
Articles Liés
Voir plus- Tendances technologiques à surveiller en 2023
- Comment l'intelligence artificielle apporte un nouveau travail quotidien aux équipes des centres de données
- L'intelligence artificielle ou l'automatisation peuvent-elles résoudre le problème de la faible efficacité énergétique des bâtiments ?
- Co-fondateur d'OpenAI interviewé par Huang Renxun : les capacités de raisonnement de GPT-4 n'ont pas encore atteint les attentes
- Bing de Microsoft surpasse Google en termes de trafic de recherche grâce à la technologie OpenAI

