Maison >Périphériques technologiques >IA >Meta crée le premier panorama « Protein Universe » ! En utilisant un modèle de langage de 15 milliards de paramètres, plus de 600 millions de structures protéiques ont été prédites
Meta crée le premier panorama « Protein Universe » ! En utilisant un modèle de langage de 15 milliards de paramètres, plus de 600 millions de structures protéiques ont été prédites
- PHPzavant
- 2023-04-12 18:25:101460parcourir
Meta fait un nouveau pas en avant dans l'exploration de la structure des protéines !
Cette fois, ils ciblent un domaine cible plus large : la métagénomique. La « matière noire » de l'univers protéique Des séquences nucléotidiques entières isolées et analysées sont souvent utilisées pour étudier des communautés microbiennes spécifiques, telles que les protéines qui vivent sur la peau humaine, dans le sol ou dans des échantillons d'eau.
Au cours des dernières décennies, la métagénomique a été un domaine très actif à mesure que nous en apprenons davantage sur tous les micro-organismes qui vivent dans, sur et chez les humains et dans l'environnement. 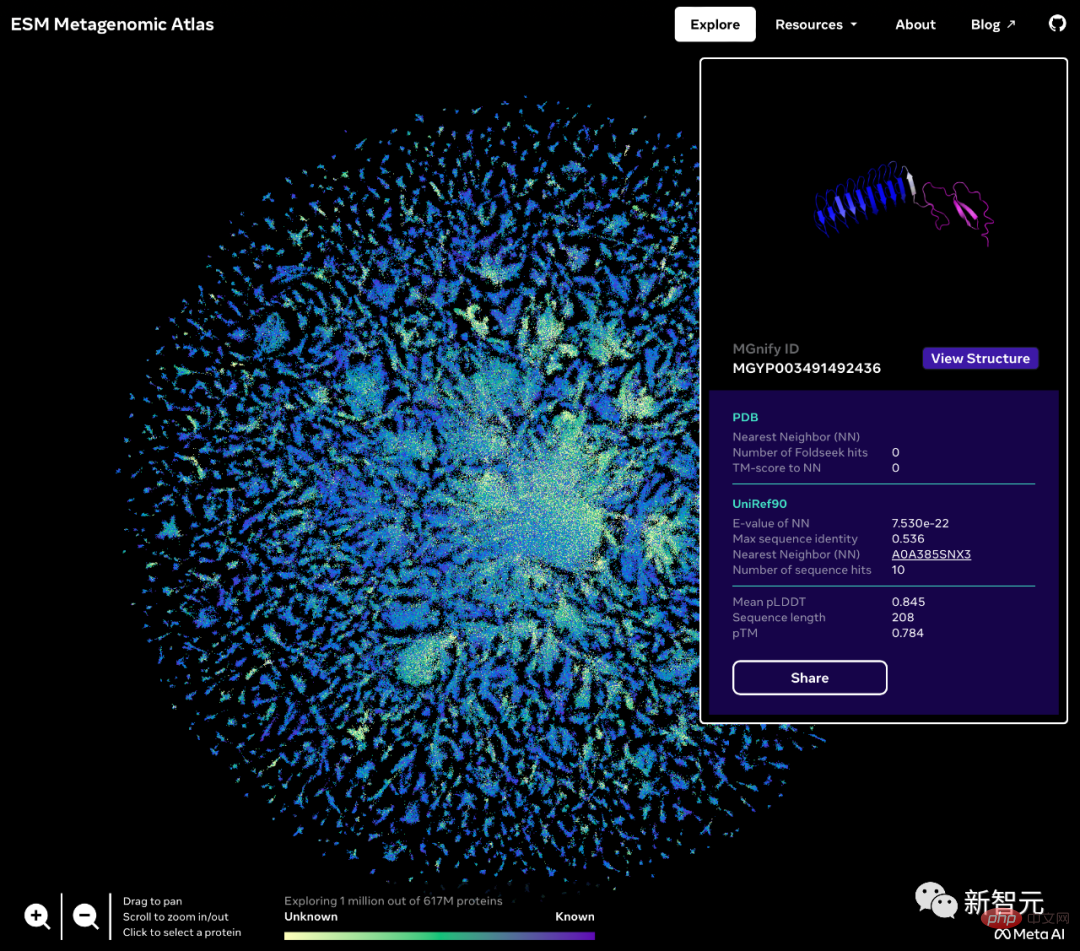
Adresse papier : https://www.biorxiv.org/content/10.1101/2022.07.20.500902v2
Meta a déclaré que le décodage de la structure métagénomique aidera à résoudre le mystère de longue date de l'histoire de l'évolution humaine, Aide les humains guérissent les maladies et purifient l’environnement plus efficacement.
Prédiction de la structure des protéines, 60 fois plus rapide !

Ces dernières années, les progrès du séquençage génétique ont permis de cataloguer des milliards de séquences protéiques métagénomiques.
Cependant, bien que l’existence de ces séquences protéiques soit connue, mieux comprendre leurs propriétés biologiques constitue un défi de taille.
 Afin d'obtenir les structures de séquence de ces milliards de protéines, une percée dans la vitesse de prédiction est cruciale.
Afin d'obtenir les structures de séquence de ces milliards de protéines, une percée dans la vitesse de prédiction est cruciale.
Ce processus, même avec les outils les plus avancés et les ressources informatiques d'un grand établissement de recherche, peut prendre plusieurs années.
Meta a donc formé un grand modèle de langage pour apprendre des modèles évolutifs et générer des prédictions de structure précises directement à partir des séquences protéiques de bout en bout, tout en maintenant la précision et en prédisant 60 fois plus rapidement que les méthodes de pointe actuelles.
En fait, grâce à cette nouvelle capacité de prédiction de structure, Meta a prédit les séquences de plus de 600 millions de protéines métagénomiques sur la carte en seulement deux semaines à l'aide d'un cluster d'environ 2 000 GPU. 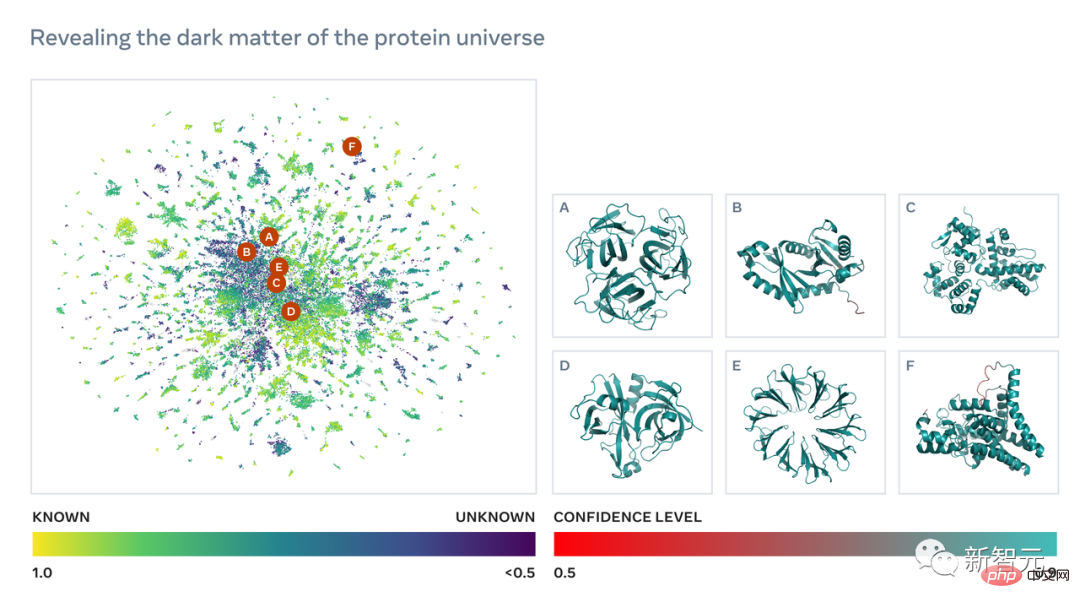
La carte métagénomique publiée par Meta s'appelle ESM Atlas, qui couvre presque les prédictions de l'ensemble de la base de données publique de séquences métagénomiques MGnify90.
Meta a déclaré qu'ESM Atlas est la plus grande base de données de structure prédite à haute résolution à ce jour, 3 fois plus grande que les bases de données de structure de protéines existantes, et la première base de données à couvrir de manière exhaustive et à grande échelle les protéines métagénomiques.
Ces structures protéiques offrent une vue sans précédent sur l'étendue et la diversité de la nature et ont le potentiel d'accélérer la découverte d'applications pratiques des protéines dans des domaines tels que la médecine, la chimie verte, les applications environnementales et les énergies renouvelables.
Le nouveau modèle de langage utilisé pour prédire la structure des protéines comporte 15 milliards de paramètres, ce qui en fait le plus grand « modèle de langage protéique » à ce jour. 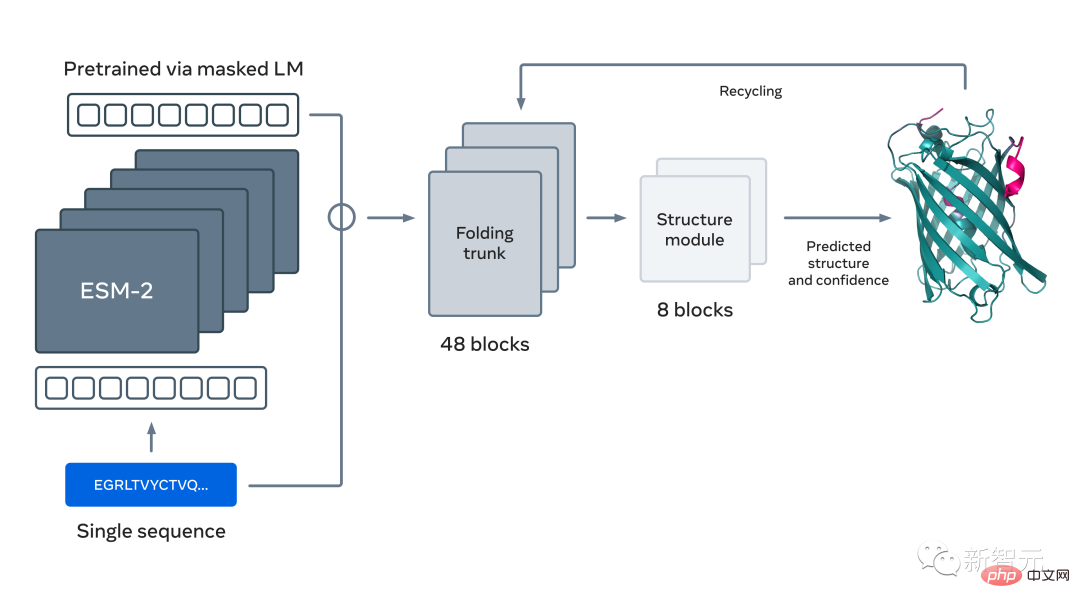
Il peut être difficile de comprendre la comparaison de vitesse entre les trois en parlant d'ordre de grandeur. Il suffit de regarder l'image ci-dessous pour comprendre.
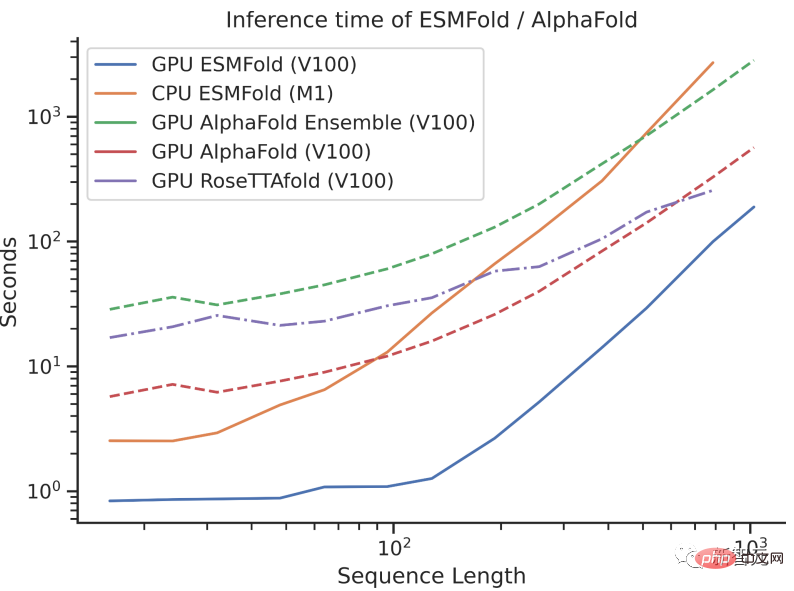
La sortie de la base de données ESM Atlas a permis au modèle de langage étendu à 15 milliards de paramètres d'être utilisé le plus largement possible.
Cela permet aux scientifiques de rechercher et d'analyser des structures jusqu'alors non caractérisées à l'échelle de centaines de millions de protéines et de découvrir de nouvelles protéines utiles en médecine et dans d'autres applications.
Le modèle linguistique est vraiment "universel"
Tout comme le texte, les protéines peuvent également être écrites sous forme de séquences de caractères.
Parmi eux, chaque "caractère" qui constitue une protéine correspond à l'un des 20 éléments chimiques standards - l'acide aminé. Et chaque acide aminé possède des propriétés différentes.
Mais c'est un grand défi de comprendre ce "langage biologique".
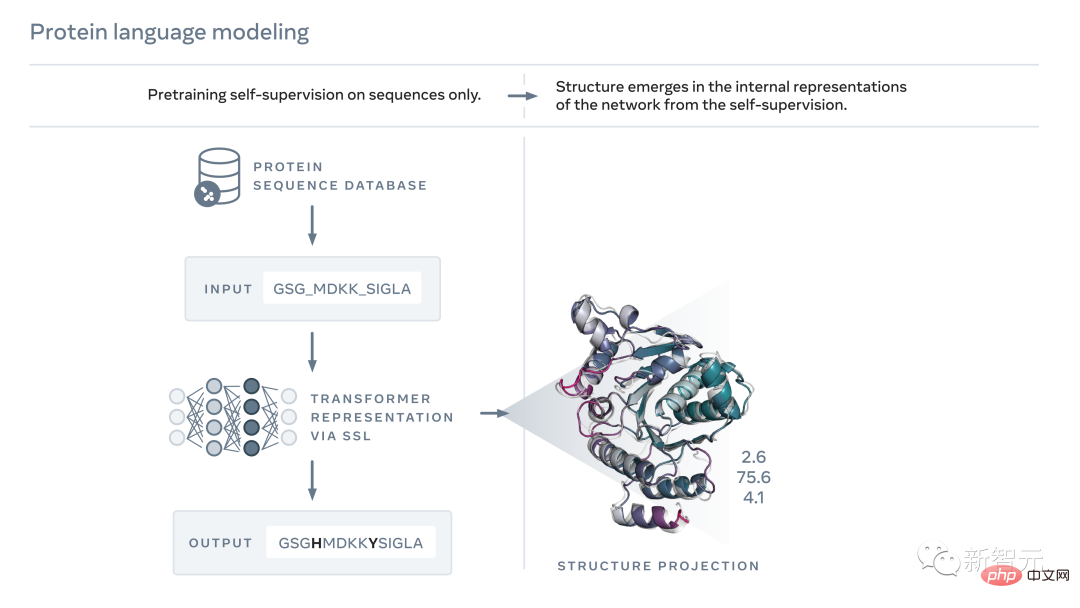
Bien que, comme je viens de le dire, une séquence protéique et un morceau de texte puissent être écrits sous forme de caractères, il existe des différences profondes et fondamentales entre eux.
D'une part, le nombre de combinaisons différentes de ces "caractères" est un nombre astronomique. Par exemple, pour une protéine composée de 200 acides aminés, il existe 20^200 séquences possibles, soit plus que le nombre d'atomes de l'univers actuellement explorable.
D'autre part, chaque séquence d'acides aminés se plie en une forme tridimensionnelle selon les lois de la physique. De plus, toutes les séquences ne se replient pas en structures cohérentes ; beaucoup se replient en formes désordonnées, mais c'est cette forme insaisissable qui détermine la fonction de la protéine.
Par exemple, si un certain acide aminé apparaît à une position, cet acide aminé s'associe généralement à un certain acide aminé à une autre position. Ensuite, ils sont susceptibles d’interagir dans la structure pliée ultérieure.
L'intelligence artificielle peut apprendre et lire ces modèles en observant les séquences protéiques, puis en déduire la structure réelle de la protéine.
En 2019, Meta a présenté la preuve que les modèles de langage apprennent les propriétés des protéines, telles que leur structure et leur fonction.

Adresse papier : https://www.pnas.org/doi/10.1073/pnas.2016239118
Un modèle formé à l'aide d'un apprentissage auto-supervisé tel que le masquage peut combler correctement les lacunes d'un paragraphe de texte. Par exemple, « Voulez-vous __, nous sommes ________ ».
Avec cette méthode, Meta entraîne un modèle de langage basé sur des millions de séquences protéiques naturelles, comblant ainsi les lacunes des séquences protéiques, telles que "GL_KKE_AHY_G".
Des expériences montrent que ce modèle peut être entraîné pour découvrir des informations sur la structure et la fonction des protéines.
En 2020, Meta a publié ESH1b, qui était à l'époque le modèle de langage protéique le plus avancé. Il a été utilisé dans diverses applications, notamment pour aider les scientifiques à prédire l'évolution du nouveau coronavirus et à découvrir les causes des maladies génétiques.

Adresse papier : https://www.biorxiv.org/content/10.1101/2022.08.25.505311v1
Maintenant, Meta a élargi l'échelle de cette approche, créant le modèle de langage protéique de nouvelle génération ESM-2, Il s'agit d'un grand modèle avec 15 milliards de paramètres.
À mesure que le modèle passe de 8 millions de paramètres à 15 millions de paramètres, les informations émergeant de la représentation interne permettent de prédire la structure tridimensionnelle à une résolution atomique.
Comprenez le « langage protéique » et rendez la vie plus transparente
Depuis des milliards d'années, l'évolution des êtres vivants a formé un langage protéique, qui peut former des molécules complexes et dynamiques grâce à de simples blocs de construction. Apprendre à lire le langage des protéines est une étape importante dans notre compréhension du monde naturel.
L'IA peut nous fournir de nouveaux outils pour comprendre le monde naturel, tout comme un microscope, nous permettant d'observer le monde à une échelle presque infinitésimale et ouvrant une nouvelle compréhension de la vie. L’IA peut nous aider à comprendre la vaste diversité de la nature et à voir la biologie sous un nouveau jour.
Actuellement, la plupart des recherches sur l’IA visent à permettre aux ordinateurs de comprendre le monde d’une manière similaire à celle des humains. Le langage des protéines est incompréhensible pour les humains, même pour les outils informatiques les plus puissants.
Ainsi, l'importance de ce travail de Meta est de révéler les énormes avantages de l'IA dans tous les domaines, à savoir : les grands modèles de langage qui ont fait des progrès dans la traduction automatique, la compréhension du langage naturel, la reconnaissance vocale et la génération d'images sont également capables d'apprendre Informations approfondies pertinentes sur la biologie.
Cette fois, Meta rend ce travail public, partage des données et des résultats et s'appuie sur les idées des autres. Nous espérons que la publication de cet atlas structurel à grande échelle et de ce modèle de repliement rapide des protéines pourra promouvoir de nouveaux progrès scientifiques et nous permettre de mieux comprendre. le monde qui vous entoure.
Références :
https://ai.facebook.com/blog/protein-folding-esmfold-metagenomics/?utm_source=twitter&utm_medium=organic_social&utm_campaign=blog
Ce qui précède est le contenu détaillé de. pour plus d'informations, suivez d'autres articles connexes sur le site Web de PHP en chinois!
Articles Liés
Voir plus- Tendances technologiques à surveiller en 2023
- Comment l'intelligence artificielle apporte un nouveau travail quotidien aux équipes des centres de données
- L'intelligence artificielle ou l'automatisation peuvent-elles résoudre le problème de la faible efficacité énergétique des bâtiments ?
- Co-fondateur d'OpenAI interviewé par Huang Renxun : les capacités de raisonnement de GPT-4 n'ont pas encore atteint les attentes
- Bing de Microsoft surpasse Google en termes de trafic de recherche grâce à la technologie OpenAI

