Maison >Périphériques technologiques >IA >Stanford et Microsoft se sont associés pour utiliser des modèles de diffusion pour générer des structures protéiques, ce qui est open source
Stanford et Microsoft se sont associés pour utiliser des modèles de diffusion pour générer des structures protéiques, ce qui est open source
- 王林avant
- 2023-04-11 11:58:021773parcourir
Les protéines sont vitales à la vie et jouent un rôle dans presque tous les processus biologiques. D’une part, ils peuvent transmettre des signaux entre neurones, identifier des envahisseurs microscopiques et activer des réponses immunitaires. D’autre part, les protéines ont été largement étudiées comme médiateurs thérapeutiques dans le cadre du traitement de maladies. Ainsi, en générant de nouvelles structures protéiques physiquement pliables, la porte est ouverte à de nouvelles façons d’exploiter les voies cellulaires pour traiter les maladies.
Dans cet article, des chercheurs de l'Université de Stanford, de Microsoft Research et d'autres institutions, inspirés par le processus de repliement des protéines in vivo, ont introduit un modèle de diffusion de repliement (folding diffusion, FoldingDiff), qui reflète les processus naturels de repliement des protéines pour concevoir des protéines. structures de base.

- Adresse papier : https://arxiv.org/pdf/2209.15611.pdf
- Adresse code : https://github.com/microsoft/foldingd si et si
Plus précisément, ils décrivent la structure du squelette protéique comme une série continue d'angles pour capturer les orientations relatives des résidus d'acides aminés constitutifs, et le déplacement inhérent et l'invariance en rotation de cette représentation sont extrêmes. atténue le besoin de réseaux équivariants complexes.
Cette étude a formé un modèle probabiliste de diffusion débruité basé sur le squelette du transformateur et a démontré que notre modèle peut générer de manière inconditionnelle des structures protéiques très réalistes avec une complexité et des modèles structurels similaires aux protéines natives.
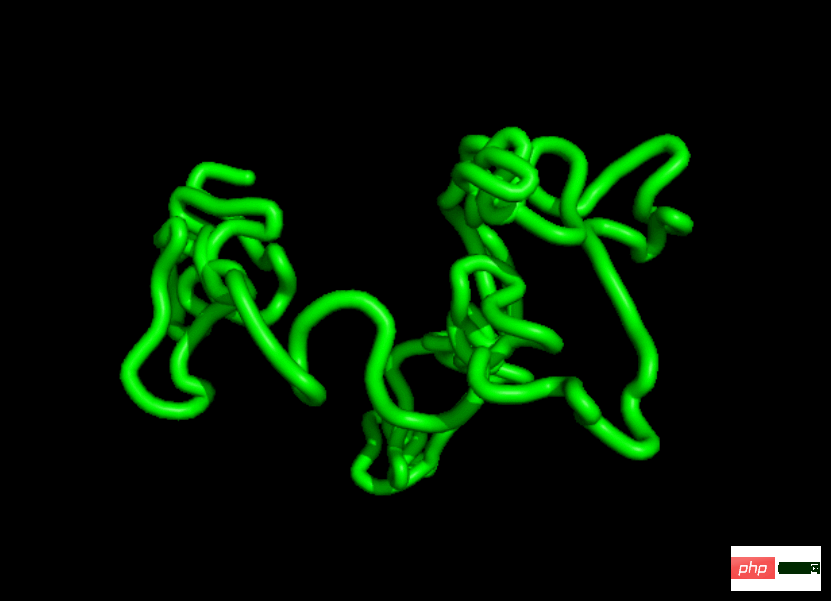
Certains internautes ont dit : Je me demande si ce modèle apportera une certaine concurrence à AlphaFold.
Méthodes et résultats
Nous pouvons comprendre les protéines comme des chaînes de résidus d'acides aminés de longueur variable. Il existe 20 acides aminés typiques, partageant le même squelette N-C_α-C à trois atomes, mais avec des côtés différents. les chaînes sont attachées à l’atome C_α (généralement noté R, voir Figure 1).
Ces résidus s'assemblent pour former des chaînes polymères qui se replient en structures 3D dont la forme détermine en grande partie la fonction de la protéine. Ces structures repliées peuvent être décrites à l'aide de quatre niveaux :
- structure primaire, qui capture simplement la séquence linéaire des acides aminés ;
- structure secondaire, qui décrit l'arrangement local des acides aminés ; , Décrit la disposition spatiale complète de tous les résidus ;
- structure quaternaire, qui décrit comment plusieurs chaînes d'acides aminés différentes se réunissent pour former un complexe plus grand.
- Cette étude propose un cadre simplifié de squelette protéique qui suit le processus biologique de repliement des protéines tout en éliminant le besoin de réseaux équivariants complexes. Plutôt que de considérer un squelette protéique, long de N acides aminés, comme une coordonnée tridimensionnelle, ils l’ont vu comme une séquence de six angles internes consécutifs. Autrement dit, étant donné la position du résidu actuel, le vecteur des six angles intérieurs décrit les positions relatives de tous les atomes du squelette dans le résidu suivant. Ces angles intérieurs peuvent être facilement calculés à l’aide de fonctions trigonométriques, en ajoutant de manière itérative des atomes au squelette protéique, puis en les reconvertissant en coordonnées cartésiennes 3D.
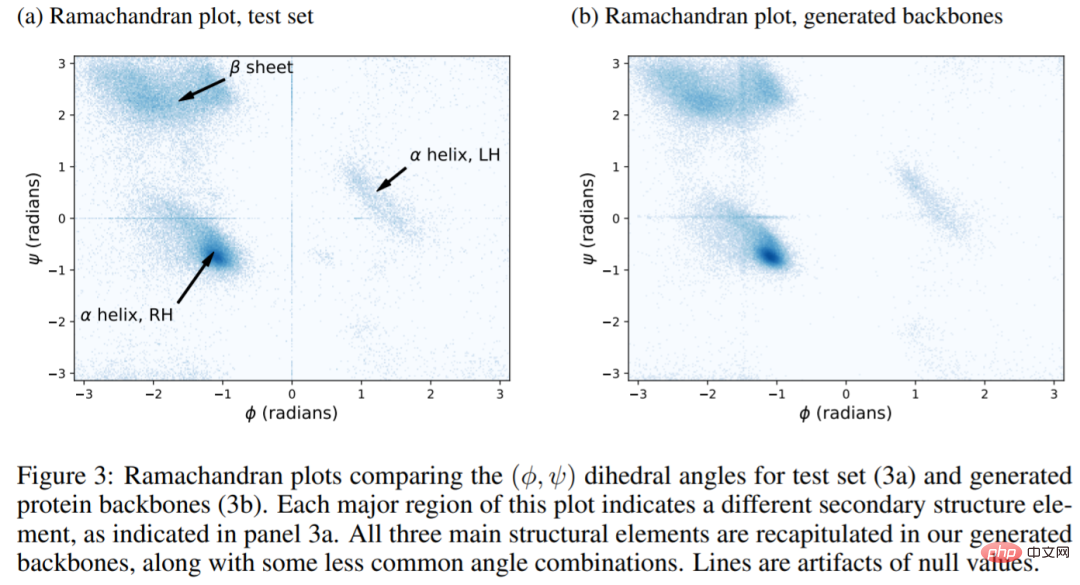
L'image ci-dessous montre les résultats d'une expérience. Le diagramme de Ramachandran de la structure naturelle (figure a) contient trois régions correspondant à l'hélice α LH, à l'hélice α RH et au feuillet β. Les trois régions sont entièrement reproduites dans la structure générée ici (Fig. 3b). En d’autres termes, FoldingDiff est capable de générer des éléments de structure secondaires au sein du squelette protéique. De plus, des expériences montrent que le modèle FoldingDiff apprend correctement que les hélices α RH sont plus courantes que les hélices α LH. Les travaux antérieurs utilisant des réseaux équivariants n'ont pas permis de distinguer ces deux types de spirales.
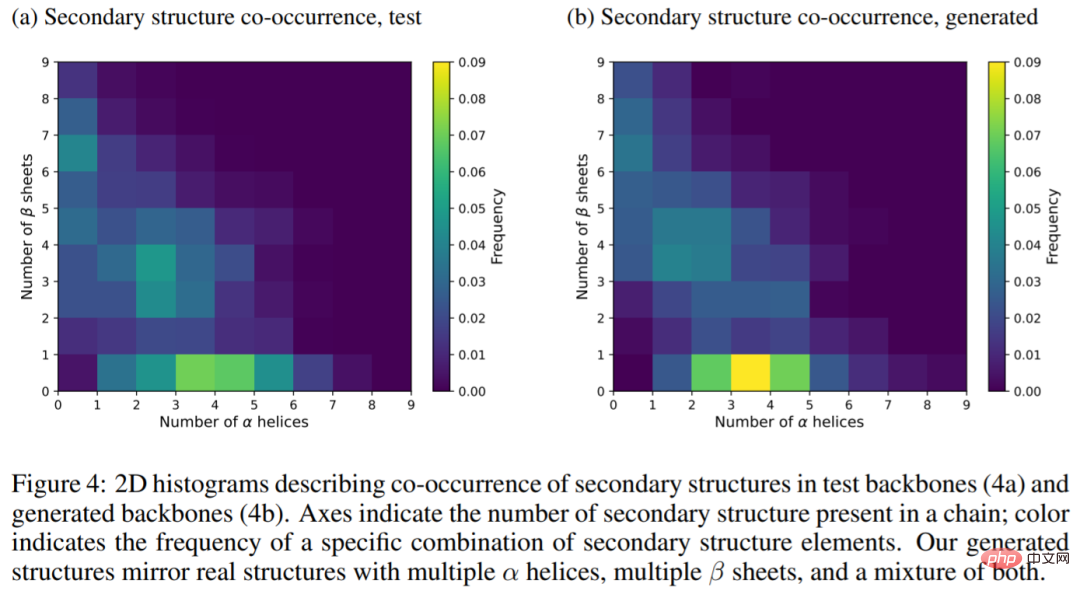
L'image ci-dessous montre l'histogramme bidimensionnel de la structure secondaire dans la chaîne principale de test (4a) et la chaîne principale générée (4b). Les résultats montrent que la structure générée reflète la vraie structure. de la protéine, avec plusieurs hélices alpha, plusieurs feuilles bêta et un mélange des deux.
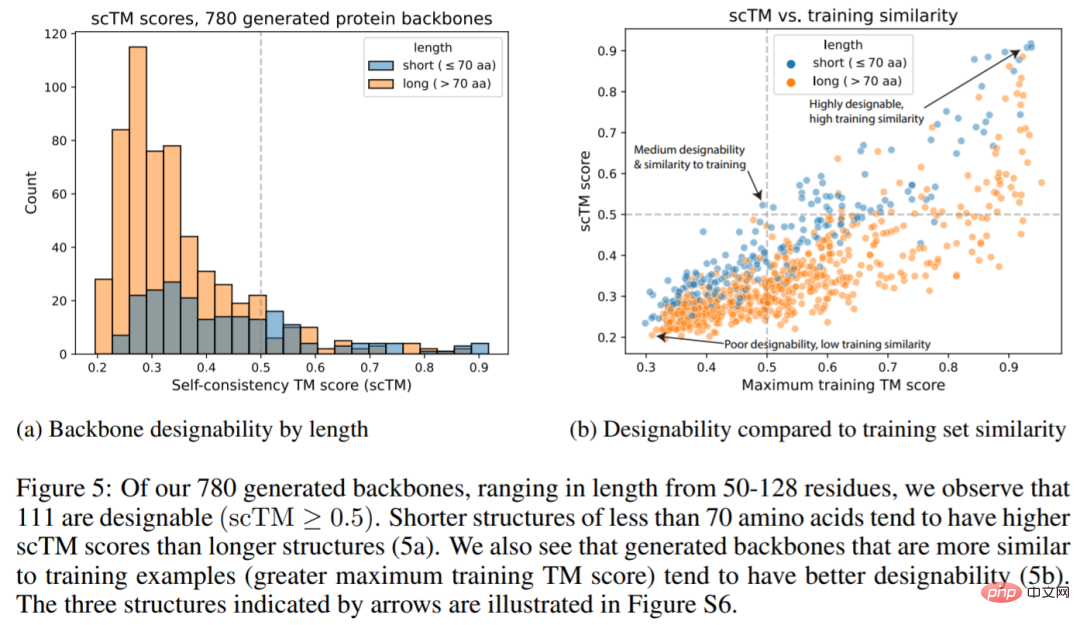
La figure ci-dessous montre que 111 des 780 structures générées (14,2 %) sont concevables avec un score scTM ≥0,5 (Fig. 5a), ce qui est supérieur à celui rapporté par Trippe et al. 11,8%. Nous voyons également que les chaînes principales générées sont plus similaires aux exemples de formation et ont tendance à avoir une meilleure conception (5b).
Pour plus d'informations, veuillez lire l'article original.
Ce qui précède est le contenu détaillé de. pour plus d'informations, suivez d'autres articles connexes sur le site Web de PHP en chinois!
Articles Liés
Voir plus- Tendances technologiques à surveiller en 2023
- Comment l'intelligence artificielle apporte un nouveau travail quotidien aux équipes des centres de données
- L'intelligence artificielle ou l'automatisation peuvent-elles résoudre le problème de la faible efficacité énergétique des bâtiments ?
- Co-fondateur d'OpenAI interviewé par Huang Renxun : les capacités de raisonnement de GPT-4 n'ont pas encore atteint les attentes
- Bing de Microsoft surpasse Google en termes de trafic de recherche grâce à la technologie OpenAI

