Maison >Périphériques technologiques >IA >Ouvrir-source ! Les Chinois de Hong Kong, le MIT et Fudan proposent le premier modèle de base de l'ARN
Ouvrir-source ! Les Chinois de Hong Kong, le MIT et Fudan proposent le premier modèle de base de l'ARN
- PHPzavant
- 2023-04-09 15:11:031258parcourir
Contrairement au domaine des protéines, la recherche dans le domaine de l'ARN manque souvent de données d'annotation suffisantes. Par exemple, les données 3D ne contiennent que plus de 1 000 ARN. Cela limite considérablement le développement de méthodes d’apprentissage automatique dans les tâches de prédiction structure-fonction de l’ARN.
Afin de compenser le manque de données annotées, cet article présente un modèle de base qui peut fournir de riches connaissances structurelles et fonctionnelles pour divers types de recherche sur l'ARN - le modèle de fondation de l'ARN (ARN-FM). En tant que premier modèle de base d'ARN au monde formé de manière non supervisée sur la base de 23 millions de séquences d'ARN non marquées, RNA-FM exploite les modèles évolutifs et structurels contenus dans les séquences d'ARN.
Il convient de noter que RNA-FM n'a besoin que de correspondre à un simple modèle en aval ou de fournir uniquement une intégration, et il peut atteindre de bien meilleures performances que SOTA dans de nombreuses tâches en aval, telles que l'amélioration de la prédiction de la structure secondaire de 20 % et de la distance. la prédiction cartographique peut être améliorée de 30 %. Des expériences à grande échelle ont prouvé que le modèle est hautement généralisable et peut même être utilisé pour le COVID-19 et les fragments régulateurs de l’ARNm.

- Préimpression papier : https://arxiv.org/abs/2204.00300
- Code et modèle : https://github.com/ml4bio/RNA - FM
- Serveur : https://proj.cse.cuhk.edu.hk/rnafm
Introduction
Ces dernières années, les méthodes de calcul biologique basées sur l'apprentissage profond ont fait des percées dans le domaine de protéines L'étape la plus célèbre est le cadre de prédiction de la structure 3D des protéines de bout en bout AlphaFold2 développé par l'équipe Google DeepMind. Cependant, les protéines ne sont qu'un type parmi de nombreuses molécules biologiques (ADN/ARN), en tant que source de production de protéines, elles contiennent plus d'informations de base que ces dernières et ont une valeur de recherche plus importante.
De manière générale, les protéines sont les produits de la traduction de l'ARN utilisé pour le codage, c'est-à-dire qu'un ARNm fixe peut être traduit en une séquence protéique fixe. En fait, cette partie de l’ARN codant ne représente que 2 % de toutes les séquences d’ARN, et les 98 % restants sont des ARN non codants (ARNnc). Bien que les ARNnc ne soient pas directement « traduits » en protéines, ils se replient en structures tertiaires dotées de fonctions spécifiques et jouent un rôle régulateur dans le processus de traduction de l’ARNm ou d’autres fonctions biologiques. Par conséquent, l’analyse de la structure et de la fonction des ARNnc est une recherche plus fondamentale et plus complexe que l’analyse des protéines.
Cependant, par rapport au domaine des protéines où les méthodes informatiques sont plus matures, la prédiction de la structure et de la fonction basée sur l'ARN en est encore à ses débuts, et les méthodes informatiques initialement applicables au domaine des protéines sont difficiles à migrer directement vers le domaine de l'ARN. . La principale limitation de ces méthodes informatiques est que l’annotation des données ARN est généralement difficile à obtenir et nécessite beaucoup de ressources expérimentales et de temps pour compléter l’annotation d’une petite quantité de données. La plupart des méthodes informatiques nécessitent une grande quantité de données annotées. pour que la supervision atteigne des performances élevées. Bien qu’il n’y ait pas beaucoup de données annotées, le domaine de l’ARN a en réalité accumulé beaucoup de données de séquences non annotées. La méthode de cet article consiste à utiliser ces données non étiquetées pour fournir des informations supplémentaires efficaces pour diverses tâches en aval.
Sur la base de cette considération, Les équipes du laboratoire d'intelligence artificielle de Hong Kong, du MIT, de Fudan et de Shanghai ont proposé un modèle de base Un modèle de base d'ARN formé sur 23 millions de séquences d'ARN pur non marquées de manière non supervisée (ARN-FM) . Bien que les données ne fournissent pas d’informations d’annotation pendant le processus de formation, RNA-FM exploite toujours les modèles évolutifs et structurels contenus dans ces séquences d’ARN de manière non supervisée.
Si RNA-FM peut être appliqué efficacement aux tâches de prédiction de la structure et des fonctions de l'ARN en aval, ces méthodes informatiques bénéficieront sûrement des connaissances résumées par RNA-FM, améliorant ainsi les performances. Le cadre de pré-formation en amont, de migration en aval et d'application de RNA-FM est présenté dans la figure ci-dessous.
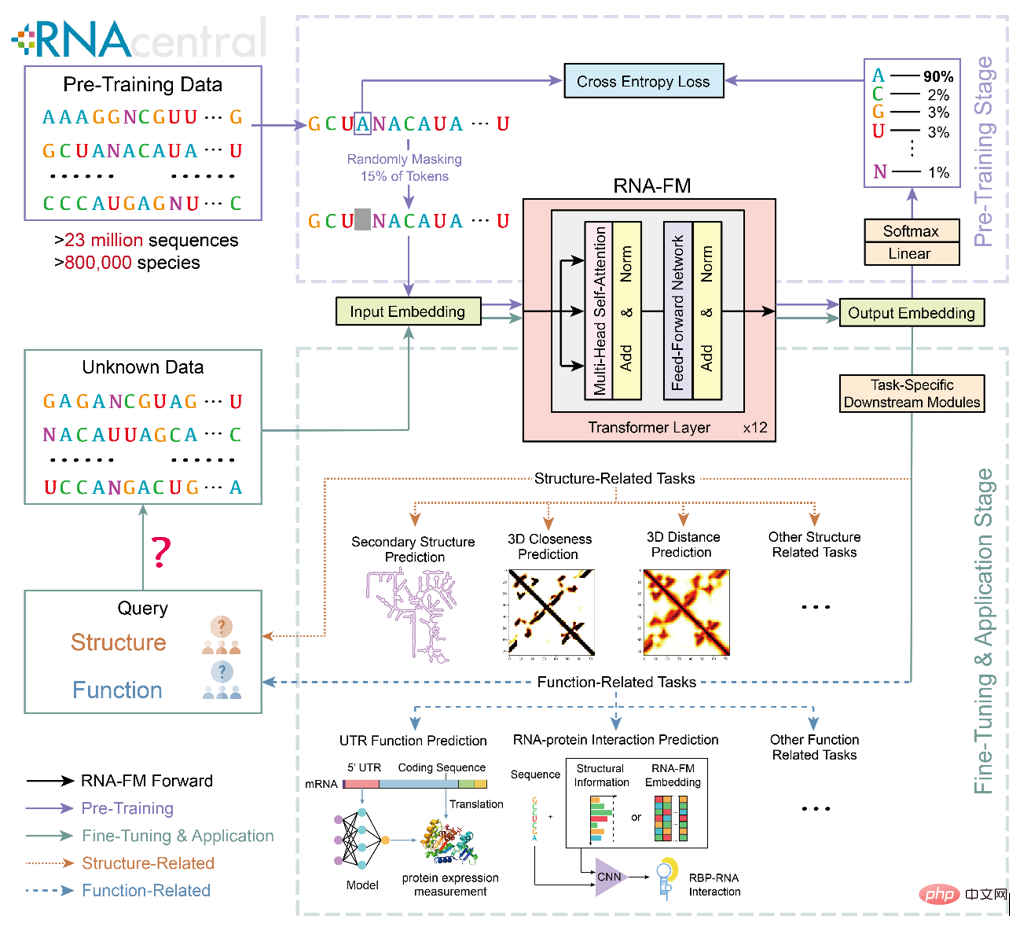
Aperçu de la recherche
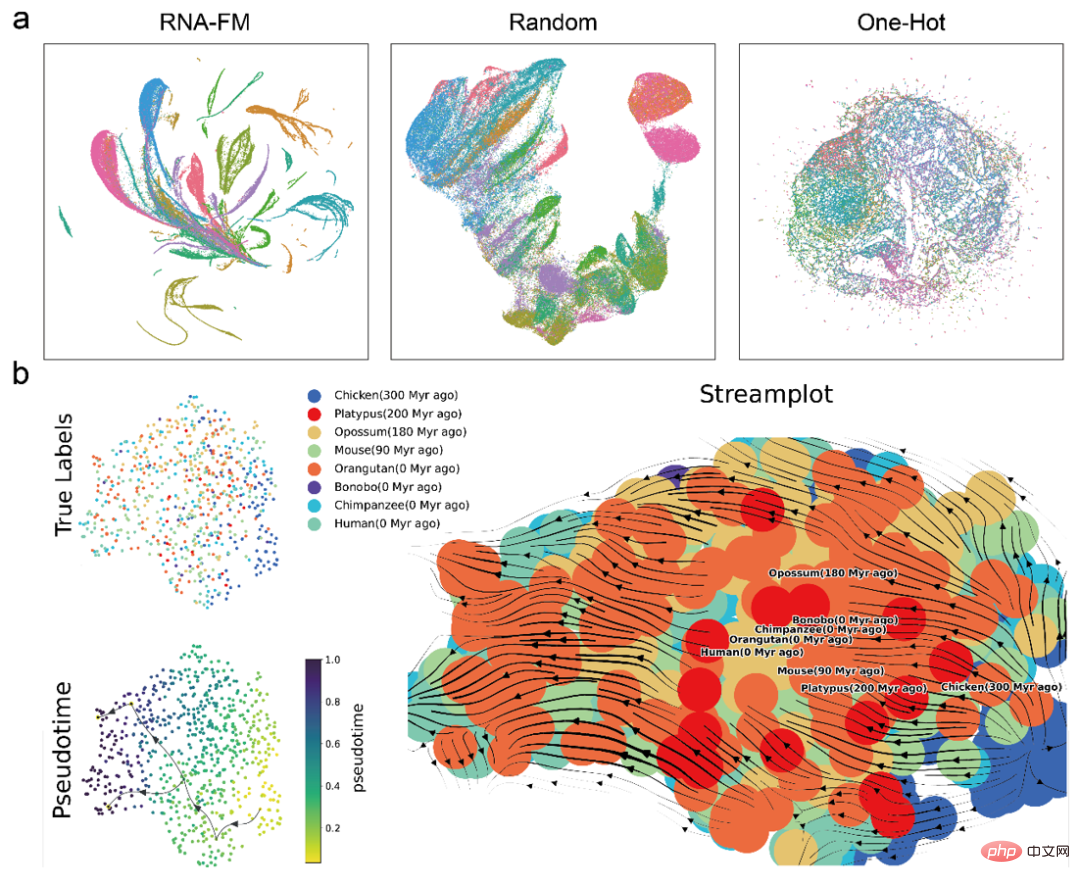
Afin de confirmer si l'ARN-FM pré-entraîné a acquis des « connaissances » à partir d'une grande quantité de données non étiquetées et quel type de « connaissances » il a appris, l'article a mené une série de tests d'intégration Analyse .
Tout d'abord, une simple comparaison de regroupement de diverses caractéristiques a été effectuée directement via UMAP, et il a été constaté que les intégrations d'ARN-FM pré-entraînés formaient des groupes d'espèces d'ARN plus évidents que les autres intégrations. Cela signifie que l’intégration de l’ARN-FM contient des informations structurelles ou fonctionnelles permettant de distinguer les espèces d’ARN.
Ensuite, l'article utilise également l'inférence de trajectoire pour prédire l'évolution des ARNlnc de différentes espèces grâce à l'intégration d'ARN-FM. D'après le diagramme ci-dessous, le pseudo-temps d'évolution prévu entre les espèces est à peu près cohérent avec les informations réelles sur l'évolution des espèces, ce qui indique que l'incorporation d'ARN-FM contient également une partie des informations évolutives.
Il convient de noter qu'il s'agit d'informations communautaires sur les espèces d'ARN ou d'informations évolutives sur lncRNA, l'ARN-FM n'a pas été directement exposé à ces étiquettes pendant la formation. RNA-FM découvre des modèles liés à la structure, à la fonction et à l'évolution à partir de séquences pures de manière entièrement auto-supervisée.
Plus de résultats expérimentaux
En plus d'analyser directement l'intégration de l'ARN-FM, l'article tente également d'introduire l'ARN-FM dans diverses tâches de prédiction de la structure de l'ARN en aval, y compris Structure secondaire, la prédiction des contacts, la prédiction des distances et la prédiction des structures tertiaires ont toutes réalisé des améliorations significatives.
Particulièrement dans la prédiction de la structure secondaire, l'article utilise RNA-FM comme épine dorsale et seulement un simple réseau ResNet comme modèle en aval, surpassant les 12 autres états de l'état sur deux ensembles de données publics -art La méthode est 3 à 5 points de pourcentage meilleure que le meilleur UFold sur le score F1. Dans la comparaison directe avec UFold, RNA-FM surpasse UFold dans la plupart des catégories d'ARN. Si RNA-FM est combiné avec E2Efold, une amélioration supplémentaire des performances de 5 % peut être obtenue.

Afin de vérifier la valeur d'application pratique du modèle, l'article utilise RNA-FM pour effectuer une analyse complète des données COVID-19 , y compris l'utilisation de RNA-FM pour prédire avec précision le COVID -19 éléments régulateurs clés du génome de référence (29 870 nt) et utiliser l’intégration d’ARN-FM pour prédire approximativement les tendances évolutives des principales variantes du COVID-19. 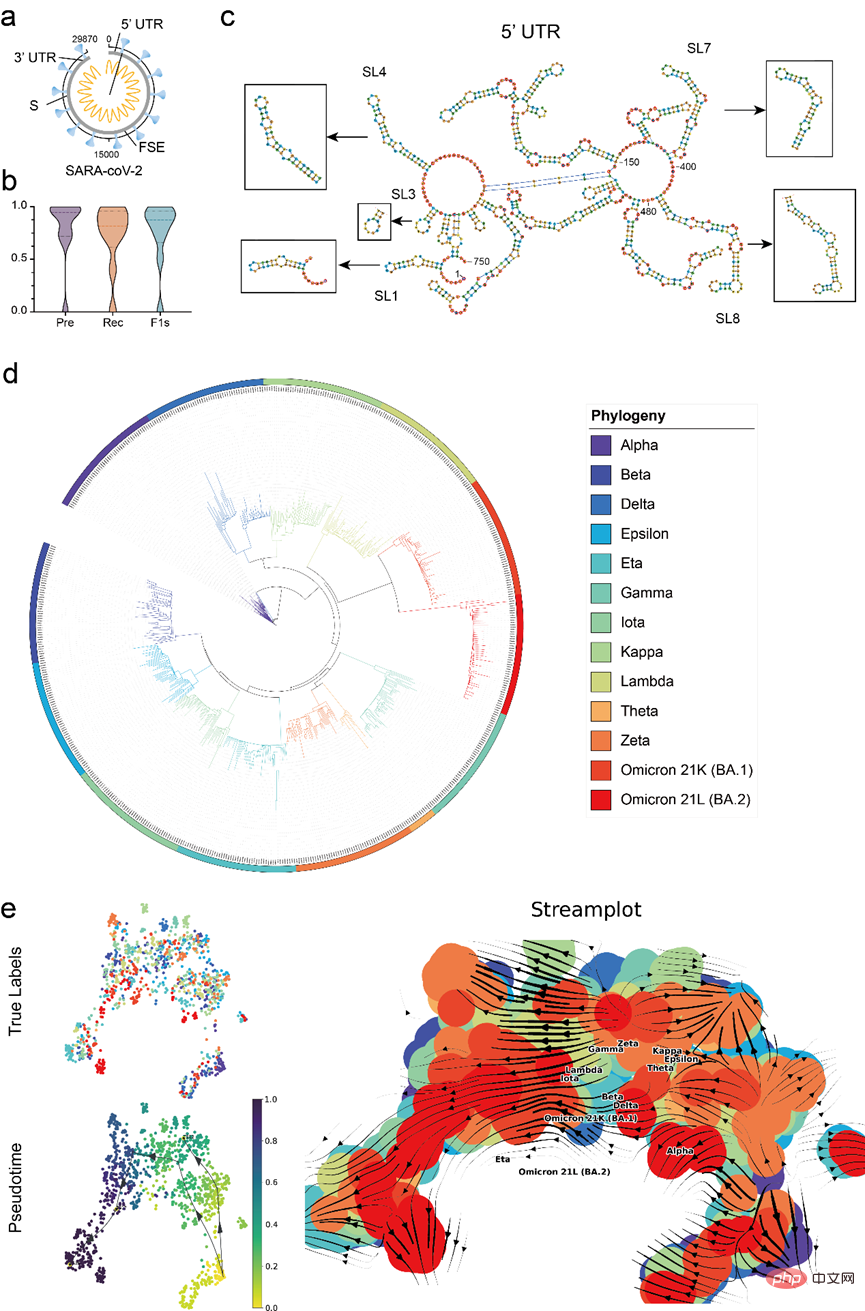
De manière générale, la structure d'une molécule détermine sa fonction. Puisque l'ARN-FM peut parfaitement accomplir la tâche de prédiction de la structure de l'ARN, l'ARN-FM peut-il également être utilisé pour améliorer les résultats de la prédiction de la fonction ?
Par conséquent, l'article tente en outre d'introduire l'ARN-FM dans les tâches de prédiction de la fonction de l'ARN en aval, telles que l'utilisation de l'intégration de l'ARN-FM pour prédire les rôles des protéines ARN.
Des expériences ont prouvé que l'introduction de l'intégration d'ARN-FM a amélioré les performances du modèle et, dans certains cas, a permis d'obtenir des résultats de prédiction qui correspondent aux informations de structure secondaire réelles en entrée.
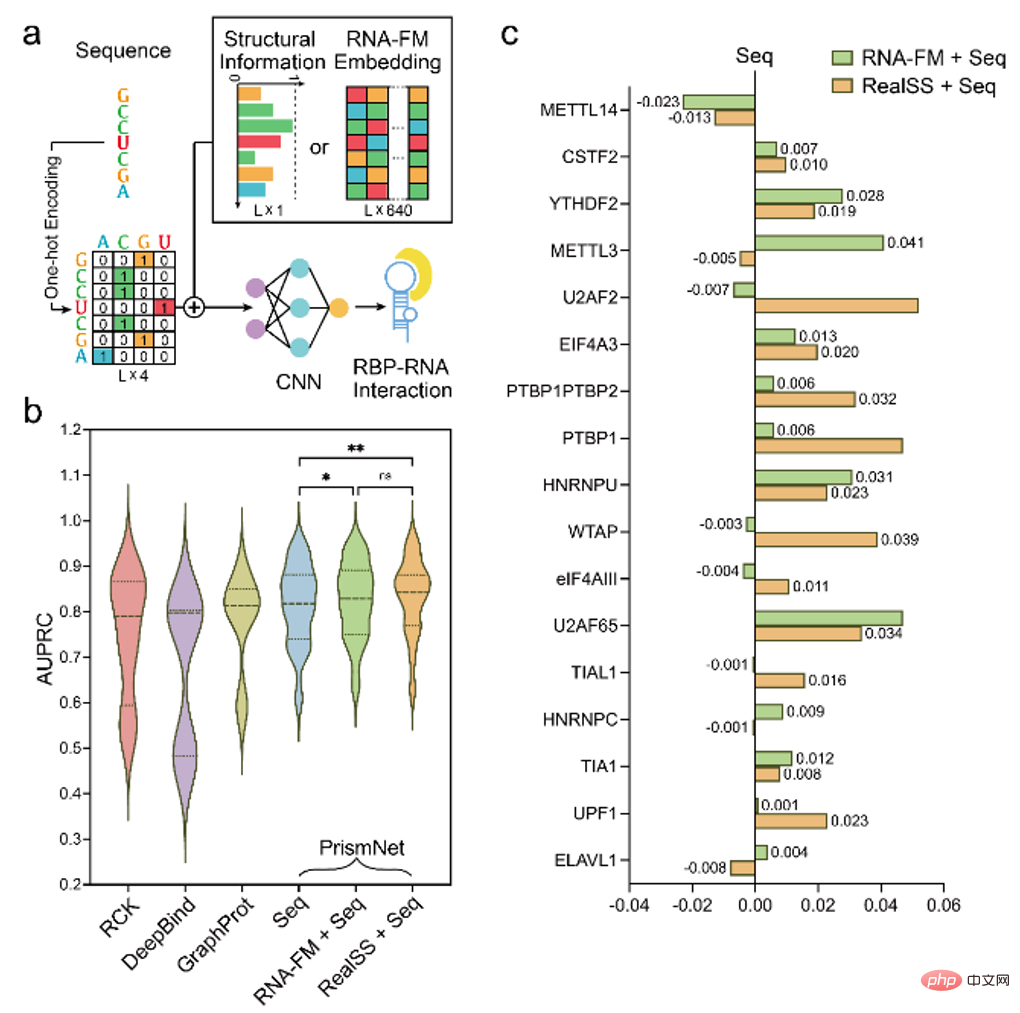
Afin d'explorer si l'ARN-FM basé sur la formation d'ARNnc peut être généralisé à d'autres ARN, l'articletente enfin d'utiliser l'ARN-FM pour prédire la fonction de l'expression des protéines sur la base du 5'UTR sur l'ARNm. Bien que l'ARNm n'appartienne pas à l'ARNnc, le 5'UTR qu'il contient est une région qui n'est pas traduite mais qui a des fonctions régulatrices, ce qui est cohérent avec les caractéristiques de l'ARNnc et n'apparaît pas dans les données d'entraînement.
Comme vous pouvez le voir sur la figure ci-dessous, les modèles qui incluent l'intégration RNA-FM sont toujours meilleurs que les modèles qui n'en incluent pas. Bien que l’amélioration des performances soit relativement limitée, elle montre en partie que RNA-FM possède également une certaine capacité de généralisation sur les données non-ncRNA.

Conclusion
En général, cet article pré-entraîne le modèle de langage RNA-FM avec des données de séquence d'ARN non étiquetées et effectue une vérification complète sur une série de tâches différentes telles que la structure ou la fonction par des méthodes directes ou indirectes, prouvant que l'ARN. -FM peut en effet améliorer efficacement les performances des méthodes de calcul dans les tâches en aval.
L'émergence de l'ARN-FM a atténué dans une certaine mesure la situation actuelle des données annotées par l'ARN et a fourni à d'autres chercheurs une interface pratique pour accéder à de grandes quantités de données non étiquetées. Elle servira de modèle de base dans le domaine de l'ARN. . Apporter un soutien et une aide solides à diverses recherches dans ce domaine.
À propos de l'auteur
Cet article a deux co-premiers auteurs. Chen Jiayang est assistant de recherche à l'Université chinoise de Hong Kong. Hu Zhihang est doctorant à l'Université chinoise de Hong Kong.
Cet article a deux auteurs correspondants. Sun Siqi, jeune chercheur au Laboratoire des systèmes complexes intelligents de l'Université de Fudan et au Laboratoire d'intelligence artificielle de Shanghai, page d'accueil https://intersun.github.io.
Li Yu, professeur adjoint à l'Université chinoise de Hong Kong, professeur adjoint invité au MIT James Collins Lab, chercheur scientifique au Broad Institute du MIT et de Harvard, chercheur invité au Wyss Institute de l'université de Harvard, Forbes 30 Under 30 Liste Asie – Promotion 2022, Santé et Science. Page d'accueil : https://liyu95.com.
Ce qui précède est le contenu détaillé de. pour plus d'informations, suivez d'autres articles connexes sur le site Web de PHP en chinois!
Articles Liés
Voir plus- Tendances technologiques à surveiller en 2023
- Comment l'intelligence artificielle apporte un nouveau travail quotidien aux équipes des centres de données
- L'intelligence artificielle ou l'automatisation peuvent-elles résoudre le problème de la faible efficacité énergétique des bâtiments ?
- Co-fondateur d'OpenAI interviewé par Huang Renxun : les capacités de raisonnement de GPT-4 n'ont pas encore atteint les attentes
- Bing de Microsoft surpasse Google en termes de trafic de recherche grâce à la technologie OpenAI

