Maison >développement back-end >Tutoriel Python >COMMENT TÉLÉCHARGER UN FICHIER CSV SUR DJANGO REST
COMMENT TÉLÉCHARGER UN FICHIER CSV SUR DJANGO REST
- DDDoriginal
- 2024-11-05 19:25:02878parcourir
Télécharger un fichier CSV vers Django REST (surtout dans un environnement atomique) est une tâche simple, mais m'a laissé perplexe jusqu'à ce que je découvre quelques astuces que je partagerais avec vous.
Dans cet article, j'utiliserai Postman (à la place d'un frontend) et partagerai également ce que vous devez définir sur Postman pour l'envoi de demandes via des images.
Ce que nous désirons
- Téléchargez CSV via Django Rest vers la base de données
- Rendre l'opération atomique, c'est-à-dire que toute erreur dans n'importe quelle ligne du csv devrait entraîner une restauration complète de l'ensemble de l'opération, afin que nous puissions éviter le stress lié à la coupe du fichier csv, c'est-à-dire le mal de tête lié à l'identification de la partie des lignes qui l'a fait. la base de données et celles qui ne l'ont pas fait en raison d'une erreur à mi-chemin !! (entrée partielle). Nous voulons donc un truc tout ou rien !!
Méthode
- En supposant que Django et Django REST soient déjà installés, la première étape serait d'installer pandas, une bibliothèque python pour la manipulation de données.
pip installe des pandas
- Suivant dans Postman : dans l'onglet corps, sélectionnez les données du formulaire et ajoutez une clé (n'importe quel nom arbitraire). Dans cette même cellule, passez la souris à l’extrême droite de la cellule et utilisez la liste déroulante pour changer l’option de texte en fichier. Postman définira automatiquement Content-Type sur multipart/form-data dans les en-têtes au moment où vous faites cela.
Pour la cellule de valeur, cliquez sur le bouton « Sélectionner les fichiers » et téléchargez le CSV. Vérifiez la capture d'écran ci-dessous

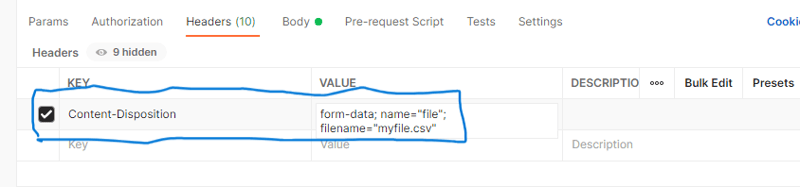
Sous les en-têtes, définissez Content-Disposition et la valeur sur form-data ; nom="fichier"; filename="votre_nom_fichier.csv". Remplacez your_file_name.csv par votre nom de fichier réel. Vérifiez la capture d'écran ci-dessous.
- Dans les vues Django, le code est le suivant :
from rest_framework import status
from rest_framework.views import APIView
from rest_framework.parsers import FileUploadParser
from rest_framework.response import Response
from .models import BiodataModel
from django.db import transaction
import pandas as pd
class UploadCSVFile(APIView):
parser_classes = [FileUploadParser]
def post(self,request):
csv_file = request.FILES.get('file')
if not csv_file:
return Response({"error": "No file provided"}, status=status.HTTP_400_BAD_REQUEST)
# Validate file type
if not csv_file.name.endswith('.csv'):
return Response({"error": "File is not CSV type"}, status=status.HTTP_400_BAD_REQUEST)
df = pd.read_csv(csv_file, delimiter=',',skiprows=3,dtype=str).iloc[:-1]
df = df.where(pd.notnull(df), None)
bulk_data=[]
for index, row in df.iterrows():
try:
row_instance= BiodataModel(
name=row.get('name'),
age=row.get('age'),
address =row.get('address'))
row_instance.full_clean()
bulk_data.append(row_instance)
except Exception as e:
return Response({"error": f'Error at row {index + 2} -> {e}'}, status=status.HTTP_400_BAD_REQUEST)
try:
with transaction.atomic():
BiodataModel.objects.bulk_create(bulk_data)
except Exception as e:
return Response({"error": f'Bulk create error--{e}'}, status=status.HTTP_400_BAD_REQUEST)
return Response({"msg":"CSV file processed successfully"}, status=status.HTTP_201_CREATED)
Explication du code ci-dessus :
Le code commence par l'importation des packages nécessaires, la définition d'une vue basée sur les classes et la définition d'une classe d'analyseur (FileUploadParser). La première partie de la méthode post dans la classe tente d'obtenir le fichier de request.FILES et de vérifier sa disponibilité.
Ensuite une validation mineure vérifie qu'il s'agit bien d'un CSV en vérifiant l'extension.
La partie suivante le charge dans un dataframe pandas (un peu comme une feuille de calcul) :
df = pd.read_csv(csv_file, delimiter=',',skiprows=3,dtype=str).iloc[:-1]
Je vais expliquer certains des arguments passés à la fonction de chargement :
skiproues
En lisant le fichier csv chargé, il convient de noter que le csv dans ce cas est transmis sur un réseau, donc certaines métadonnées telles que des éléments sont ajoutées au début et à la fin du fichier. Ces choses peuvent être ennuyeuses et ne sont pas sous forme de valeurs séparées par des virgules (csv), elles peuvent donc en fait générer des erreurs d'analyse. Cela explique pourquoi j'ai utilisé skiprows=3, pour ignorer les 3 premières lignes contenant les métadonnées et l'en-tête et atterrir directement sur le corps du csv. Si vous supprimez les skiprows ou utilisez un nombre inférieur, vous pourriez peut-être obtenir une erreur du type : Erreur de tokenisation des données. Erreur C ou vous remarquerez peut-être les données à partir de l'en-tête.
dtype=str
Pandas aime faire preuve d'intelligence en essayant de deviner le type de données de certaines colonnes. Je voulais toutes les valeurs sous forme de chaîne, j'ai donc utilisé dtype=str
délimiteur
Spécifie comment les cellules sont séparées. La valeur par défaut est généralement une virgule.
iloc[:-1]
J'ai dû utiliser iloc pour découper le dataframe, en supprimant les métadonnées à la fin du df.
Ensuite, la ligne suivante df = df.where(pd.notnull(df), None) convertit toutes les valeurs NaN en None. NaNi est une valeur de remplacement que les pandas utilisent pour représenter Aucun.
Le bloc suivant est un peu délicat. Nous parcourons chaque ligne du dataframe, instancions les données de ligne avec le BiodataModel, effectuons une validation au niveau du modèle (et non au niveau du sérialiseur) avec la méthode full_clean() car la création en bloc contourne la validation de Django, puis ajoutons nos opérations de création à une liste appelée données_en vrac. Ouais, ajoutez pas encore exécuté ! N'oubliez pas que nous essayons d'effectuer une opération atomique (au niveau du lot), nous voulons donc tout ou rien. Enregistrer les lignes individuellement ne nous donnera pas un comportement tout ou rien.
Puis pour la dernière partie significative. Dans un bloc transaction.atomic() (qui fournit un comportement tout ou rien), nous exécutons BiodataModel.objects.bulk_create(bulk_data) pour enregistrer toutes les lignes à la fois.
Encore une chose. Notez la variable d'index et le bloc except dans la boucle for. Dans le message d'erreur du bloc except, j'ai ajouté 2 à la variable d'index dérivée de df.iterrows() car la valeur ne correspondait pas exactement à la ligne sur laquelle elle se trouvait, lorsqu'elle était examinée dans un fichier Excel. Le bloc except détecte toute erreur et construit un message d'erreur ayant le numéro de ligne exact lorsqu'il est ouvert dans Excel, afin que le téléchargeur puisse facilement localiser la ligne dans le fichier Excel !
Merci d'avoir lu !!!
VERSIONS DES OUTILS UTILISÉS
from rest_framework import status
from rest_framework.views import APIView
from rest_framework.parsers import FileUploadParser
from rest_framework.response import Response
from .models import BiodataModel
from django.db import transaction
import pandas as pd
class UploadCSVFile(APIView):
parser_classes = [FileUploadParser]
def post(self,request):
csv_file = request.FILES.get('file')
if not csv_file:
return Response({"error": "No file provided"}, status=status.HTTP_400_BAD_REQUEST)
# Validate file type
if not csv_file.name.endswith('.csv'):
return Response({"error": "File is not CSV type"}, status=status.HTTP_400_BAD_REQUEST)
df = pd.read_csv(csv_file, delimiter=',',skiprows=3,dtype=str).iloc[:-1]
df = df.where(pd.notnull(df), None)
bulk_data=[]
for index, row in df.iterrows():
try:
row_instance= BiodataModel(
name=row.get('name'),
age=row.get('age'),
address =row.get('address'))
row_instance.full_clean()
bulk_data.append(row_instance)
except Exception as e:
return Response({"error": f'Error at row {index + 2} -> {e}'}, status=status.HTTP_400_BAD_REQUEST)
try:
with transaction.atomic():
BiodataModel.objects.bulk_create(bulk_data)
except Exception as e:
return Response({"error": f'Bulk create error--{e}'}, status=status.HTTP_400_BAD_REQUEST)
return Response({"msg":"CSV file processed successfully"}, status=status.HTTP_201_CREATED)
Ce qui précède est le contenu détaillé de. pour plus d'informations, suivez d'autres articles connexes sur le site Web de PHP en chinois!

