Maison >Périphériques technologiques >IA >Une équipe de l'Institut de technologie informatique de l'Académie chinoise des sciences a proposé CarbonNovo, une conception de novo de bout en bout basée sur l'IA de structures et de séquences protéiques.
Une équipe de l'Institut de technologie informatique de l'Académie chinoise des sciences a proposé CarbonNovo, une conception de novo de bout en bout basée sur l'IA de structures et de séquences protéiques.
- 王林original
- 2024-08-21 21:09:32805parcourir

Éditeur | ScienceAI
Récemment, l'équipe de recherche dirigée par Zhang Haicang de l'Institut de technologie informatique de l'Académie chinoise des sciences a proposé à CarbonNovo de concevoir conjointement la protéine structure et séquence du squelette de bout en bout.
Cette recherche a été publiée lors de la conférence d'apprentissage automatique ICML 2024 sous le titre "CarbonNovo : Joint Design of Protein Structure and Sequence Using a Unified Energy-based Model".

Introduction générale
Les protéines sont des macromolécules importantes pour les fonctions biologiques. La conception de protéines de novo vise à créer des protéines entièrement nouvelles et a de larges applications dans le développement de médicaments et l’ingénierie enzymatique.
Ces dernières années, la conception de novo de protéines basée sur l'IA s'est développée rapidement et a été utilisée avec succès dans des domaines tels que la conception d'anticorps et la conception de médicaments à petites protéines. Par rapport aux méthodes de conception traditionnelles, elle a considérablement amélioré le taux de réussite et l'efficacité de la conception.
La conception de protéines par l'IA a bénéficié de deux avancées technologiques majeures ces dernières années :
Le premier est le modèle AlphaFold2 dans le domaine de la prédiction de la structure des protéines, qui fournit une architecture de modèle de réseau neuronal de base et une séquence de protéines pour le domaine de l'informatique des protéines, y compris la conception de protéines. Méthodes de représentation et de représentation de structure, ainsi que des stratégies de formation avancées (formation à la distillation, formation de bout en bout) et d'autres technologies
Deuxièmement, le développement rapide de l'AIGC dans le domaine du texte, de l'image et de la vidéo ; La génération fournit un modèle de génération mature pour la conception de protéines, par exemple DDPM, SDE, Flow Matching, Bayesian Flow Network, etc. Les idées principales des modèles de conception de protéines représentatifs, tels que RFDiffusion et Chroma, sont d'intégrer ces deux technologies et d'intégrer le réseau de représentation de séquences et de structures protéiques dans un cadre de modèle génératif basé sur l'IA.
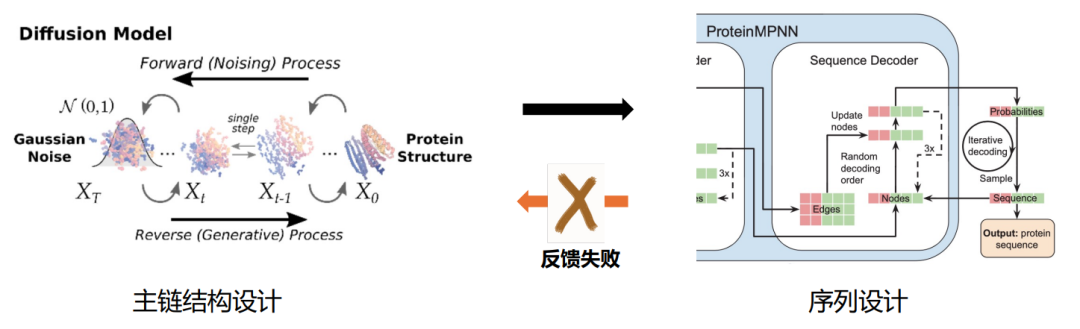
La conception de protéines de novo comprend principalement deux étapes : la conception de la structure du squelette protéique et la conception des séquences (Figure 1). En conséquence, les modèles traditionnels actuels adoptent généralement un cadre « en deux étapes » : pendant le processus de formation, le module de conception de structure et le module de conception de séquence sont formés séparément pendant le processus d'inférence, la structure de la chaîne principale est d'abord générée, puis la structure optimale ; La structure de la chaîne principale est générée. De manière représentative, les deux logiciels RFDiffusion et ProteinMPNN sont utilisés sur le terrain pour générer tour à tour la structure et la séquence de la chaîne principale.
Le cadre « en deux étapes » pour la conception de protéines de novo présente des limites inhérentes :
CarbonNovo réalise une conception conjointe de bout en bout de la structure et de la séquence
En réponse aux limites du cadre « en deux étapes » pour la conception des protéines, une équipe de recherche dirigée par Zhang Haicang de l'Institute of Computing Technology, L'Académie chinoise des sciences a proposé à CarbonNovo de concevoir conjointement des structures de chaînes principales de protéines de bout en bout. L'article a été récemment publié lors de la conférence sur l'apprentissage automatique ICML 2024.
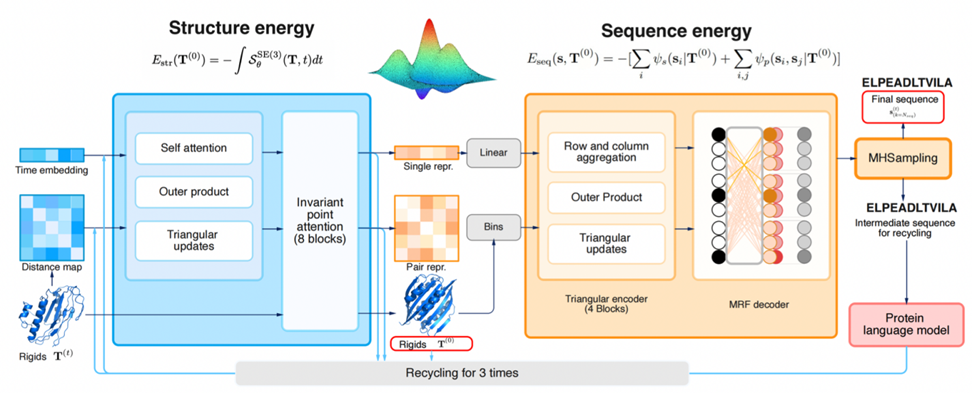
Figure 2 : CarbonNovo génère des structures et des séquences protéiques de bout en bout. (Source : article)
Les principales contributions de CarbonNovo sont résumées comme suit :
Modèle énergétique commun de la structure-séquence des protéines
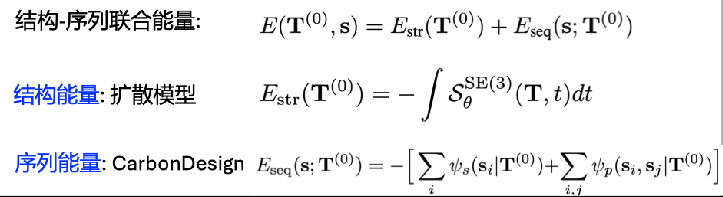
在經典物理模型下,天然蛋白質構象具有比較低的自由能,這也是蛋白質結構預測和設計的一般假設。基於此,CarbonNovo 建立了蛋白質結構和序列的聯合能量模型:
圖 2 展示了 CarbonNovo 的具體生成過程:
CarbonNovo 產生蛋白質結構-序列的效能評估

圖 4:在不同蛋白質長度下的表現比較。 (資料來源:論文)

消融實驗
#🎜#p (資料來源:論文)

#🎜#f fee#a#p -sheets 結構到all alpha-helices 結構的漸變。 (資料來源:論文)
在影像產生領域,人臉圖片插值/漸層是生成模型比較經典的應用。作者也嘗試利用 CarbonNovo 進行蛋白質結構的內插。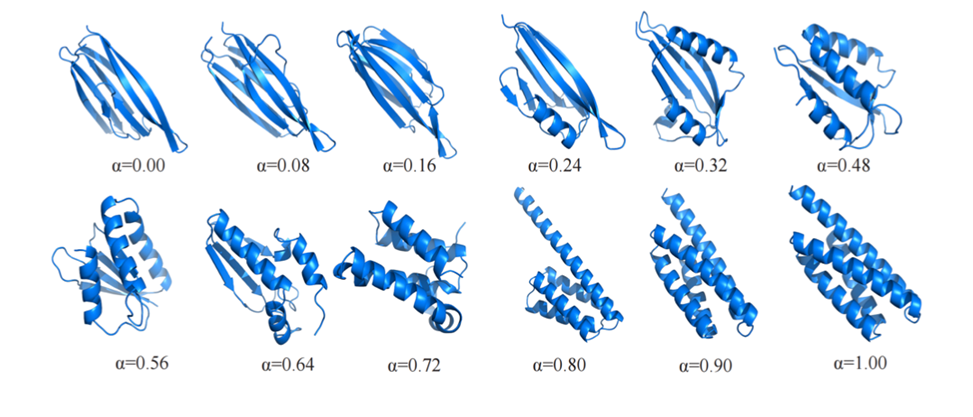 圖 5 展示了一個代表例子,隨著在隱空間逐漸增加 all alpha-helices 結構向量的權重,產生的 all beta-sheets 結構會逐漸過渡到 all alpha-helices 結構。
圖 5 展示了一個代表例子,隨著在隱空間逐漸增加 all alpha-helices 結構向量的權重,產生的 all beta-sheets 結構會逐漸過渡到 all alpha-helices 結構。
這是領域內第一個關於蛋白質結構的插值實驗,也體現了 CarbonNovo 學習到的蛋白質隱空間比較緊緻。
結語
最後作者指出,雖然 CarbonNovo 主要專注於蛋白質單體設計,但它也可以很容易擴展到蛋白質複合物設計和條件設計,如多肽設計、抗體設計等。
作者團隊目前正在與生物實驗團隊合作,以濕實驗的方式驗證 CarbonNovo 設計的蛋白質。
作者所在 CarbonMatrix 團隊,長期致力於 AI 蛋白質設計和 AI 藥物設計,正在建立生物大分子結構設計和預測的統一生成模型。
其研究成果多次發表在ICML、NeurIPS 等頂級機器學習會議和 Nature Machine Intelligence、Nature Communications 等頂級學術期刊上,目前也在和生物實驗室合作,積極推動AI 模型在藥物設計領域的產業化落地。
Ce qui précède est le contenu détaillé de. pour plus d'informations, suivez d'autres articles connexes sur le site Web de PHP en chinois!
Articles Liés
Voir plus- Tendances technologiques à surveiller en 2023
- Comment l'intelligence artificielle apporte un nouveau travail quotidien aux équipes des centres de données
- L'intelligence artificielle ou l'automatisation peuvent-elles résoudre le problème de la faible efficacité énergétique des bâtiments ?
- Co-fondateur d'OpenAI interviewé par Huang Renxun : les capacités de raisonnement de GPT-4 n'ont pas encore atteint les attentes
- Bing de Microsoft surpasse Google en termes de trafic de recherche grâce à la technologie OpenAI

