Maison >Périphériques technologiques >IA >Pour prédire la spécificité de liaison protéine-ADN, l'équipe de l'USC développe une nouvelle méthode d'apprentissage profond géométrique
Pour prédire la spécificité de liaison protéine-ADN, l'équipe de l'USC développe une nouvelle méthode d'apprentissage profond géométrique
- PHPzoriginal
- 2024-08-19 13:34:101293parcourir
编辑 | 萝卜皮
预测蛋白质-DNA 结合特异性是一项具有挑战性但又至关重要的任务,有助于理解基因调控。蛋白质-DNA 复合物通常与选定的 DNA 靶位结合,而蛋白质则以不同程度的结合特异性与广泛的 DNA 序列结合。这些信息无法在单一结构中直接获取。
为了获取这些信息,南加州大学(University of Southern California)和华盛顿大学(University of Washington)的研究人员提出了深度结合特异性预测器(DeepPBS),这是一种几何深度学习模型,旨在根据蛋白质-DNA 结构预测结合特异性。
DeepPBS 可以提取可解释的界面残基蛋白质重原子重要性得分。当在蛋白质残基水平上聚合时,这些得分通过诱变实验进行验证。应用于针对特定 DNA 序列的设计蛋白质,DeepPBS 被证明可以预测实验测量的结合特异性。
该研究以「Geometric deep learning of protein–DNA binding specificity」为题,于 2024 年 8 月 5 日发布在《Nature Methods》。

转录因子通过与特定DNA序列的结合来调节生命过程,这种结合机制包括静电相互作用、脱氧核糖堆积效应,以及氢键的形成等。
蛋白质-DNA的结构信息通常通过X射线晶体学、核磁共振波谱或低温电子显微镜等实验方法获取,并存储在蛋白质数据库(PDB)中。这些结构通常展示了结合的 DNA 序列及其物理化学相互作用,但无法涵盖所有可能的结合序列。
另一方面,高通量实验如蛋白质结合微阵列、SELEX-seq 等能够捕捉潜在的结合序列范围,但缺乏结构信息。
因此,结合结构数据和高通量实验数据对于全面理解转录因子的结合特异性至关重要。
目前,预测特定蛋白质序列在蛋白质家族中的结合特异性,仍然是一个具有挑战性且尚未解决的问题。结合背景下的结构变化以及庞杂的机制多样性加剧了这一困难。
「蛋白质-DNA 复合物的结构包含通常与单个 DNA 序列结合的蛋白质。为了理解基因调控,了解蛋白质与任何 DNA 序列或基因组区域的结合特异性非常重要。」南加州大学的 Remo Rohs 教授说。
在最新的研究中,南加州大学和华盛顿大学的研究人员引入了结合特异性深度预测器 (DeepPBS)。
Rohs 解释道:「DeepPBS 是一种人工智能工具,它取代了高通量测序或结构生物学实验来揭示蛋白质-DNA 结合特异性。」
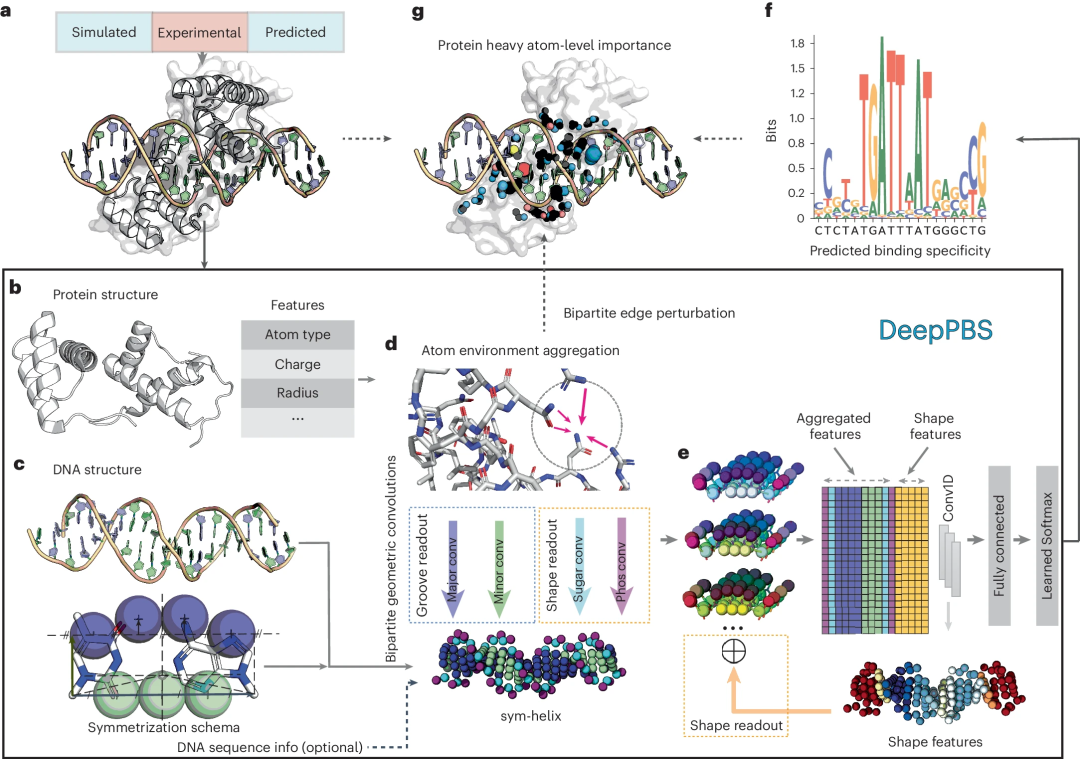
该深度学习模型旨在捕捉蛋白质-DNA 相互作用的物理化学和几何背景,以预测结合特异性,表示为基于给定蛋白质-DNA结构的位置权重矩阵(PWM)。DeepPBS 跨蛋白质家族发挥作用,并充当结构确定和结合特异性确定实验之间的桥梁。
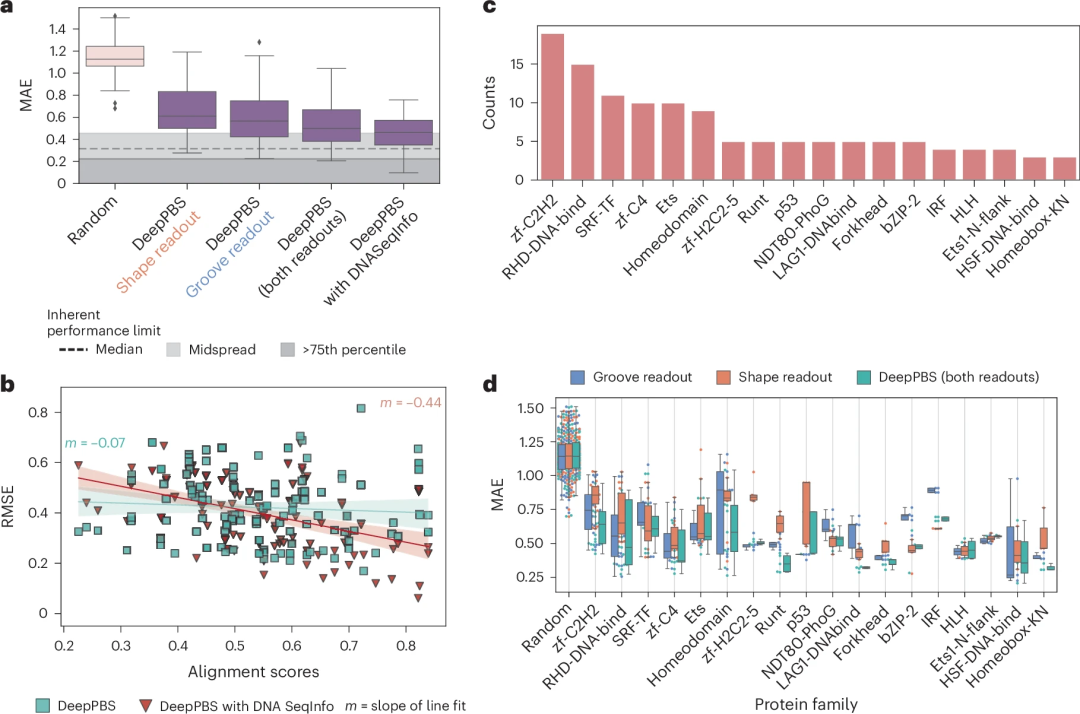
图示:DeepPBS 用于预测跨蛋白质家族的结合特异性的性能。(来源:论文)
DeepPBS 的输入不仅限于实验结构。蛋白质结构预测方法(包括 AlphaFold、OpenFold 和 RoseTTAFold)以及蛋白质-DNA 复合物建模器(例如 RoseTTAFoldNA (RFNA)、RoseTTAFold All-Atom、MELD-DNA 和 AlphaFold3)的快速发展,导致可供分析的结构数据数量呈指数级增长。
这一场景凸显了对用于分析蛋白质-DNA 结构的通用计算模型的需求日益增长。研究人员展示了 DeepPBS 如何与结构预测方法结合使用,以预测没有可用实验结构的蛋白质的特异性。
此外,通过使用 DeepPBS 反馈优化结合 DNA,可以改进蛋白质-DNA 复合物的设计。研究人员表明,此流程与最近的家族特定模型 rCLAMPS 相比性能相当,同时更具通用性:具体而言,DeepPBS 不受蛋白质家族限制,可以处理生物组装并可以预测 DNA 侧链偏好。
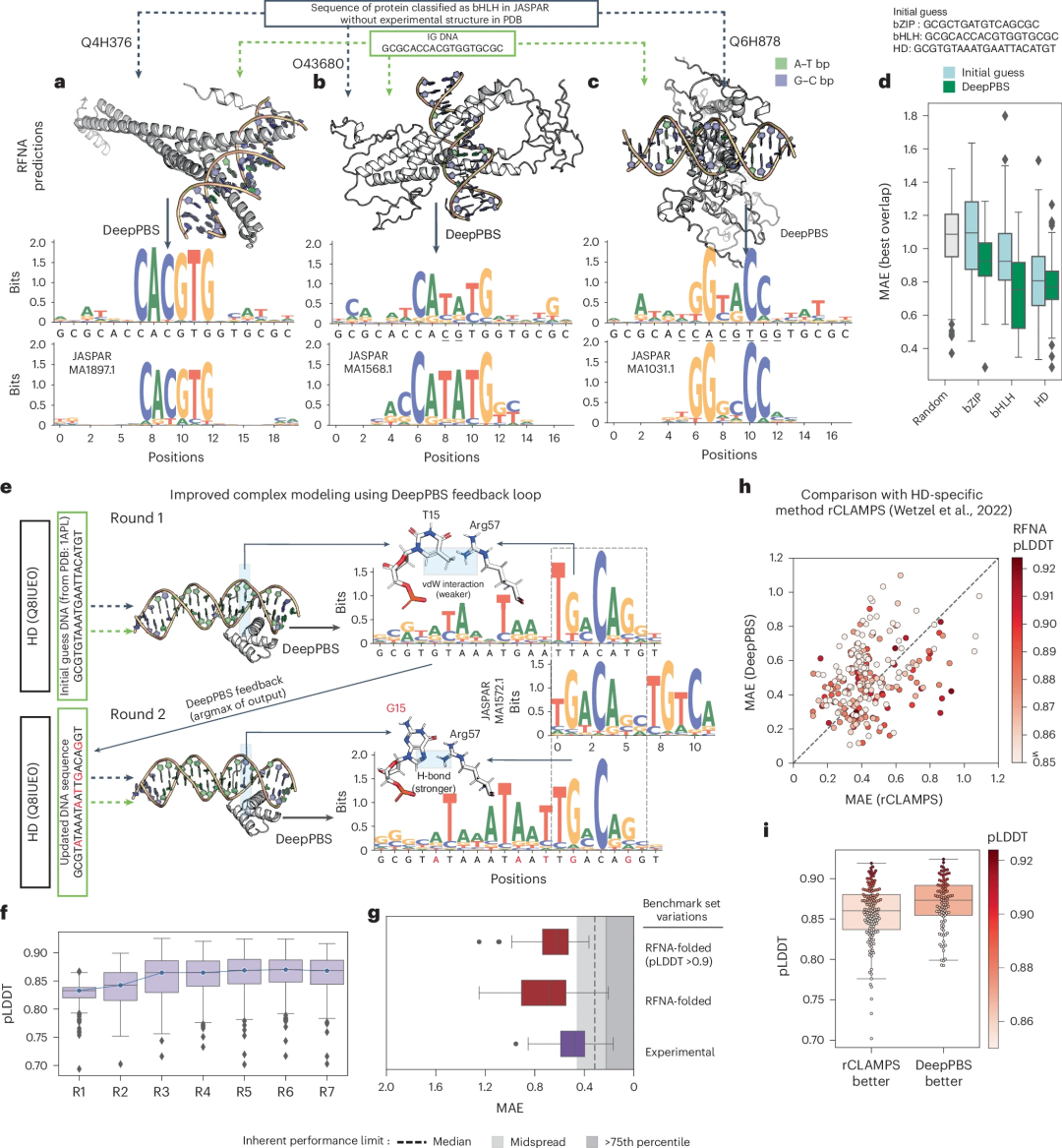
Illustration : Application de DeepPBS pour prédire la structure des complexes protéine-ADN. (Source : article)
En termes d'interprétabilité, les scores « d'importance relative » (RI) de différents atomes lourds dans les protéines interagissant avec l'ADN peuvent être extraits de DeepPBS.
Dans le cadre d'une étude de cas sur des protéines importantes pour le développement du cancer, les chercheurs ont analysé l'interface p53-ADN via ces scores RI et les ont liés à la littérature existante pour validation.
De plus, les scores DeepPBS concordent bien avec les connaissances existantes et peuvent être regroupés pour produire un accord raisonnable avec les expériences de mutagenèse par balayage d'alanine.
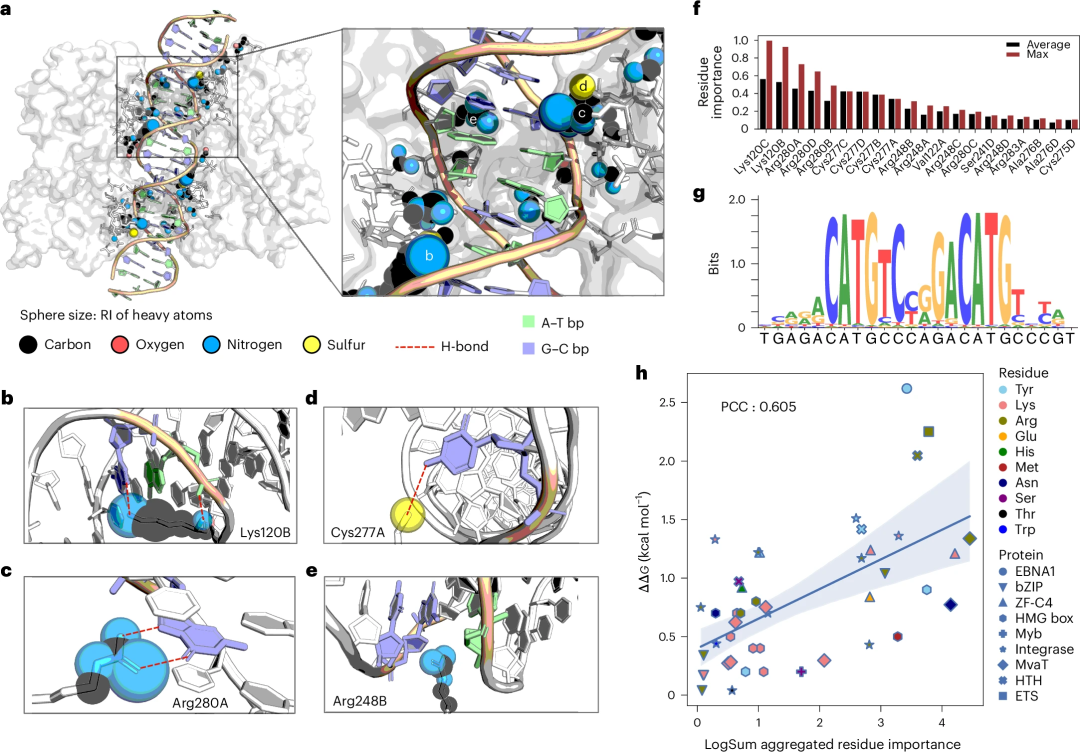
Illustration : Prendre la visualisation des scores d'importance DeepPBS dans l'interface p53-DNA comme exemple pour étudier et effectuer une vérification expérimentale. (Source : article)
Dans d'autres études de preuve de principe, les chercheurs ont appliqué DeepPBS à des complexes protéine-ADN conçus in silico ciblant des séquences d'ADN spécifiques issues d'une expérience récente combinant la conception structurelle et la recherche sur la mutagenèse de l'ADN. DeepPBS peut également être utilisé pour analyser les trajectoires de simulation moléculaire.
"Il est important que les chercheurs trouvent une méthode qui fonctionne pour toutes les protéines et ne se limite pas à une famille de protéines bien étudiée. Cette méthode nous permet également de concevoir de nouvelles protéines", a déclaré Rohs.
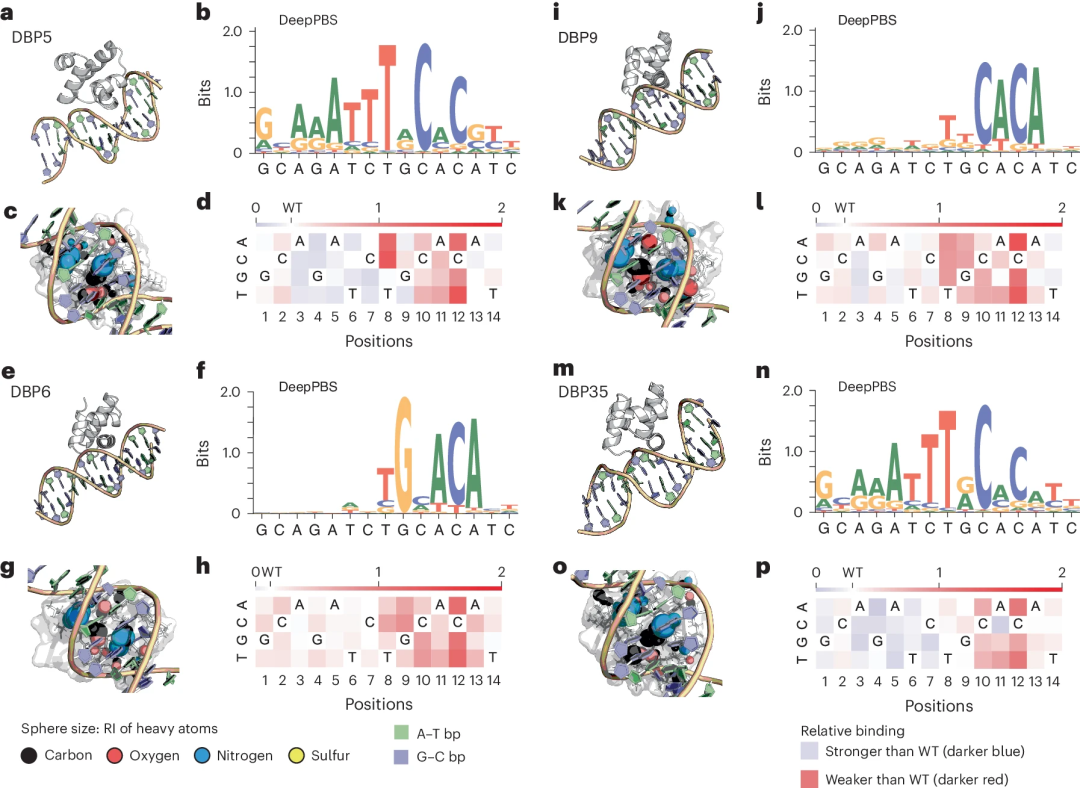
Illustration : Application de DeepPBS à des échafaudages HTH conçus in silico ciblant des séquences d'ADN spécifiques. (Source : article)
La version actuelle de DeepPBS a des limites inhérentes. Il est conçu pour l’ADN double brin et ne fonctionne pas encore avec l’ADN simple brin, l’ARN ou les bases chimiquement modifiées.
Cependant, le modèle a le potentiel d'être étendu pour s'adapter à ces différents scénarios ainsi qu'à d'autres interactions polymère-polymère, et potentiellement à des mutations mécanistes. L'architecture DeepPBS peut être optimisée et étendue en termes d'améliorations applicatives et techniques.
Néanmoins, Rohs a déclaré que DeepPBS aura un large éventail d'applications. Cette nouvelle approche de recherche pourrait accélérer la conception de nouveaux médicaments et traitements ciblant des mutations spécifiques dans les cellules cancéreuses, ainsi que conduire à de nouvelles découvertes en biologie synthétique et à des applications dans la recherche sur l'ARN.
DeepPBS : https://deeppbs.usc.edu
Ce qui précède est le contenu détaillé de. pour plus d'informations, suivez d'autres articles connexes sur le site Web de PHP en chinois!
Articles Liés
Voir plus- Tendances technologiques à surveiller en 2023
- Comment l'intelligence artificielle apporte un nouveau travail quotidien aux équipes des centres de données
- L'intelligence artificielle ou l'automatisation peuvent-elles résoudre le problème de la faible efficacité énergétique des bâtiments ?
- Co-fondateur d'OpenAI interviewé par Huang Renxun : les capacités de raisonnement de GPT-4 n'ont pas encore atteint les attentes
- Bing de Microsoft surpasse Google en termes de trafic de recherche grâce à la technologie OpenAI

