Maison >Périphériques technologiques >IA >Avec une grande efficacité et sans avoir besoin d'étiquettes, l'équipe de Google utilise l'IA pour extraire des données cliniques, améliorer la découverte de gènes et la prédiction des maladies. Elle est publiée dans la sous-revue Nature.
Avec une grande efficacité et sans avoir besoin d'étiquettes, l'équipe de Google utilise l'IA pour extraire des données cliniques, améliorer la découverte de gènes et la prédiction des maladies. Elle est publiée dans la sous-revue Nature.
- WBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBoriginal
- 2024-07-19 21:45:12558parcourir

Éditeur | ScienceAI
Les systèmes de santé modernes génèrent de grandes quantités de données cliniques de haute dimension (HDCD), telles que des cartes de la fonction pulmonaire, la photopléthysmographie (PPG), les enregistrements d'électrocardiogramme (ECG), les tomodensitogrammes et l'imagerie IRM. les données ne peuvent pas être résumées par un seul nombre binaire ou continu.
Comprendre le lien entre notre génome et le HDCD améliorera non seulement notre compréhension de la maladie, mais sera également essentiel au développement de traitements contre la maladie.
Récemment, l'équipe de génomique de Google Research a progressé dans l'utilisation du HDCD pour caractériser les maladies et les caractéristiques biologiques.
L'équipe de recherche a proposé un modèle d'apprentissage profond non supervisé, Representation Learning for Low-Dimensional Embedding Gene Discovery (REGLE), pour découvrir l'association entre les variantes génétiques et le HDCD.
REGLE, en tant que nouvelle méthode de découverte de gènes, peut exploiter des informations cachées dans des données cliniques de grande dimension, est efficace sur le plan informatique, ne nécessite pas d'étiquettes de maladie et peut intégrer des informations provenant de connaissances définies par des experts.
Dans l'ensemble, REGLE contient des informations cliniquement pertinentes au-delà de ce qui est capturé par les signatures existantes définies par des experts, permettant ainsi d'améliorer la découverte de gènes et la prédiction des maladies.
Une recherche pertinente intitulée « L'apprentissage de représentations non supervisées sur des données cliniques de grande dimension améliore la découverte et la prédiction génomiques » a été publiée dans « Nature Genetics » le 8 juillet.

Lien papier : https://www.nature.com/articles/s41588-024-01831-6
Révéler les informations cachées dans le HDCD
Étude sur le lien entre les gènes et le HDCD A Une approche simple consiste à effectuer un GWAS sur chaque coordonnée de données. Par exemple, vous pouvez étudier les changements dans la valeur de chaque pixel d'une image médicale. Cette approche est coûteuse en calcul et a une faible puissance pour détecter des associations significatives en raison des corrélations élevées entre les coordonnées voisines et d'une charge de test multiple importante.
Une approche plus courante consiste à se concentrer sur un petit nombre de caractéristiques définies par des experts (EDF) extraites du HDCD en tant que caractéristiques cibles ou phénotypes pour GWAS. L'EDF peut inclure des caractéristiques cliniquement connues telles que la capacité vitale forcée (CVF) issue de la spirométrie ou le volume expiratoire forcé en 1 seconde (VEMS).
Bien que ces EDF soient des fonctionnalités importantes découvertes par les experts, on suppose qu'ils ne capturent pas entièrement les signaux codés en HDCD, donc exécuter GWAS sur ces signaux peut ne pas exploiter tout le potentiel du HDCD.
REGLE vise à surmonter ces limitations en utilisant des modèles d'auto-encodeurs variationnels (VAE). La méthode se compose de trois étapes principales :
(1) Apprendre la représentation non linéaire, de faible dimension et démêlée (c'est-à-dire l'encodage ou l'intégration) du HDCD via VAE ;
(2) Effectuer un GWAS indépendamment pour chaque coordonnée codée ;
(3) Utiliser les scores de risque polygénique (PRS) à partir des coordonnées de codage comme scores génétiques pour les fonctions biologiques générales, puis potentiellement combiner ces scores pour créer des PRS pour des maladies ou des traits spécifiques (étant donné un petit nombre d'étiquettes de maladie). Notamment, REGLE permet également d'inclure sélectivement les EDF pertinents dans l'entrée du décodeur dans l'architecture VAE modifiée, encourageant ainsi l'encodeur à n'apprendre que les signaux résiduels non représentés par les EDF.
Détection de nouveaux locus génétiques pour la fonction pulmonaire et circulatoire
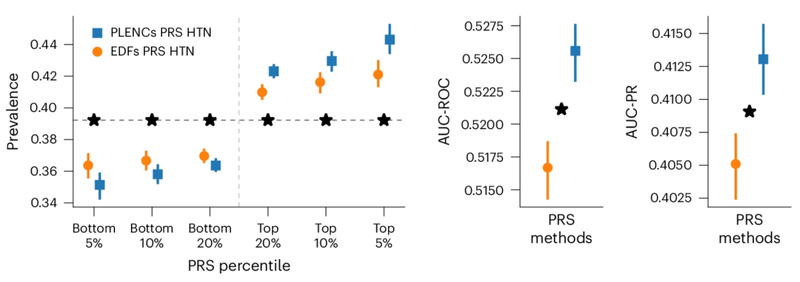
Les chercheurs ont démontré la puissance de REGLE en utilisant deux modalités de données cliniques de grande dimension : la spirométrie, qui mesure la fonction pulmonaire, et la spirométrie, qui mesure la fonction cardiovasculaire. .PPG. Les deux peuvent être collectés de manière non invasive et relativement peu coûteuse dans des cliniques ou sur des appareils portables grand public, et les deux modalités ont des caractéristiques bien connues). Par rapport aux études d'association à l'échelle du génome avec la spirométrie et les signatures PPG des mêmes dimensions, l'étude REGLE sur le codage appris a récupéré la majorité des locus génétiques connus associés à la fonction pulmonaire et circulatoire, tout en détectant également d'autres sites (par exemple , le site important de PPG a augmenté de 45%). Si ces sites sont validés par des analyses plus approfondies et des expériences en laboratoire humide, ils ont le potentiel de devenir de nouvelles cibles médicamenteuses. Score de risque génétique amélioréUn score de risque polygénique (PRS) est un résumé de l'impact estimé de nombreuses variantes génétiques sur un trait spécifique, exprimé sous la forme d'un nombre unique. Les PRS créés par des études d'association pangénomiques sur les inclusions REGLE peuvent être combinés en utilisant seulement quelques signatures de maladie pour générer un PRS pour cette maladie spécifique.研究人員觀察到,與現有方法(例如由專家定義的特徵、PCA 和PRS)相比,由肺量圖編碼創建的肺功能PRS 改善了 COPD 和氣喘預測,並且比風險譜兩端的特徵PRS更有效地將風險組分層。氣喘和COPD 的多個獨立資料集(COPDGene、eMERGE III、Indiana Biobank 和EPIC-Norfolk)中的多個指標(AUC-ROC、AUC-PR 和Pearson 相關性)在統計學上顯著改善,如下所示。

類似地,從 PPG 的 REGLE 嵌入中得到的 PRS 可以改善高血壓和收縮壓 (SBP) 預測。在三個獨立資料集(COPDGene、eMERGE III 和 EPIC-Norfolk)以及英國生物庫的保留測試集中評估了由 PPG 編碼和 PPG 特徵產生的高血壓和 SBP PRS。
觀察到,在多個資料集中,使用來自 PPG 編碼的 PRS 比使用來自專家定義特徵的 PRS 具有一致的改進趨勢,無論是高血壓還是 SBP。

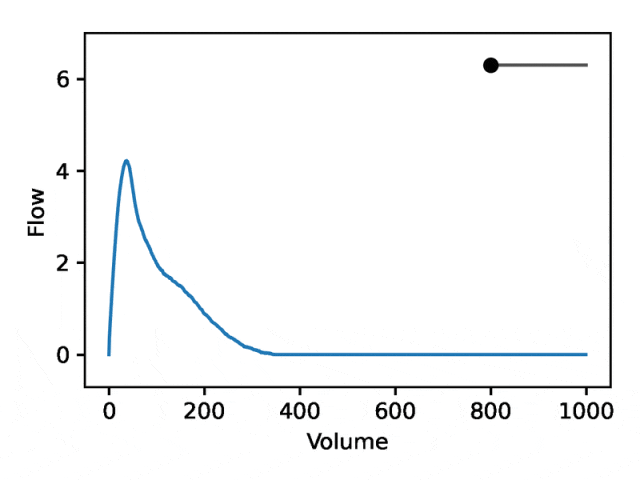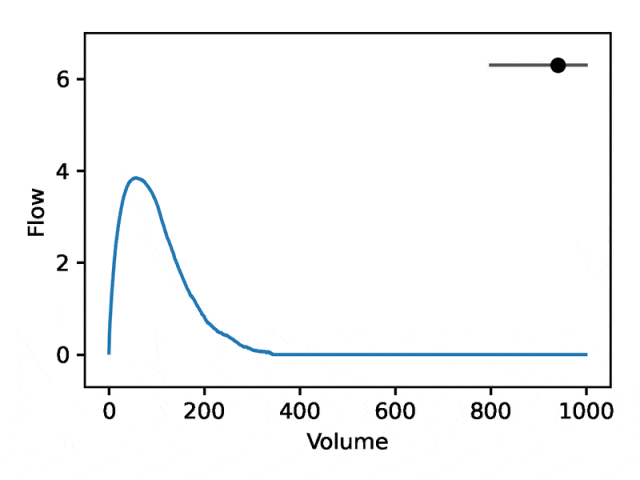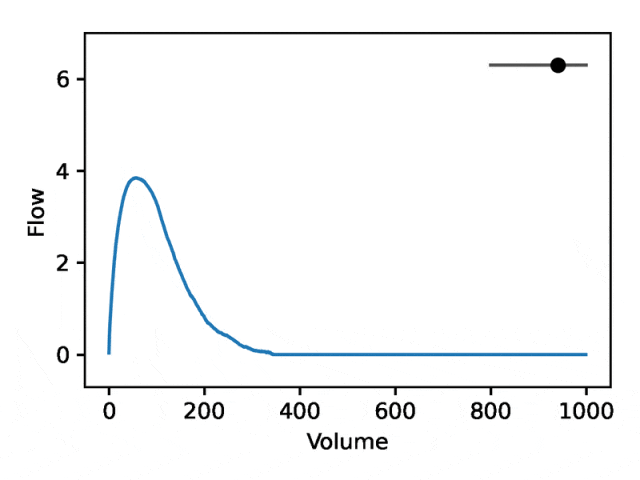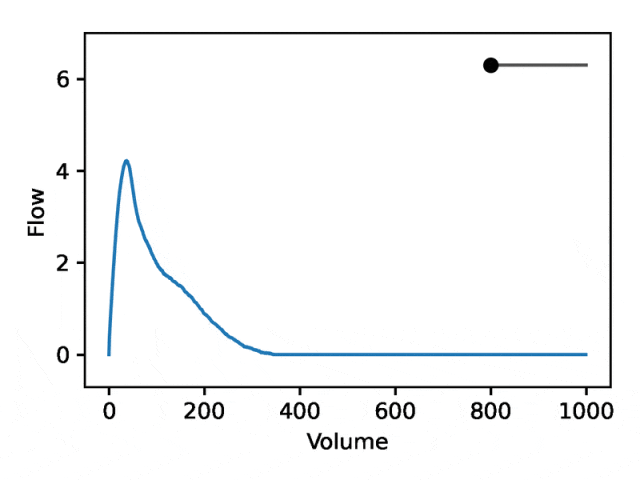
部分可解釋的嵌入
利用REGLE 的生成特性,透過固定專家定義特徵的值並改變一個編碼座標而將其他編碼座標保持為零來研究編碼座標對肺量圖形狀的影響。然後,僅使用訓練模型的解碼器部分產生相應的肺量圖。
典型的流量-體積肺量圖由兩個不同的部分組成:(1)相對較短的部分以達到峰值流量,其中流量隨著體積的增加而單調增加;(2)肺量圖的主要部分,其中流量單調減少。
下圖顯示,改變第一個座標相當於擴大或縮小第二部分(負斜率),同時保持第一部分相對固定。事實上,曲線第二部分的凹度被肺病學家稱為凹陷,這是氣道阻塞的指標,標準 EDF 無法很好地表示出來。
闡明人類特徵和疾病的遺傳基礎
REGLE 是一種無監督學習方法,可執行遺傳分析、改進的新基因位點發現和風險預測。由於難以大規模手動發現 EDF,因此無監督學習 HDCD 表示對基因組發現很有吸引力。
REGLE 框架也透過修改傳統的 VAE 架構來支援在建模中原則性地使用這些特徵。在兩種臨床數據模式(肺量圖和 PPG)中展示了 REGLE,它們可以在臨床環境中進行常規測量,也可以透過智慧型手機或穿戴式裝置被動和非侵入性地測量。
REGLE 提供了一種在沒有標記數據的情況下識別遺傳對器官功能影響的機制,並允許將專家特徵納入模型。它還提供了一種使用很少的標籤來創建疾病和特徵特異性 PRS 的方法。未來,這種類似的方法將越來越多地用於進一步闡明人類特徵和疾病的遺傳基礎。
參考內容:https://research.google/blog/harnessing-hidden-genetic-information-in-clinical-data-with-regle/
Ce qui précède est le contenu détaillé de. pour plus d'informations, suivez d'autres articles connexes sur le site Web de PHP en chinois!
Articles Liés
Voir plus- Tendances technologiques à surveiller en 2023
- Comment l'intelligence artificielle apporte un nouveau travail quotidien aux équipes des centres de données
- L'intelligence artificielle ou l'automatisation peuvent-elles résoudre le problème de la faible efficacité énergétique des bâtiments ?
- Co-fondateur d'OpenAI interviewé par Huang Renxun : les capacités de raisonnement de GPT-4 n'ont pas encore atteint les attentes
- Bing de Microsoft surpasse Google en termes de trafic de recherche grâce à la technologie OpenAI

