Maison >Périphériques technologiques >IA >Les performances de SOTA, la méthode d'IA de prédiction d'affinité protéine-ligand multimodale de Xiamen, combinent pour la première fois des informations sur la surface moléculaire
Les performances de SOTA, la méthode d'IA de prédiction d'affinité protéine-ligand multimodale de Xiamen, combinent pour la première fois des informations sur la surface moléculaire
- WBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBoriginal
- 2024-07-17 18:37:101337parcourir

Éditeur | KX
Dans le domaine de la recherche et du développement de médicaments, prédire avec précision et efficacité l'affinité de liaison des protéines et des ligands est crucial pour le criblage et l'optimisation des médicaments. Cependant, les études actuelles ne prennent pas en compte le rôle important des informations sur la surface moléculaire dans les interactions protéine-ligand.
Sur cette base, des chercheurs de l'Université de Xiamen ont proposé un nouveau cadre d'extraction de caractéristiques multimodales (MFE), qui combine pour la première fois des informations sur la surface des protéines, la structure et la séquence 3D, et utilise un mécanisme d'attention croisée pour différents modes. Alignement des fonctionnalités entre les états.
Les résultats expérimentaux montrent que cette méthode atteint des performances de pointe dans la prédiction de l'affinité de liaison protéine-ligand. De plus, les études d’ablation démontrent l’efficacité et la nécessité des informations sur la surface des protéines et de l’alignement des caractéristiques multimodales dans ce cadre.
Une recherche connexe intitulée « Prédiction de l'affinité de liaison protéine-ligand multimodale basée sur la surface » a été publiée sur « Bioinformatics » le 21 juin.

Recherche sur la prédiction de l'affinité de liaison protéine-ligand
En tant qu'étape clé de la découverte de médicaments, la prédiction de l'affinité de liaison protéine-ligand a été étudiée de manière approfondie depuis longtemps, ce qui est crucial pour un dépistage efficace et précis des médicaments.
Les outils traditionnels de découverte de médicaments assistés par ordinateur utilisent des fonctions de notation (SF) pour estimer approximativement l'affinité de liaison protéine-ligand, mais avec une faible précision. Les méthodes de simulation de dynamique moléculaire peuvent fournir des estimations d’affinité de liaison plus précises, mais sont souvent coûteuses et longues.
Avec le développement de la technologie informatique et l’abondance croissante de données biologiques à grande échelle, les méthodes basées sur l’apprentissage profond ont montré un grand potentiel dans le domaine de la prédiction de l’affinité de liaison protéine-ligand.
Cependant, les recherches actuelles utilisent principalement des représentations basées sur des séquences ou des structures pour prédire l'affinité de liaison protéine-ligand, et il existe relativement peu d'études sur les informations de surface des protéines qui sont cruciales pour les interactions protéine-ligand.
Une surface moléculaire est une représentation de haut niveau de la structure d'une protéine, qui présente des motifs chimiques et géométriques qui servent d'empreintes digitales des modèles d'interaction de la protéine avec d'autres biomolécules. Par conséquent, certaines études ont commencé à utiliser les informations sur la surface des protéines pour prédire l’affinité de liaison protéine-ligand.
Mais les méthodes existantes se concentrent principalement sur les données monomodales et ignorent les informations multimodales des protéines. De plus, lors du traitement de l'information multimodale des protéines, les méthodes traditionnelles connectent généralement les caractéristiques de différentes modalités de manière directe sans tenir compte de l'hétérogénéité entre elles, ce qui entraîne l'incapacité d'exploiter efficacement la complémentarité entre les modalités.
Nouveau cadre d'extraction de caractéristiques multimodales
Ici, les chercheurs proposent un nouveau cadre d'extraction de caractéristiques multimodales (MFE) qui combine pour la première fois des informations provenant de la surface des protéines, de la structure 3D et de la séquence.
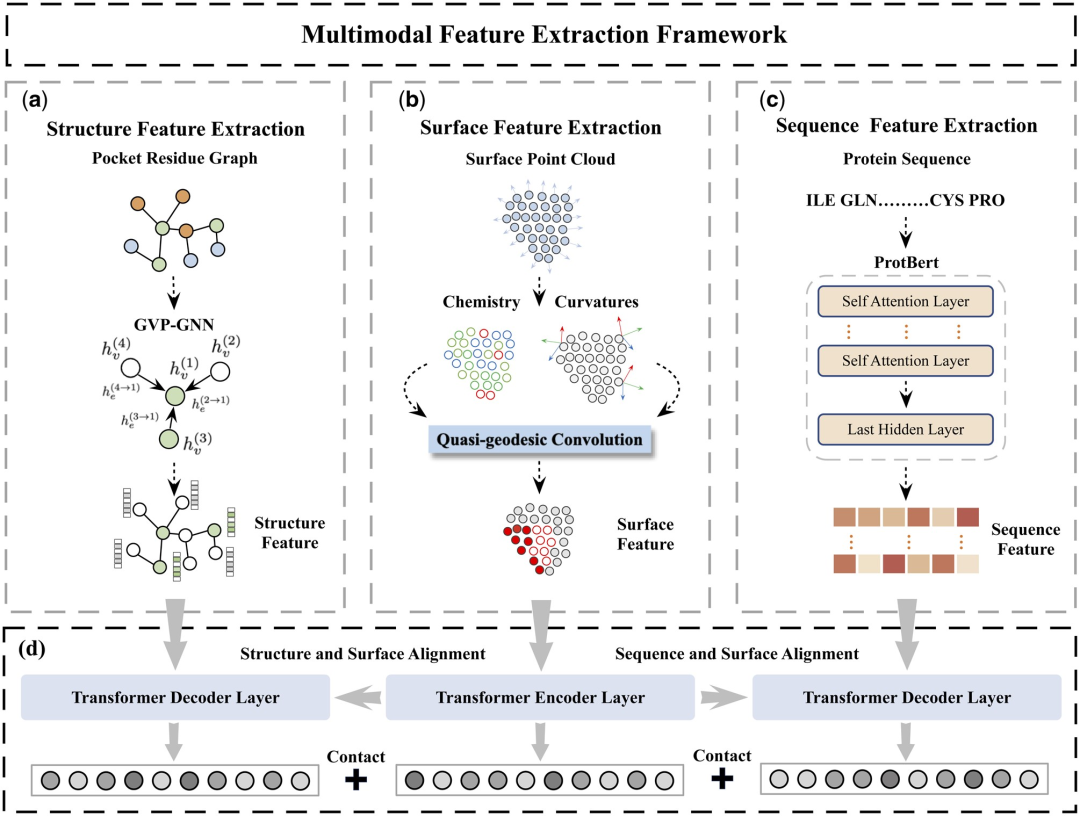
Plus précisément, l'étude a conçu deux composants principaux : le module d'extraction de caractéristiques protéiques et le module de comparaison de caractéristiques multimodales.
Le module d'extraction de caractéristiques des protéines est utilisé pour extraire les incorporations initiales à partir des informations sur la surface, la structure et la séquence des protéines.
Dans le module de comparaison de fonctionnalités multimodales, le mécanisme d'attention croisée est utilisé pour réaliser une comparaison de fonctionnalités entre la structure protéique, l'intégration de séquences et l'intégration de surface afin d'obtenir une intégration de fonctionnalités unifiée et riche en informations.
Comparé aux méthodes de pointe actuelles, le cadre proposé permet d'obtenir les meilleurs résultats dans la tâche de prédiction de l'affinité de liaison protéine-ligand.
Performance SOTA
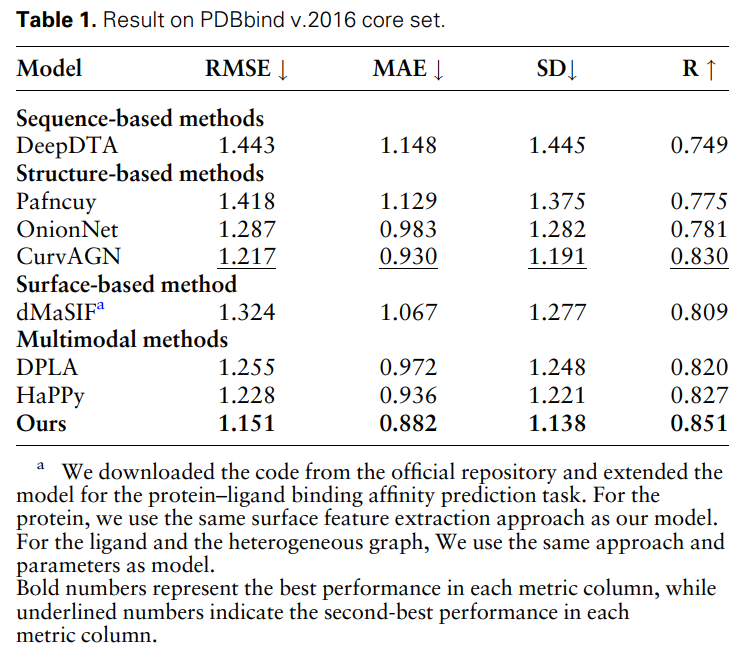
Le Tableau 1 montre les résultats du MFE et d'autres modèles de base sur la tâche de prédiction de l'affinité de liaison protéine-ligand. Tous les modèles ont utilisé la même méthode de partitionnement des ensembles de formation et de validation et ont été testés sur l'ensemble de base PDBbind (version 2016). On peut constater que la méthode MFE atteint des performances SOTA par rapport à toutes les références.
Étude d'ablation
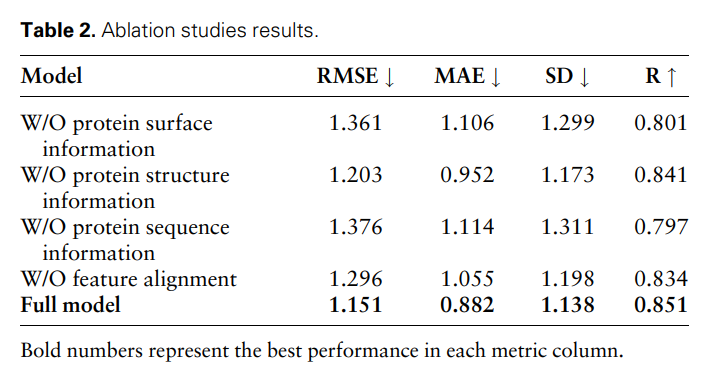
Pour prouver davantage l'efficacité et la nécessité de différentes caractéristiques modales et comparaisons de caractéristiques, les chercheurs ont mené les études d'ablation suivantes : sans informations sur la surface des protéines, sans informations sur la structure des protéines, sans o informations sur les séquences protéiques et alignements sans particularités. Les résultats sont présentés dans le tableau 2 et la figure 2.
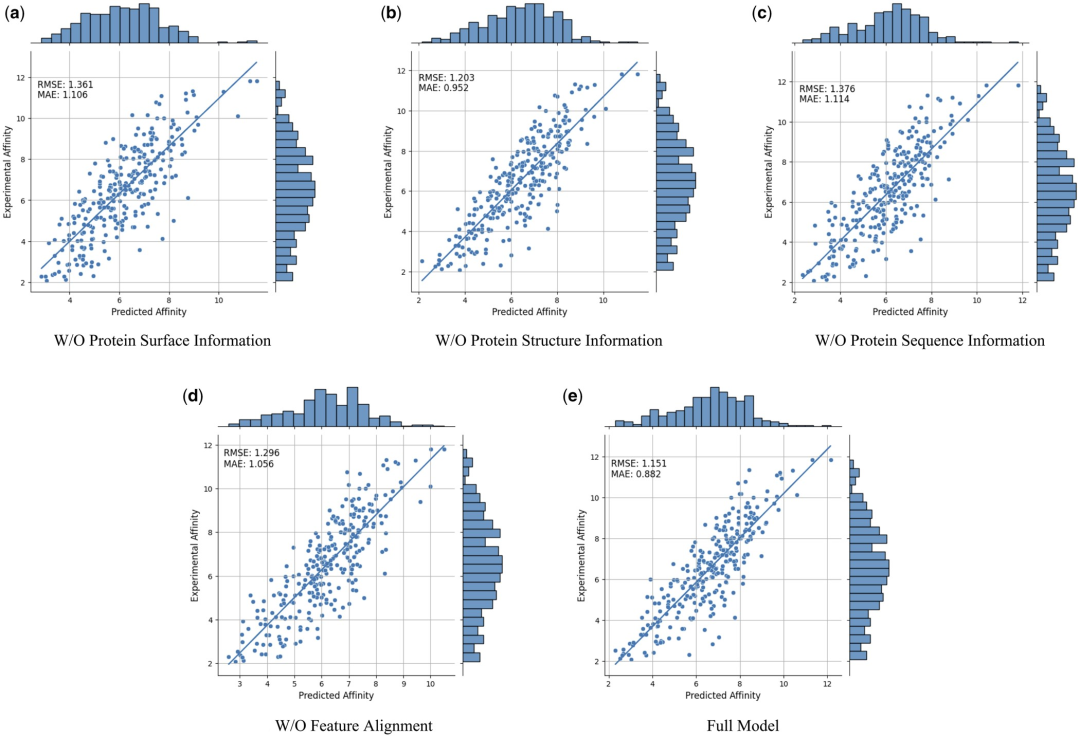
Figure 2 : Résultats de l’étude d’ablation. (Source : article)
Les résultats montrent que lorsque les informations de surface sont supprimées, les performances chutent considérablement, indiquant que les informations de surface jouent un rôle crucial dans le modèle. De même, l'exclusion des informations structurelles ou de séquence entraîne une dégradation des performances, tandis que l'élimination des informations de séquence entraîne une dégradation plus prononcée. En effet, les informations de séquence contiennent des informations globales sur la protéine, ce qui est crucial pour que le modèle comprenne pleinement la protéine.
De plus, sans comparaison des fonctionnalités, les performances du modèle diminueront. Cela souligne l'importance de la comparaison des caractéristiques dans le traitement des données multimodales, car elle contribue à réduire l'hétérogénéité entre les différentes caractéristiques modales, améliorant ainsi la capacité du modèle à intégrer efficacement différentes caractéristiques modales.
Analyse des hyperparamètres
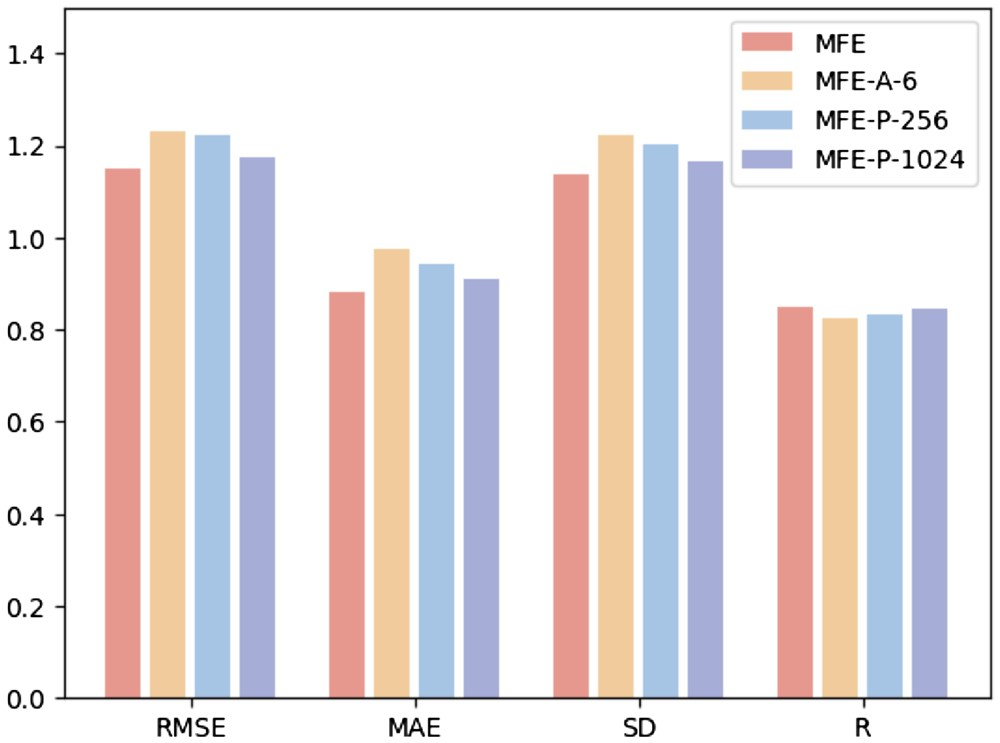
Afin d'étudier l'impact de différents hyperparamètres sur les performances du modèle, les chercheurs ont mené les trois expériences suivantes : (i) MFE-A-6 : n'utilisez que 6 types d'atomes de base pour représenter les produits chimiques. propriétés de la surface, notamment l'hydrogène, le carbone, l'azote, l'oxygène, le phosphore et le soufre ; (ii) MFE-P-256 : seuls les 256 points de surface les plus proches du centre du ligand sont sélectionnés comme surface de la poche protéique ; -P -1024 : Sélectionnez les 1024 points de surface les plus proches du centre du ligand comme surface de la poche protéique.
La figure 3 montre les résultats de trois méthodes différentes de sélection d'hyperparamètres sur la tâche de prédiction d'affinité de liaison protéine-ligand.
Analyse et visualisation de l'alignement des caractéristiques
Afin d'étudier en profondeur l'impact de l'alignement des caractéristiques sur les performances du modèle, les chercheurs ont utilisé l'analyse en composantes principales (ACP) pour effectuer une réduction de dimensionnalité et une sommation de la surface des protéines, de la structure et séquencer les fonctionnalités dans l'ensemble de test Analyse visuelle. Cette approche vise à déterminer si l'alignement des fonctionnalités peut atténuer l'hétérogénéité entre les intégrations multimodales.
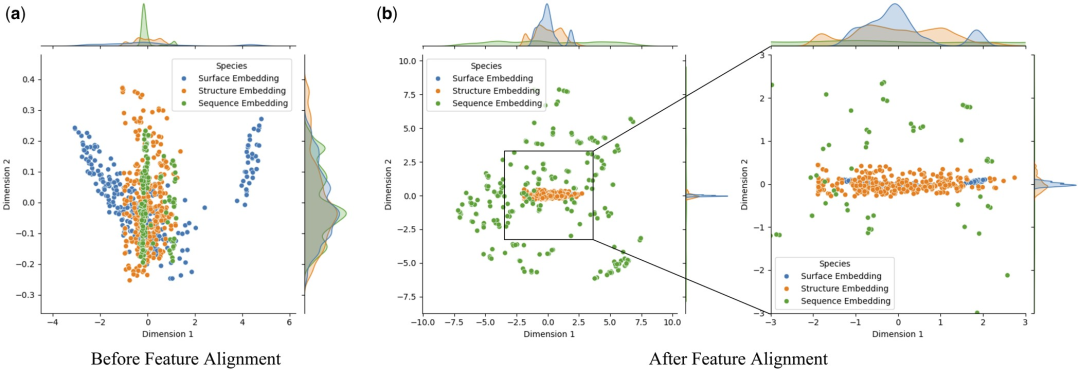
La recherche a révélé que l'alignement des caractéristiques améliorait considérablement la cohérence entre la surface, la structure et l'intégration des séquences des protéines. Cela est dû à l'optimisation des interactions de fonctionnalités multimodales dans Transformer via le mécanisme d'attention, qui calcule les pondérations d'attention entre différentes fonctionnalités. Cela améliore la capacité du modèle à capturer des informations clés, permettant aux données de différentes modalités d'être plus étroitement regroupées dans l'espace des fonctionnalités, réduisant ainsi le bruit et les erreurs dans l'identification par le modèle des interactions protéine-ligand.
Enfin, les chercheurs ont conclu : « En résumé, en étudiant la surface des protéines, nous pouvons mieux comprendre comment les protéines interagissent avec d’autres biomolécules. Dans des travaux futurs, nous explorerons plus en profondeur les surfaces des protéines pour révéler leur application plus large. bioinformatique"
Remarque : la couverture provient d'Internet
.Ce qui précède est le contenu détaillé de. pour plus d'informations, suivez d'autres articles connexes sur le site Web de PHP en chinois!
Articles Liés
Voir plus- Tendances technologiques à surveiller en 2023
- Comment l'intelligence artificielle apporte un nouveau travail quotidien aux équipes des centres de données
- L'intelligence artificielle ou l'automatisation peuvent-elles résoudre le problème de la faible efficacité énergétique des bâtiments ?
- Co-fondateur d'OpenAI interviewé par Huang Renxun : les capacités de raisonnement de GPT-4 n'ont pas encore atteint les attentes
- Bing de Microsoft surpasse Google en termes de trafic de recherche grâce à la technologie OpenAI

