Maison >Périphériques technologiques >IA >La molécule est efficace à 100 %, les ligands sont conçus à partir de zéro et l'Université du Hunan propose un cadre de caractérisation moléculaire basé sur des fragments
La molécule est efficace à 100 %, les ligands sont conçus à partir de zéro et l'Université du Hunan propose un cadre de caractérisation moléculaire basé sur des fragments
- PHPzoriginal
- 2024-07-11 16:12:59621parcourir
Applications et défis des descripteurs moléculaires
Les descripteurs moléculaires sont largement utilisés en modélisation moléculaire. Cependant, dans le domaine de la découverte moléculaire assistée par l’IA, il existe un manque de représentations moléculaires naturellement applicables, complètes et originales, ce qui affecte les performances et l’interprétabilité des modèles.
Proposition du cadre t-SMILES
Le cadre de caractérisation moléculaire multi-échelle basé sur des fragments t-SMILES résout le problème de la caractérisation moléculaire. Le framework utilise des chaînes de type SMILES pour décrire les molécules et prend en charge les modèles de séquence en tant que modèles génératifs. Les algorithmes de code de
t-SMILES
t-SMILES dispose de trois algorithmes de code : TSSA, TSDY et TSID.
Résultats expérimentaux
Les expériences montrent que les molécules générées par le modèle t-SMILES ont une validité théorique de 100 % et une nouveauté élevée, ce qui est meilleur que le modèle basé sur SOTA SMILES.
De plus, le modèle t-SMILES évite le surajustement et maintient la similarité sur les ensembles de données étiquetés à faibles ressources tout en obtenant une nouveauté plus élevée.
Informations publiées
L'étude, intitulée « t-SMILES : un cadre de représentation moléculaire basé sur des fragments pour la conception de ligands de novo », a été publiée dans « Nature Communications » le 11 juin.

Recherche sur la méthode de représentation moléculaire basée sur SMILES
La caractérisation efficace des molécules est un facteur clé affectant les performances des modèles de renseignement.
Les réseaux de neurones graphiques (GNN) sont populaires pour leur capacité à générer des molécules efficaces à 100 %, mais leurs capacités d'expression sont limitées.
Spécification d'entrée linéaire moléculaire simplifiée (SMILES), en tant que représentation linéaire, est susceptible de produire des chaînes chimiquement invalides. DeepSMILES et SELFIES sont des améliorations alternatives, mais présentent toujours des problèmes.
De plus, la recherche montre que les modèles de langage (LM) peuvent surpasser la plupart des GNN dans l'apprentissage de molécules volumineuses et complexes. Récemment, les LM basés sur Transformers ont démontré leur capacité à générer du texte qui ressemble beaucoup à l'écriture humaine.
Inspirés par ces idées, les chercheurs ont choisi SMILES comme choix de départ pour la description des fragments et l'ont combiné à une technologie avancée de traitement du langage naturel pour gérer des tâches de modélisation moléculaire basées sur des fragments, qui peuvent fusionner le modèle graphique pour accorder plus d'attention à la topologie moléculaire et LM L'avantage d'une forte capacité d'apprentissage.
Générer de nouvelles molécules 100% efficaces, meilleures que SOTA
Par conséquent, l'équipe de l'Université du Hunan a proposé un nouveau cadre de description moléculaire t-SMILES (tree-based SMILES) basé sur des molécules fragmentées. Le framework contient trois algorithmes de codage t-SMILES : TSSA (t-SMILES avec atomes partagés), TSDY (t-SMILES avec atomes virtuels mais pas d'identifiants) et TSID (t-SMILES avec identifiants et atomes virtuels).

Le nouveau framework t-SMILES
- génère des arbres moléculaires acycliques (AMT), représentant des molécules fragmentées.
- Convertissez AMT en arbre binaire complet (FBT).
- Effectuez un parcours en largeur sur FBT pour obtenir la chaîne t-SMILES.
Par rapport à SMILES
t-SMILES n'introduit que deux nouveaux symboles "&" et "^" pour coder une topologie moléculaire multi-échelle et hiérarchique. L'algorithme
t-SMILES
fournit un cadre évolutif et adaptable qui peut théoriquement prendre en charge un large éventail de schémas de sous-structure.
Le modèle basé sur t-SMILES
est capable d'apprendre des informations de structure topologique de haut niveau tout en traitant des informations détaillées sur la sous-structure.
Système multi-codes
L'algorithme t-SMILES peut construire un système multi-codes pour la description moléculaire, où :
- Les SMILES classiques peuvent être intégrés comme un cas particulier de t-SMILES (TS_Vanilla).
- Plusieurs descriptions peuvent collaborer pour améliorer les performances globales.
Illustration : distribution de tokens pour le code TSSA, SMILES et SELFIES. (Source : article)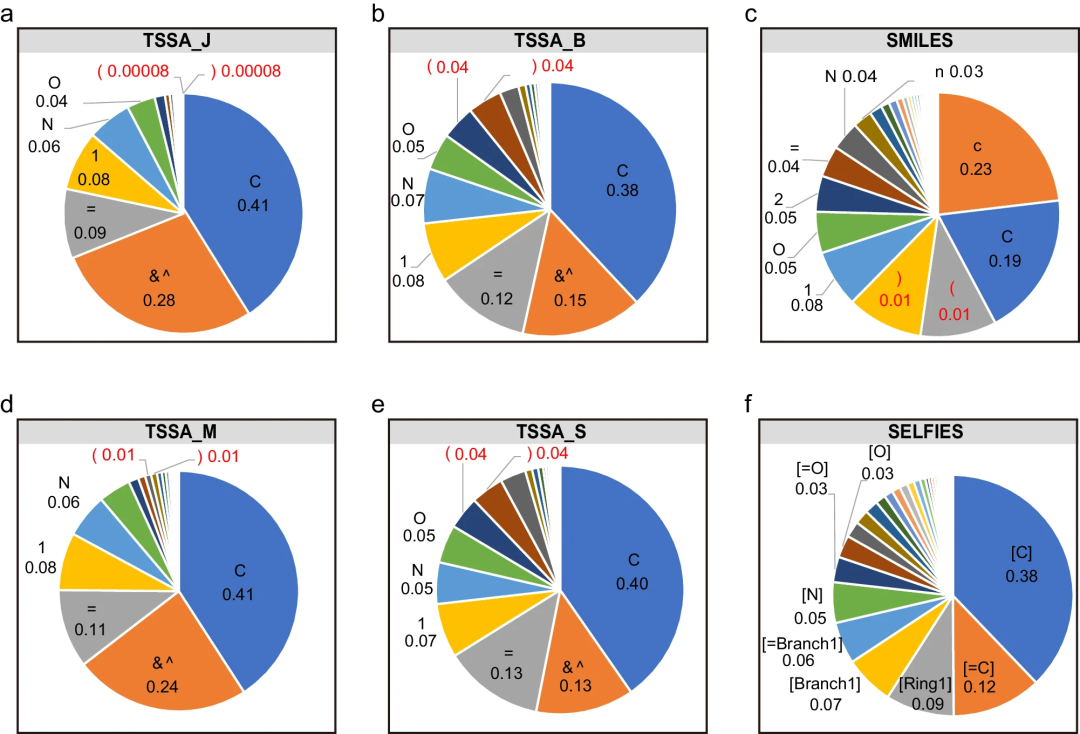
Tout d’abord, les chercheurs ont systématiquement évalué t-SMILES en approfondissant ses caractéristiques uniques. Par la suite, des expériences ont été menées à l'aide de TSSA et TSDY sur deux ensembles de données étiquetés à faibles ressources, JNK332 et AID170633.
La recherche se concentre sur les limites de t-SMILES et de ses alternatives, qui sont obtenues en tirant parti de l'augmentation standard des données et de modèles affinés pré-entraînés. Vingt tâches ciblées sur ChEMBL ont été évaluées en parallèle à l'aide de TSDY, TSSA et TSID. Des expériences approfondies ont également été réalisées sur ChEMBL, Zinc et QM9 pour comparer t-SMILES et ses alternatives en utilisant des configurations similaires. En outre, divers modèles de base basés sur des fragments et modèles SOTA GNN sont comparés.
Enfin, une étude d'ablation est réalisée pour confirmer l'efficacité du modèle génératif basé sur SMILES avec reconstruction. Pour évaluer l'adaptabilité et la flexibilité de l'algorithme t-SMILES, quatre algorithmes de fragmentation précédemment publiés ont été utilisés pour décomposer les molécules, notamment JTVAE, BRICS, MMPA et Scaffold. Trois métriques ont été utilisées dans différentes expériences : un benchmark d'apprentissage distribué, un benchmark orienté vers un objectif et la métrique de distance de Wasserstein pour les propriétés physicochimiques.
Des expériences comparatives détaillées montrent que les nouvelles molécules générées par le modèle t-SMILES sont 100% théoriquement valides et meilleures que le modèle basé sur SOTA SMILES. Par rapport à SMILES, DSMILES et SELFIES, la solution globale de t-SMILES peut éviter les problèmes de surajustement et améliorer considérablement les performances équilibrées sur des ensembles de données à faibles ressources, que ce soit en utilisant l'augmentation des données ou un modèle pré-entraîné puis affiné.
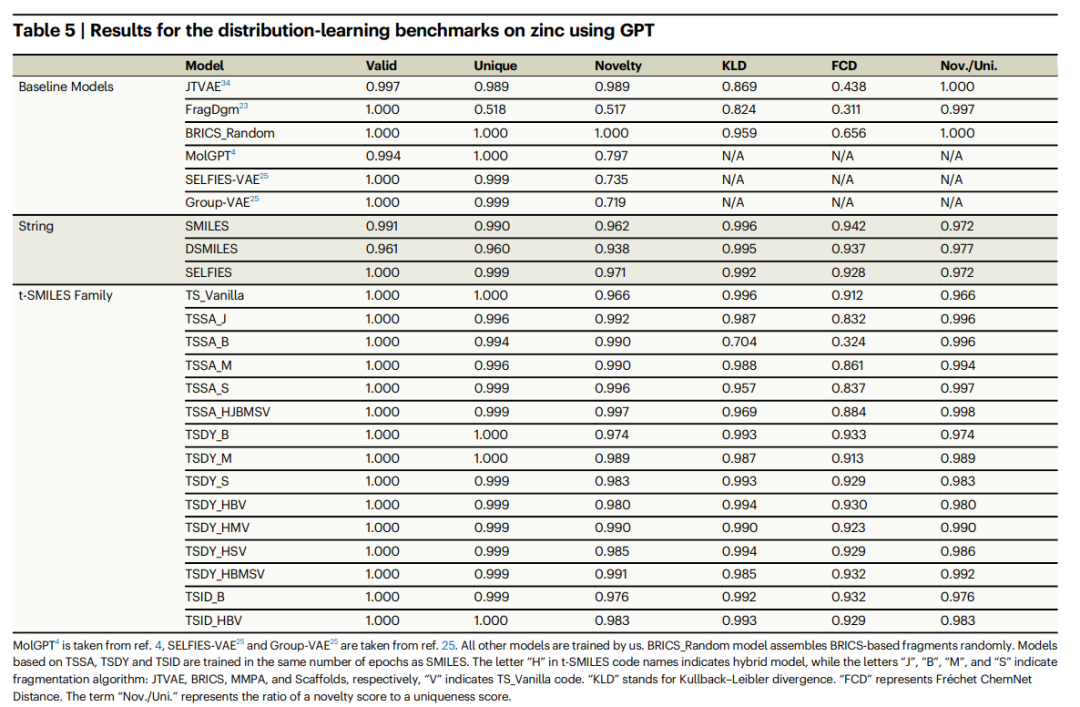
De plus, le modèle t-SMILES est capable de capturer habilement les propriétés physicochimiques des molécules, garantissant que les molécules générées conservent une similarité avec la distribution des molécules d'entraînement. Cela améliore considérablement les performances par rapport aux modèles de base existants basés sur des fragments et des graphiques. En particulier, le modèle t-SMILES avec algorithme de reconstruction orienté objectif présente des avantages évidents par rapport à SMILES, DSMILES, SELFIES et SOTA CReM dans les tâches orientées objectif.
Limitations et marge d'amélioration
- LLM peut comprendre une grammaire anglaise bien formatée. Par conséquent, il reste à explorer en profondeur si la structure arborescente de t-SMILES peut être apprise et comment LM peut aller au-delà des corrélations statistiques des surfaces pour acquérir des connaissances chimiques sur les molécules.
- Cette recherche se concentre sur le codage de molécules fragmentées en séquences, de sorte que seuls les algorithmes de fragmentation publiés sont utilisés comme exemples pour créer des « mots chimiques ». Les recherches futures pourront exploiter t-SMILES pour explorer d’autres algorithmes de fragmentation afin d’interpréter plus en profondeur les phrases et leurs significations chimiques, ce qui est en réalité plus difficile que la PNL.
- Bien que t-SMILES ait été conçu pour améliorer les performances des descriptions moléculaires et contourner les limites de SMILES, l'étude n'a pas expérimenté de molécules plus complexes. Cela fera l’objet de recherches futures.
- Enfin, c'est un début prometteur pour coder des molécules fragmentées dans des chaînes de type SMILES. Des recherches plus approfondies pourraient explorer des algorithmes avancés pour la reconstruction et l’optimisation moléculaires, des modèles génératifs améliorés et des techniques évolutives. De plus, la recherche peut se concentrer sur les tâches de propriété, de rétrosynthèse et de prédiction des réactions.
Remarque : La couverture provient d'Internet
Ce qui précède est le contenu détaillé de. pour plus d'informations, suivez d'autres articles connexes sur le site Web de PHP en chinois!
Articles Liés
Voir plus- Tendances technologiques à surveiller en 2023
- Comment l'intelligence artificielle apporte un nouveau travail quotidien aux équipes des centres de données
- L'intelligence artificielle ou l'automatisation peuvent-elles résoudre le problème de la faible efficacité énergétique des bâtiments ?
- Co-fondateur d'OpenAI interviewé par Huang Renxun : les capacités de raisonnement de GPT-4 n'ont pas encore atteint les attentes
- Bing de Microsoft surpasse Google en termes de trafic de recherche grâce à la technologie OpenAI

