Maison >Périphériques technologiques >IA >Sous-journal Nature, avec un taux de précision de 96 %, l'IA prédit les interactions protéine-ligand à partir de séquences
Sous-journal Nature, avec un taux de précision de 96 %, l'IA prédit les interactions protéine-ligand à partir de séquences
- PHPzoriginal
- 2024-07-11 12:56:20795parcourir
Dans le développement de médicaments, il est crucial de déterminer l'affinité de liaison et l'effet fonctionnel des ligands de petites molécules sur les protéines. Les méthodes informatiques actuelles peuvent prédire ces propriétés d’interaction protéine-ligand, mais sans structures protéiques à haute résolution, la précision est souvent perdue et les effets fonctionnels ne peuvent être prédits.
Des chercheurs de l'Université Monash et de l'Université Griffith ont développé PSICHIC (PhySIcoCHhemICal Graph Neural Network), un cadre qui combine des contraintes physicochimiques directement à partir de séquences de données décodant les empreintes digitales d'interaction. Cela permet à PSICHIC de décoder les mécanismes à l’origine des interactions protéine-ligand, atteignant ainsi une précision et une interprétabilité de pointe.
Formé sur les mêmes paires protéine-ligand sans données structurelles, PSICHIC a obtenu des résultats comparables, voire dépassés, aux principales méthodes basées sur la structure dans les prédictions d'affinité de liaison. L'empreinte interprétable de
PSICHIC identifie les résidus protéiques et les atomes de ligand impliqués dans l'interaction et aide à révéler les déterminants de la sélectivité des interactions protéine-ligand.
L'étude s'intitulait « Réseau neuronal à graphes physicochimiques pour l'apprentissage des empreintes digitales d'interaction protéine-ligand à partir de données de séquence » et a été publiée dans « Nature Machine Intelligence » le 17 juin 2024.

Dans le processus de découverte de médicaments, comprendre l'affinité de liaison et les effets fonctionnels des ligands à petites molécules sur les protéines est essentiel, car l'interaction sélective du ligand avec une protéine spécifique détermine l'effet attendu. du médicament.
Cependant, bien que les méthodes informatiques actuelles soient capables de prédire les propriétés d'interaction protéine-ligand, en l'absence de structures protéiques à haute résolution, la précision de la prédiction diminue souvent et il existe également des difficultés à prédire les effets fonctionnels.
Bien que les méthodes basées sur les séquences présentent plus d'avantages en termes de coût et de ressources (par exemple, elles ne nécessitent pas de processus coûteux de détermination de structure expérimentale), ces méthodes sont souvent confrontées au problème de degrés de liberté excessifs dans la correspondance de modèles, ce qui peut facilement conduire à un surajustement. et des capacités de généralisation limitées, créant ainsi un écart de performance par rapport aux méthodes basées sur la structure ou les composites.
Physical Chemistry Graph Neural Network
Une équipe de recherche de l'Université Monash et de l'Université Griffith a développé PSICHIC (Physical Chemistry Graph Neural Network), une méthode pour décoder directement les protéines-ligands à partir de données de séquence selon les principes physiques et chimiques des empreintes digitales d'interaction corporelle. méthode. Contrairement aux modèles précédents basés sur des séquences, PSICHIC intègre de manière unique des contraintes physico-chimiques pour atteindre une précision et une interprétabilité de pointe.
En tant que méthode basée sur des séquences 2D, PSICHIC génère et impose ces contraintes sur un tracé 2D en appliquant un algorithme de clustering, permettant à PSICHIC de s'adapter principalement aux modèles rationnels sous-jacents qui déterminent les interactions protéine-ligand pendant l'entraînement.

(Source : article)
Validation et comparaison des performances
Après un entraînement sur les mêmes paires protéine-ligand sans données structurelles, PSICHIC surpasse dans les prédictions d'affinité de liaison. -Les méthodes basées sur la structure artistique et les méthodes composites les rivalisent, voire les surpassent.
Les résultats expérimentaux sur les ensembles de données PDBBind v2016 et PDBBind v2020 montrent que PSICHIC surpasse les autres méthodes basées sur des séquences, telles que TransCPI, MolTrans et DrugBAN, sur plusieurs indicateurs.
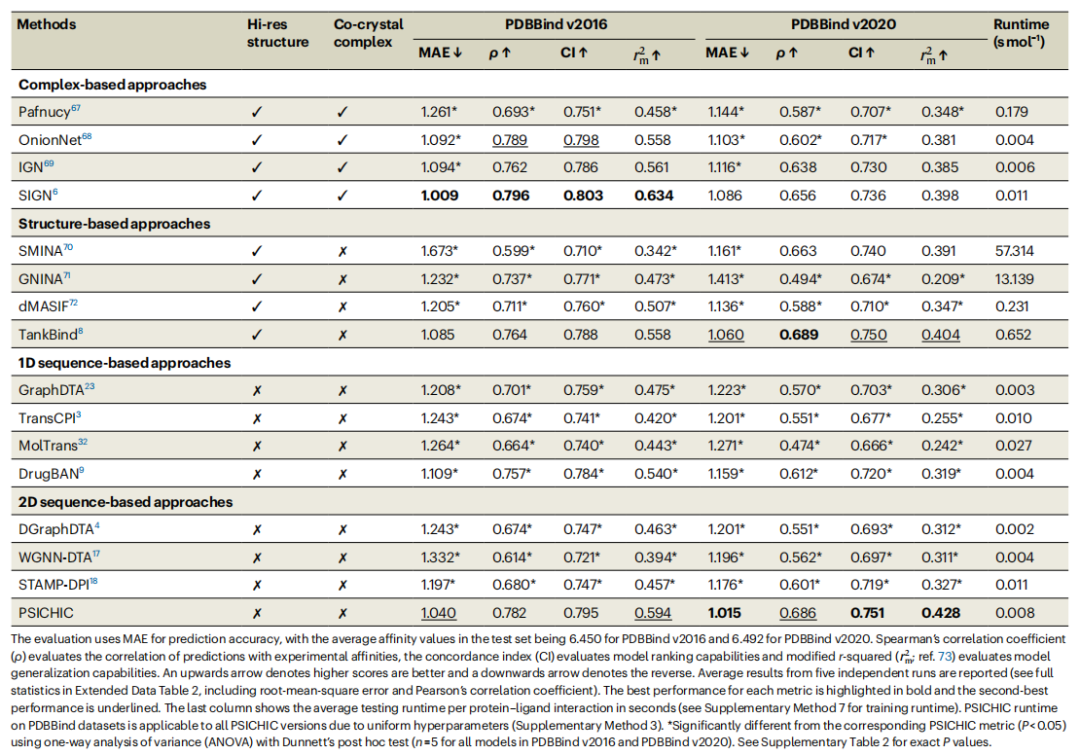
Résumé statistique des performances des prédictions d'affinité de liaison protéine-ligand sur les benchmarks PDBBind v2016 et PDBBind v2020. (Source : article)
Plus précisément :
- PSICHIC montre une erreur de prédiction plus faible et un indice de corrélation plus élevé, notamment en termes de précision de prédiction et de capacité de généralisation.
- PSICHIC atteint une précision allant jusqu'à 96 % dans la prédiction des effets fonctionnels.
Aussi :
- PSICHIC excelle dans l’identification des sites de liaison et des groupes fonctionnels clés du ligand.
- Dans l'analyse de plusieurs structures complexes protéine-ligand (telles que PDB 6K1S et 6OXV), PSICHIC a pu localiser avec précision des résidus de liaison importants et des groupes fonctionnels de ligands, vérifiant sa capacité de décodage direct des protéines-ligands dans les données de séquence. modèles d’interaction corporelle.
- Cette capacité se reflète particulièrement dans sa capacité à prédire les sites de liaison protéine-ligand et les résidus clés à partir des données de séquence.
Illustration : Dépistage virtuel par empreintes digitales interactives. (Source : article)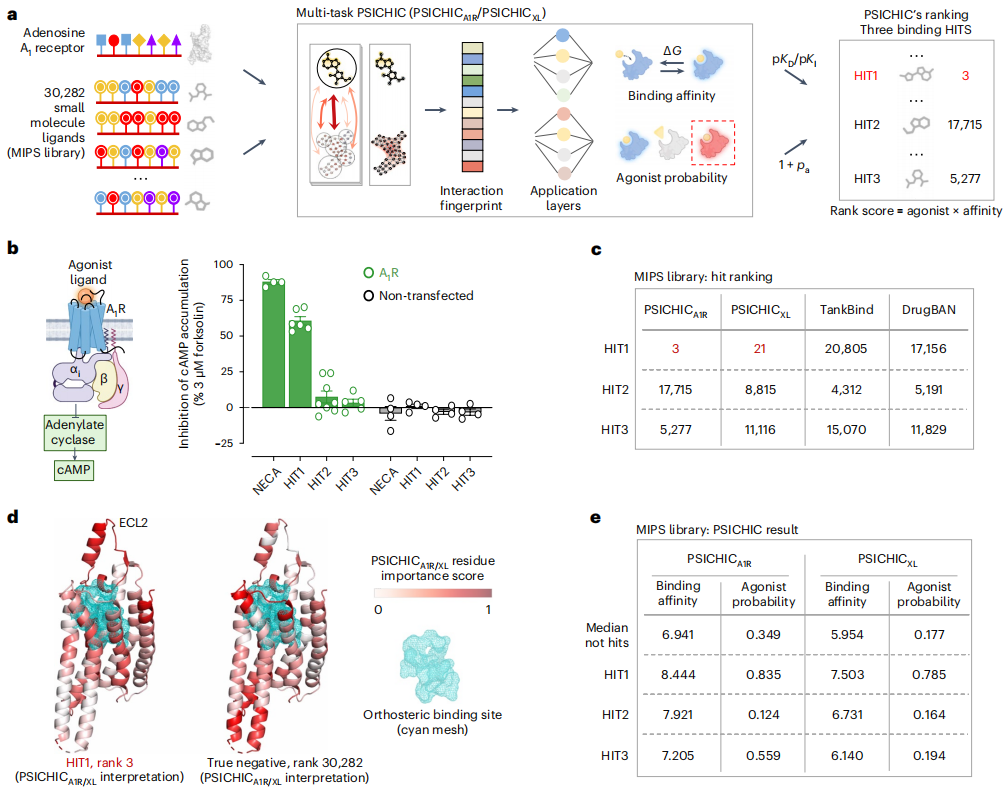
Fait intéressant, les empreintes digitales interprétables de PSICHIC montrent qu'il obtient la capacité de décoder le mécanisme sous-jacent des interactions protéine-ligand à partir des seules données de séquence, en identifiant les résidus protéiques des sites de liaison et les capacités atomiques des ligands impliqués, même lorsqu'ils sont formés uniquement sur données de séquence avec des étiquettes d’affinité de liaison et aucune information d’interaction.
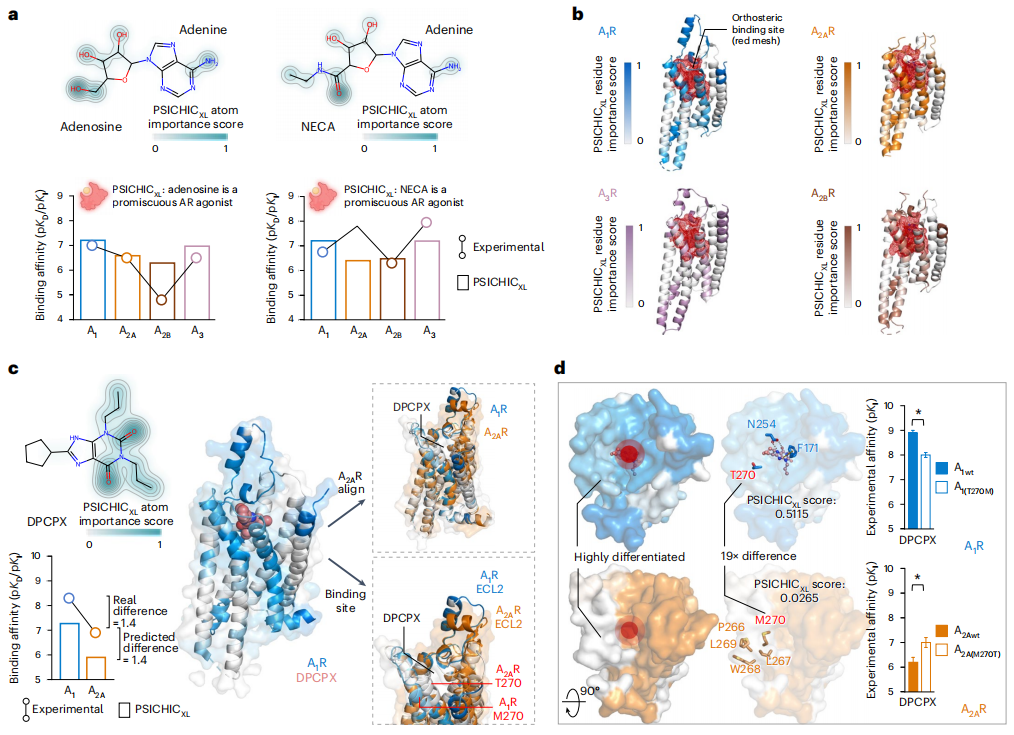
- L'empreinte digitale de l'interaction protéine-ligand décrit les caractéristiques d'interaction spécifiques entre les ligands et les résidus protéiques.
- PSICHIC propose une approche unique pour obtenir des empreintes digitales d'interaction interprétables en utilisant uniquement des données de séquence.
- PSICHIC intègre des contraintes et démontre des capacités émergentes pour révéler les mécanismes d'interaction protéine-ligand et prédire efficacement les propriétés d'interaction.
- PSICHIC élimine le besoin de données 3D, ouvrant la voie à un apprentissage robuste sur des bases de données de séquences à grande échelle.
Perspectives futures
- Étendez l'analyse PSICHIC aux complexes protéiques, tels que les GPCR complexés avec des protéines G hétérotrimériques.
- Explorer des interactions complexes telles que la régulation allostérique permet de comprendre comment les ligands allostériques régulent les ligands orthostériques au sein de cibles protéiques.
- PSICHIC a prouvé sa robustesse et son efficacité dans divers domaines d'application et dispose d'un large potentiel de développement futur.
Ce qui précède est le contenu détaillé de. pour plus d'informations, suivez d'autres articles connexes sur le site Web de PHP en chinois!
Articles Liés
Voir plus- Tendances technologiques à surveiller en 2023
- Comment l'intelligence artificielle apporte un nouveau travail quotidien aux équipes des centres de données
- L'intelligence artificielle ou l'automatisation peuvent-elles résoudre le problème de la faible efficacité énergétique des bâtiments ?
- Co-fondateur d'OpenAI interviewé par Huang Renxun : les capacités de raisonnement de GPT-4 n'ont pas encore atteint les attentes
- Bing de Microsoft surpasse Google en termes de trafic de recherche grâce à la technologie OpenAI

