Maison >Périphériques technologiques >IA >Sous-journal Nature, meilleur qu'AlphaFold, échantillonnage de tous les atomes, une méthode d'IA pour prédire la structure des peptides
Sous-journal Nature, meilleur qu'AlphaFold, échantillonnage de tous les atomes, une méthode d'IA pour prédire la structure des peptides
- WBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBoriginal
- 2024-07-11 11:49:39968parcourir

Les méthodes d'apprentissage en profondeur ont favorisé des progrès significatifs dans la prédiction à un état unique des structures biomoléculaires. Cependant, la fonctionnalité des biomolécules dépend de la gamme de conformations qu’elles peuvent prendre. Cela est particulièrement vrai pour les peptides, une classe de molécules très flexibles qui participent à divers processus biologiques et présentent un grand intérêt en termes thérapeutiques.
Philip M. Kim et Osama Abdin de l'Université de Toronto ont développé PepFlow, un modèle génératif transférable qui permet l'échantillonnage de tous les atomes directement à partir de l'espace conformationnel autorisé d'un peptide d'entrée. Les chercheurs ont formé le modèle dans un cadre de diffusion, puis ont utilisé un flux équivalent pour l'échantillonnage conformationnel.
Pour surmonter le coût prohibitif de la modélisation généralisée de tous les atomes, ils ont modularisé le processus de génération et intégré des super-réseaux pour prédire les paramètres de réseau spécifiques à une séquence. PepFlow prédit avec précision les structures peptidiques et reproduit efficacement les collections expérimentales de peptides en une fraction du temps d'exécution des méthodes traditionnelles. PepFlow peut également être utilisé pour échantillonner des conformations qui satisfont à des contraintes telles que la macrocyclisation.
"Jusqu'à présent, nous n'avons pas été en mesure de simuler la conformation complète d'un peptide." Osama Abdin, le premier auteur de l'étude, a déclaré : "PepFlow utilise l'apprentissage en profondeur pour capturer la conformation précise du peptide en quelques minutes. "Le modèle a le potentiel d'être utilisé par la conception des peptides comme liants pour guider le développement de médicaments"
L'étude était intitulée "Échantillonnage conformationnel direct à partir de paysages énergétiques peptidiques à travers une diffusion conditionnée par hyperréseau" et a été publiée dans "Nature Machine Intelligence" le 27 juin 2024.

- Les interactions protéine-peptide sont répandues dans les voies moléculaires et sont essentielles à la fonction cellulaire.
- Jusqu'à 40 % des interactions protéine-protéine impliquent la liaison peptidique.
- Les peptides agissent en liant de courtes séquences de protéines globulaires à des régions désordonnées.
Potentiel thérapeutique des peptides
- Les peptides ont une spécificité élevée et un faible risque de toxicité.
- Par rapport aux agents biologiques, les peptides ont de faibles coûts de production et une faible immunogénicité.
- La thérapie peptidique continue d’étendre sa part sur le marché pharmaceutique.
Modélisation et ingénierie des peptides
- Philip M. Kim a déclaré que le modèle PepFlow se concentre sur les peptides car ce sont des biomolécules importantes avec des activités naturelles et leurs conformations doivent être simulées pour comprendre leur fonction.
- Philip M. Kim et Osama Abdin proposent une méthode d'échantillonnage direct de tous les atomes des conformations peptidiques.
- Un échantillonnage précis et efficace de tous les atomes est extrêmement difficile, même pour les peptides courts.
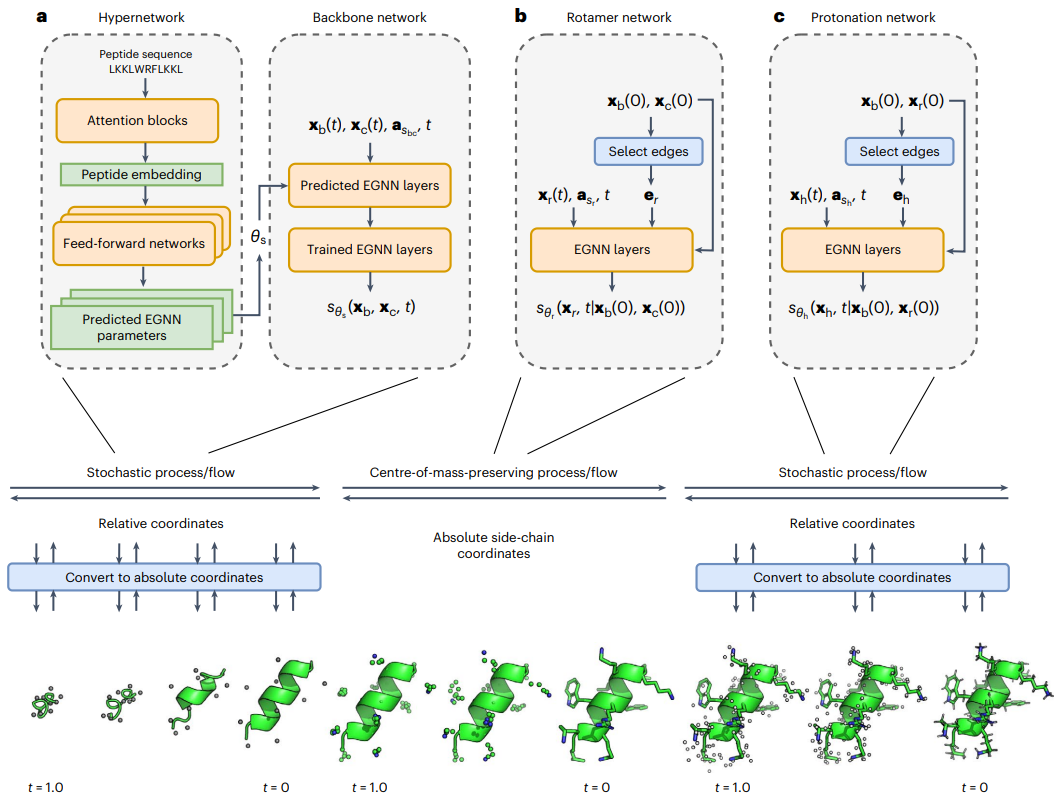
Illustration : Schéma de principe de l'architecture PepFlow. (Source : article)
Pour résoudre ce problème, ils ont développé PepFlow, un modèle génératif conditionnel modulaire en hyperréseau qui peut prédire les conformations de tous les atomes pour n'importe quelle séquence peptidique d'entrée. PepFlow est un modèle de diffusion en temps continu formé sur des conformations moléculaires connues. L'ODE de flux probabiliste correspondant est utilisé pour l'échantillonnage et la formation d'énergie.
PepFlow possède de puissantes capacités pour prédire les structures peptidiques singules et les collections de motifs linéaires courts (SLiM), et peut modéliser les structures peptidiques sous des contraintes telles que la macrocyclisation grâce à des recherches conformationnelles dans l'espace latent.
Ce modèle étend la capacité d'AlphaFold, le principal système d'IA de Google Deepmind, à prédire les structures des protéines. PepFlow surpasse AlphaFold2 en générant une gamme de conformations pour un peptide donné ; AlphaFold2 n'a pas été conçu pour résoudre ce problème ;
Ce qui distingue PepFlow, c'est l'innovation technologique qui se cache derrière. Par exemple, il s’agit d’un modèle généralisé inspiré du générateur Boltzmann, un modèle d’apprentissage automatique très avancé basé sur la physique.
« L'utilisation de la modélisation PepFlow peut donner un aperçu du véritable statut énergétique des peptides. » Abdin a déclaré : « Il a fallu deux ans et demi pour développer PepFlow et seulement un mois pour le former, mais cela vaut la peine de passer à la prochaine frontière et au-delà. Des modèles qui prédisent une seule structure d’un peptide. Dans l’ensemble, la capacité d’échantillonner avec précision et efficacité les conformations des peptides a le potentiel d’améliorer l’amarrage et la conception des peptides. Les méthodes d’amarrage peptidique commencent généralement par une bibliothèque de conformations peptidiques ancrées à la protéine d’intérêt. Une génération plus précise d’ensembles peptidiques pourrait améliorer ce processus.
PepFlow peut également être utilisé pour évaluer la propension de différentes séquences à adopter des conformations aux interfaces protéine-protéine cibles, qui peuvent à leur tour être utilisées pour concevoir des peptides inhibiteurs.
Illustration : Comparaison des ensembles générés par PepFlow et simulations de dynamique moléculaire. (Source : article) 
PepFlow présente un inconvénient important : contrairement au générateur Boltzmann, PepFlow n'a pas la capacité de repondérer les échantillons générés pour obtenir une distribution Boltzmann précise.
Bien que PepFlow soit capable d'effectuer des calculs de vraisemblance sur des échantillons générés, les calculs réalisables nécessitent l'utilisation d'estimateurs stochastiques, qui ajoutent du bruit aux valeurs calculées. De plus, PepFlow génère occasionnellement des échantillons à haute énergie, mais est incapable de capturer l’intégralité du paysage énergétique observé dans les simulations de dynamique moléculaire.
Un moyen potentiel d'améliorer PepFlow consiste à transférer le modèle développé vers d'autres cadres d'échantillonnage. Un flux normalisé a été utilisé dans les paramètres conditionnels et différentes méthodes d'échantillonnage ont été utilisées pour faciliter l'échantillonnage à partir de la distribution de Boltzmann.
Le paradigme d'appariement de flux récemment développé par la communauté universitaire sert en outre d'alternative à la formation de modèles de flux normalisés continus sans simulation. L'appariement de flux a été utilisé efficacement pour l'échantillonnage structurel de différentes molécules, y compris de petites molécules et de protéines, et peut potentiellement être utilisé pour étendre l'efficacité du cadre PepFlow.
En résumé, PepFlow est conçu pour être facilement extensible pour tenir compte d'autres facteurs, de nouvelles informations et d'utilisations potentielles.
Même dans sa première version, PepFlow est un modèle complet et efficace avec un potentiel de développement ultérieur de thérapies reposant sur la liaison peptidique pour activer ou inhiber des processus biologiques.
Lien papier : https://www.nature.com/articles/s42256-024-00860-4
Rapports associés : https://phys.org/news/2024-06-deep-outperforms-google-ai -peptide.html
Ce qui précède est le contenu détaillé de. pour plus d'informations, suivez d'autres articles connexes sur le site Web de PHP en chinois!
Articles Liés
Voir plus- Tendances technologiques à surveiller en 2023
- Comment l'intelligence artificielle apporte un nouveau travail quotidien aux équipes des centres de données
- L'intelligence artificielle ou l'automatisation peuvent-elles résoudre le problème de la faible efficacité énergétique des bâtiments ?
- Co-fondateur d'OpenAI interviewé par Huang Renxun : les capacités de raisonnement de GPT-4 n'ont pas encore atteint les attentes
- Bing de Microsoft surpasse Google en termes de trafic de recherche grâce à la technologie OpenAI