Home >Technology peripherals >AI >A team from the Institute of Computing Technology of the Chinese Academy of Sciences proposed CarbonNovo, an AI-based end-to-end de novo design of protein structures and sequences.
A team from the Institute of Computing Technology of the Chinese Academy of Sciences proposed CarbonNovo, an AI-based end-to-end de novo design of protein structures and sequences.
- 王林Original
- 2024-08-21 21:09:32868browse

编辑 | ScienceAI
近期,中国科学院计算所张海仓带领的研究团队提出了 CarbonNovo,以端到端的方式联合设计蛋白质主链结构和序列。
该研究以「CarbonNovo: Joint Design of Protein Structure and Sequence Using a Unified Energy-based Model」为题发表在机器学习会议 ICML 2024 上。

背景介绍
蛋白质是生物执行功能的重要大分子。蛋白质从头设计旨在创造全新的蛋白质,在药物开发和酶工程中有着广泛的应用。
近几年,基于 AI 的蛋白质从头设计快速发展,已被成功应用于抗体设计、小蛋白药物设计等领域,和传统设计方法相比,其显着提高了设计成功率和效率。
AI 蛋白质设计得益于近几年的两大技术突破:
一是蛋白质结构预测领域的AlphaFold2 模型,它为蛋白质计算领域包括蛋白质设计领域,提供了基础的神经网络模型架构、蛋白质序列表示和结构表示方法、以及先进的训练策略(蒸馏训练、端到端训练)等技术;
二是AIGC 在文字、图像、视频生成领域的快速发展,为蛋白质设计提供了成熟的生成模型,例如DDPM, SDE, Flow Matching,Bayesian Flow Network 等。代表性的蛋白质设计模型,例如 RFDiffusion 和 Chroma 等,主要思路都是将这两大技术融合,将蛋白质的序列和结构表示网络嵌入到基于 AI 的生成模型框架中。
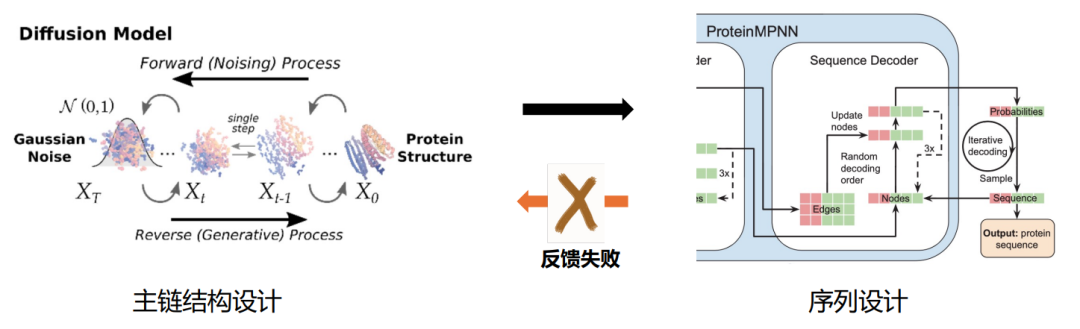
蛋白质从头设计主要包含两个步骤,蛋白质主链结构设计和序列设计(图1)。相应地,当前的主流模型通常采用「两阶段」框架进行:在训练过程中,结构设计模块和序列设计模块分别训练;在推断过程中,首先生成主链结构,然后为主链结构生成最优序列。代表性地,领域内使用 RFDiffusion 和 ProteinMPNN 这两个软件,依次生成主链结构和序列。
蛋白质从头设计的「两阶段」框架存在固有的局限性:
CarbonNovo进行端到端的结构和序列联合设计
针对蛋白质设计「两阶段」框架的局限性,中科院计算所张海仓带领的研究团队提出CarbonNovo,以端到端的方式联合设计蛋白质主链结构和序列。论文近期已于近期发表在机器学习会议 ICML 2024 上。
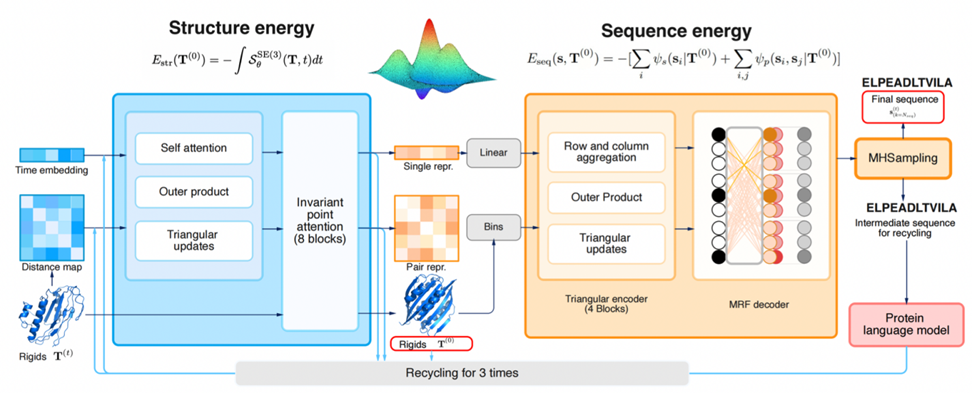
图 2:CarbonNovo 端到端生成蛋白质结构和序列。 (来源:论文)
CarbonNovo 的主要贡献总结如下:
蛋白质结构-序列的联合能量模型
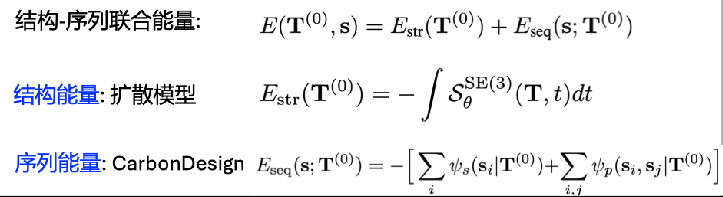
在经典物理模型下,天然蛋白质构象具有比较低的自由能,这也是蛋白质结构预测和设计的一般假设。基于此,CarbonNovo 建立了蛋白质结构和序列的联合能量模型:
图 2 展示了 CarbonNovo 的具体生成过程:
CarbonNovo 生成蛋白质结构-序列的性能评测

论文采用多种指标充分评价了 CarbonNovo 在蛋白质从头设计方面的性能 (图 3),例如,可折叠性、多样性、新颖性是领域内常用的评价指标。此外,论文还采用了 Rosetta 能量和语言模型下的似然概率(Sequence plausibility)作为评价指标。
CarbonNovo 和当前主流的「两阶段」设计模型做了比较,例如 RFdiffusion, Chroma, Genie, FrameDiff 和 FrameFlow。在最关键的可折叠性指标上 CarbonNovo 显著超过了所有基线方法,在其他指标上也显著超过基线方法或者和基线方法相当。
为了展示 CarbonNovo 在联合设计序列与结构方面的优势,作者还对比了使用 ProteinMPNN 生成序列的结果(图 3 a-c)。可以观察到,联合设计模型可以设计出更加匹配的蛋白质主链结构和序列。

作者进一步评估了 CarbonNovo 在不同长度的蛋白质设计上的性能(图 4)。在设计比较短的蛋白质时(例如长度 100),各个模型的表现相当。而随着蛋白质长度增长,CarbonNovo 的设计性能显著优于「两阶段」设计模型。
消融实验

作者训练了多个消融模型,以评估关键组件对 CarbonNovo 性能的相对贡献(图 5)。语言模型、序列设计模块和辅助训练损失对 CarbonNovo 的性能都有贡献。其中,语言模型的引入表现出最显著的贡献。此外,使用基于能量的序列设计模块相比自回归模型,也能显著提升序列设计的性能。
Case study:蛋白质结构「插值」
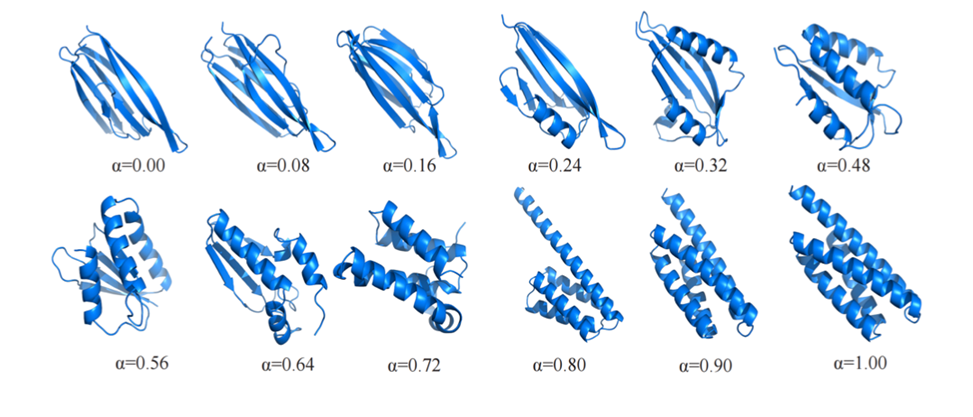
在图像生成领域,人脸图片插值/渐变是生成模型比较经典的应用。作者也尝试利用 CarbonNovo 进行蛋白质结构的插值。
图 5 展示了一个代表例子,随着在隐空间逐渐增加 all alpha-helices 结构向量的权重,生成的 all beta-sheets 结构会逐渐过渡到 all alpha-helices 结构。
This is the first interpolation experiment on protein structure in the field, and it also reflects that the protein latent space learned by CarbonNovo is relatively compact.
Conclusion
Finally, the author pointed out that although CarbonNovo mainly focuses on protein monomer design, it can also be easily extended to protein complex design and condition design, such as peptide design, antibody design, etc.
The author team is currently working with the biological experiment team to verify the protein designed by CarbonNovo through wet experiments.
The CarbonMatrix team where the author works has long been committed to AI protein design and AI drug design, and is establishing a unified generation model for the design and prediction of biological macromolecule structures.
His research results have been published in top machine learning conferences such as ICML and NeurIPS and top academic journals such as Nature Machine Intelligence and Nature Communications. He is also currently cooperating with biological laboratories to actively promote the industrialization of AI models in the field of drug design. Land.
The above is the detailed content of A team from the Institute of Computing Technology of the Chinese Academy of Sciences proposed CarbonNovo, an AI-based end-to-end de novo design of protein structures and sequences.. For more information, please follow other related articles on the PHP Chinese website!
Related articles
See more- Technology trends to watch in 2023
- How Artificial Intelligence is Bringing New Everyday Work to Data Center Teams
- Can artificial intelligence or automation solve the problem of low energy efficiency in buildings?
- OpenAI co-founder interviewed by Huang Renxun: GPT-4's reasoning capabilities have not yet reached expectations
- Microsoft's Bing surpasses Google in search traffic thanks to OpenAI technology

