Heim >Technologie-Peripheriegeräte >KI >Um mit kleinen Datenmengen eine hohe Vielseitigkeit zu erreichen, entwickelt KAIST ein neues Framework für die 3D-Molekülgenerierung für das Arzneimitteldesign
Um mit kleinen Datenmengen eine hohe Vielseitigkeit zu erreichen, entwickelt KAIST ein neues Framework für die 3D-Molekülgenerierung für das Arzneimitteldesign
- PHPznach vorne
- 2024-04-02 21:30:011354Durchsuche
Herausgeber |. Rettichhaut
Tief generative Modelle haben großes Potenzial, die Arzneimittelentwicklung zu beschleunigen. Allerdings stehen bestehende generative Modelle aufgrund der begrenzten Daten häufig vor Herausforderungen bei der Generalisierung, was zu weniger innovativen Designs führt.
Um diese Probleme anzugehen, schlugen Forscher am KAIST in Südkorea ein interaktionsbewusstes Funktionsgerüst für die 3D-Molekülerzeugung vor, das ein interaktionsgesteuertes Interaktionsdesign innerhalb der Zielbindungstasche ermöglicht. Durch die Nutzung allgemeiner Muster von Protein-Ligand-Wechselwirkungen als Vorwissen kann das Modell mit begrenzten experimentellen Daten ein hohes Maß an Allgemeingültigkeit erreichen. Gleichzeitig kann das Modell durch die Verwendung von Proteinmasse-Ligandenmasse als allgemeines Muster für Interaktionszwecke ein gutes Gleichgewicht zwischen Allgemeingültigkeit und hoher Spezifität erreichen, was Allgemeingültigkeit und Vorhersagbarkeit für das Arzneimitteldesign bietet.
Die Leistung der generierten unsichtbaren Zielliganden wurde umfassend bewertet, indem ihre Bindungssicherheit, Affinität, Diversität und Neuheit analysiert wurden. Darüber hinaus zeigt das effiziente Design potenzieller mutationsselektiver Inhibitoren die Anwendbarkeit dieses Ansatzes für das strukturbasierte Arzneimitteldesign.
Die Studie trug den Titel „3D Molecular Generative Framework for Interaction-Guided Drug Design“ und wurde am 27. März 2024 in „Nature Communications“ veröffentlicht.

Bei der Datenerfassung und bei wissenschaftlichen Problemen ist entsprechendes hierarchisches Vorwissen über Deep-Learning-Modelle entscheidend, um verallgemeinerbare Modelle zu entwickeln. AlphaFold sagt beispielsweise erfolgreich Proteinstrukturen voraus, indem es koevolutionäre Informationen und Darstellungen von Restpaaren nutzt. Tiefgreifende generative Modelle verändern das Arzneimitteldesign-Paradigma, ihre Leistung ist jedoch durch den Mangel an Aktivitätsdaten zu Arzneimittelmolekülen begrenzt, was zu geringen Generalisierungsfähigkeiten führt. Um die Leistung tiefer generativer Modelle zu verbessern, benötigen wir entsprechendes Vorwissen, um sicherzustellen, dass sie für die Verallgemeinerung von Aktivitätsdaten von Arzneimittelmolekülen geeignet sind, was für die Vorhersage anspruchsvoller Verbindungsstrukturen und -eigenschaften von entscheidender Bedeutung ist.
Aktuelle generative Funktionsmodelle verbessern die Wellenbildungsfähigkeiten des Modells, indem sie die dreidimensionale Struktur der Bindungsstelle für das strukturbasierte Ligandendesign nutzen, ohne auf Aktivitätsdaten angewiesen zu sein. Ein Well-Wave-Modell sollte die universellen Eigenschaften von Protein-Ligand-Wechselwirkungen verstehen, einschließlich Wasserstoffbrückenbindungen, Salzbrücken, hydrophoben Wechselwirkungen und π-π-Stapelung. Dies ist wichtig, um eine stabile Bindungsstruktur zu bilden und eine hohe Affinität aufrechtzuerhalten. Diese allgegenwärtigen Interaktionsmuster sind die Grundlage für die Entwicklung wirksamer Medikamente.
Basierend auf diesen Umständen schlugen KAIST-Forscher ein interaktionsbewusstes 3D-Molekülgenerierungs-Framework vor. Dieses Framework nutzt die universelle Natur von Protein-Ligand-Wechselwirkungen, um das strukturbasierte Arzneimitteldesign zu steuern. Das Framework besteht aus zwei Hauptphasen: (1) Einstellung der Bedingungen für die Interaktionserkennung und (2) Erzeugung interagierender 3D-Moleküle.
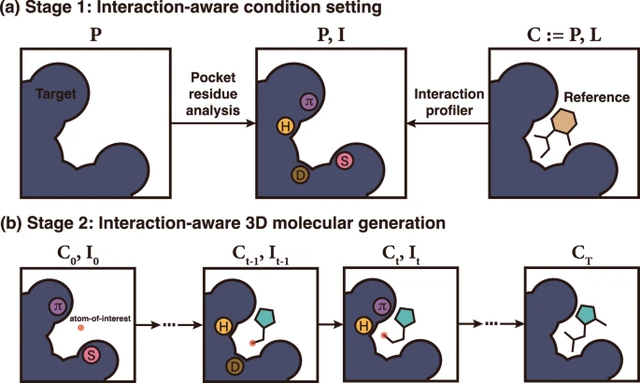
Illustration: Illustration des Rahmenkonzepts. (Quelle: Papier)
Die erste Stufe des Frameworks zielt darauf ab, die Interaktionsbedingungen I festzulegen, indem die Proteinatome für eine bestimmte Bindungsstelle P untersucht werden. Die Forscher nutzten vier Arten von Protein-Ligand-Wechselwirkungen: Wasserstoffbrückenbindungen, Salzbrücken, hydrophobe Wechselwirkungen und π-π-Stapelung. Hier berücksichtigten die Forscher nur die vier dominantesten Interaktionstypen in der Protein Data Bank (PDB), hauptsächlich weil sie für das Modelltraining den aus der PDB abgeleiteten PDBbind 2020-Datensatz verwendeten.
Gleichzeitig entwickelte das Team eine Strategie zur Regulierung der Protein-Atominteraktion. Die Forscher definieren Wechselwirkungsbedingungen als einen One-Hot-Vektor zusätzlicher Wechselwirkungstypen für eine Reihe von Proteinatomen, der angibt, ob ein Atom an einer bestimmten Wechselwirkung teilnehmen kann und welche Rolle es in der Wechselwirkung spielt.
Proteinatome werden in eine von sieben Kategorien unterteilt: Anionen, Kationen, Wasserstoffbrückenbindungsdonoren und -akzeptoren, aromatische, hydrophobe und nicht wechselwirkende Atome. Anstatt die gesamten Interaktionsinformationen als einen einzigen Interaktions-Fingerabdruck darzustellen, zielt die Strategie des Teams darauf ab, Interaktionsbedingungen lokal festzulegen.
In dieser Arbeit bestimmten die Forscher hauptsächlich die Wechselwirkungskategorien von Beutelatomen durch zwei Strategien.
Da während der Generierungsphase nicht immer Informationen zu Rezeptor-Ligand-Interaktionen verfügbar sind, werden Kriterien für Interaktionskategorien vordefiniert, um Interaktionsbedingungen durch Analyse jedes Proteinatoms festzulegen. Diese Bedingungseinstellung wird als Nicht-Referenz-Interaktionsbedingung bezeichnet.
Während der Trainingsphase werden Ground-Truth-Strukturen von Protein-Ligand-Komplexen verwendet, um Interaktionsbedingungen zu extrahieren.
Die Forscher schlugen außerdem ein tiefes generatives Modell namens DeepICL für das Reverse Engineering von Liganden vor, das basierend auf der dreidimensionalen Umgebung der Tasche und den Interaktionsbedingungen der ersten Stufe nach und nach Atome im Liganden erzeugt.
Obwohl die Zieltasche abhängig vom gebundenen Liganden und seiner Bindungshaltung unterschiedliche Kombinationen von Protein-Ligand-Wechselwirkungstypen bilden kann, besteht das Ziel des Teams darin, ein bedingtes generatives 3D-Modell namens DeepICL zu verwenden, um ein Modell zurückzuentwickeln, das bestimmte Wechselwirkungen erfüllt. Das Modell kann auf jede Art von Protein angewendet werden. Forscher nutzen lokale Interaktionsbedingungen in den Untertaschen, an die Liganden binden sollen, anstatt die gesamten Interaktionsinformationen zu nutzen, um unerwünschte Tendenzen zu bestimmten Taschen oder Ligandenstrukturen zu verhindern.
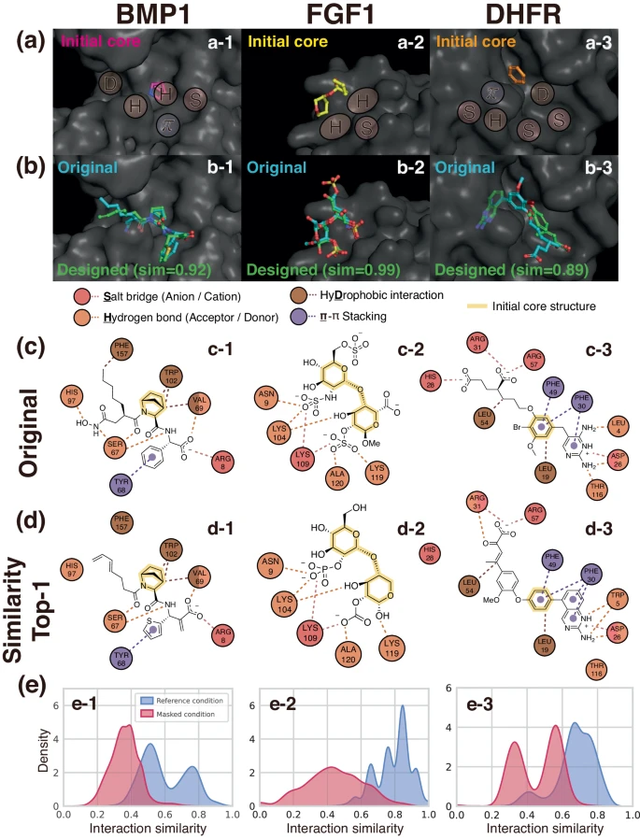
Abbildung: Beispiel einer interaktionsbewussten Ausarbeitung bedingter Liganden. (Quelle: Papier)
Um die Fähigkeit des Frameworks zu demonstrieren, ein allgemeines strukturbasiertes Arzneimitteldesign durchzuführen, verwendeten die Forscher anstelle der Verwendung typischer Benchmarks, die aus 105 bis 107 computergenerierten Protein-Ligand-Bindungsstrukturen bestehen, nur Daten aus der PDBbind-Datenbank Es wurden 104 reale Kristallstrukturen ausgewählt, da ein gutes Generalisierungsmodell auch für Daten im kleinen Maßstab erfolgreich geeignete Merkmale extrahieren kann.
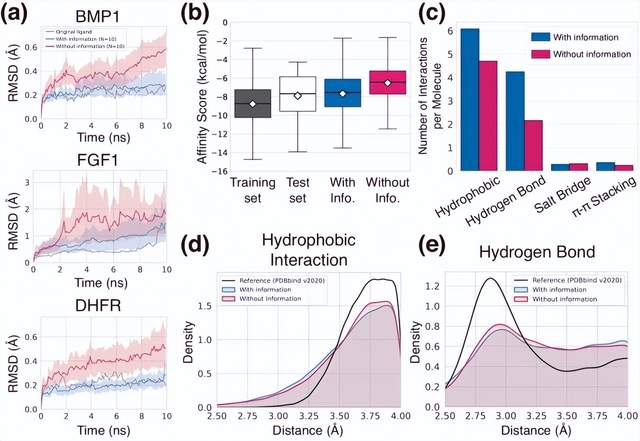
Illustration: Die Universalität des generativen Rahmens. (Quelle: Papier)
Die Forscher bewerteten ihr Modell, indem sie verschiedene Aspekte der Eigenschaften der erzeugten unsichtbaren Zielliganden analysierten – Bindungsstabilität, Affinität, geometrische Musterung, Diversität und Neuheit.
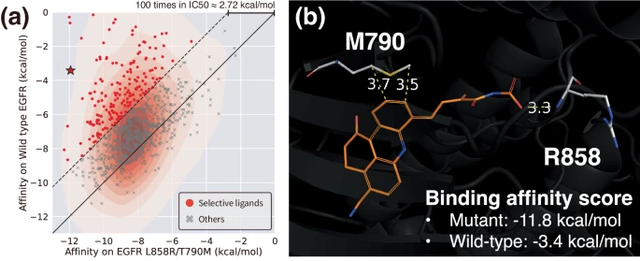
aAbbildung: Die Modulation der Selektivität durch ortsspezifische Wechselwirkungen steuert das Ligandendesign. (Quelle: Papier)
Die Forscher nutzten dieses Modell, um praktische Probleme zu lösen, bei denen bestimmte Interaktionsstellen eine entscheidende Rolle spielen, und demonstrierten damit die Anwendbarkeit ihres Ansatzes für das strukturbasierte Arzneimitteldesign.
Link zum Papier:https://www.nature.com/articles/s41467-024-47011-2
Das obige ist der detaillierte Inhalt vonUm mit kleinen Datenmengen eine hohe Vielseitigkeit zu erreichen, entwickelt KAIST ein neues Framework für die 3D-Molekülgenerierung für das Arzneimitteldesign. Für weitere Informationen folgen Sie bitte anderen verwandten Artikeln auf der PHP chinesischen Website!
In Verbindung stehende Artikel
Mehr sehen- Java-Methode zur Bestimmung, ob es sich um eine Primzahl (Primzahl) handelt
- Luxshare Precision: Das Unternehmen verfügt über ausgereifte Fähigkeiten und eine Geschäftsgrundlage für den Einstieg in aufstrebende Branchen wie humanoide Roboter
- In C++ geschrieben, ermitteln Sie die Anzahl der Primzahlen in einem Subarray
- Die flexible Crowdsourcing-Plattform unterstützt hochwertige Daten und eine effiziente menschliche Ausrichtung für die Modellindustrie im großen Maßstab

