Heim >Technologie-Peripheriegeräte >KI >Google Scientist Nature kommentiert: Wie künstliche Intelligenz das Gehirn besser verstehen kann
Google Scientist Nature kommentiert: Wie künstliche Intelligenz das Gehirn besser verstehen kann
- 王林nach vorne
- 2023-11-10 08:21:47882Durchsuche
Zusammengestellt |. Green Dill
Am 7. November 2023 veröffentlichte Viren Jain, leitender Forschungswissenschaftler bei Google Research und Leiter der Connectomics-Abteilung im Google-Team, einen Artikel mit dem Titel „Wie künstliche Intelligenz das Gehirn besser verstehen kann“. Übersichtsartikel „Nature“ (Wie KI zu einem besseren Verständnis des Gehirns führen könnte).
Link zum Papier: https://www.nature.com/articles/d41586-023-03426-3
Können Computer so programmiert werden, dass sie das Gehirn simulieren? Diese Frage stellen sich Mathematiker, Theoretiker und Experimentatoren schon lange – sei es aus dem Wunsch heraus, künstliche Intelligenz (KI) zu schaffen, oder weil ihr Verhalten nur dann verstanden werden kann, wenn Mathematik oder Computer komplexe Systeme wie das Gehirn reproduzieren können. Um diese Frage zu beantworten, entwickeln Forscher seit den 1940er Jahren vereinfachte Modelle der neuronalen Netze des Gehirns. Tatsächlich lässt sich die heutige Explosion des maschinellen Lernens auf frühe Arbeiten zurückführen, die von biologischen Systemen inspiriert waren.
Allerdings erlauben die Ergebnisse dieser Bemühungen den Forschern nun, eine etwas andere Frage zu stellen: Kann maschinelles Lernen verwendet werden, um Rechenmodelle zu erstellen, die die Gehirnaktivität simulieren?
Im Mittelpunkt dieser Entwicklungen stehen zunehmende Mengen an Gehirndaten. Seit den 1970er Jahren haben Neurowissenschaftler Konnektome erstellt, Karten neuronaler Verbindungen und Morphologie, die statische Darstellungen des Gehirns zu einem bestimmten Zeitpunkt erfassen, und diese Forschung wurde seitdem intensiviert. Zusätzlich zu diesen Fortschritten haben Forscher auch ihre Fähigkeit verbessert, funktionelle Aufzeichnungen zu erstellen, die Veränderungen der neuronalen Aktivität im Laufe der Zeit mit der Auflösung einzelner Zellen messen können. Mittlerweile können Forscher im Bereich der Transkriptomik die Genaktivität in Gewebeproben messen und sogar kartieren, wann und wo diese Aktivität auftritt.
Bisher wurden nur wenige Versuche unternommen, diese verschiedenen Datenquellen zu verbinden oder sie gleichzeitig aus dem gesamten Gehirn derselben Probe zu sammeln. Da jedoch der Detaillierungsgrad, die Größe und die Anzahl der Datensätze, insbesondere für die Gehirne relativ einfacher Modellorganismen, zunehmen, machen maschinelle Lernsysteme einen neuen Ansatz für die Gehirnmodellierung möglich. Dabei werden künstliche Intelligenzprogramme auf Konnektom- und andere Daten trainiert, um die neuronale Aktivität zu reproduzieren, die man in biologischen Systemen erwarten würde.
Informatische Neurowissenschaftler und andere müssen einige Herausforderungen lösen, bevor sie maschinelles Lernen nutzen können, um Simulationen des gesamten Gehirns zu erstellen. Ein hybrider Ansatz, der Informationen aus traditionellen Gehirnmodellierungstechniken mit maschinellen Lernsystemen kombiniert, die auf verschiedenen Datensätzen trainiert werden, kann jedoch den gesamten Aufwand präziser und informativer machen.
Gehirnkartierung
Die Suche nach einer Kartierung des Gehirns begann vor fast einem halben Jahrhundert mit 15 Jahren sorgfältiger Forschung am Nematoden Caenorhabditis elegans. In den letzten zwei Jahrzehnten haben Entwicklungen im Bereich der automatisierten Gewebeschnitte und Bildgebung den Forschern den Zugang zu anatomischen Daten erleichtert, während Fortschritte in der Computertechnik und der automatisierten Bildanalyse die Analyse dieser Datensätze verändert haben.
Connectomes wurden nun für das gesamte Gehirn von C. elegans, der Larve und dem erwachsenen Drosophila melanogaster sowie für kleine Teile (ein Tausendstel bzw. ein Millionstel) der Gehirne von Mäusen und Menschen generiert.
Die bisher erstellten Anatomiediagramme weisen große Lücken auf. Bildgebende Methoden konnten elektrische Verbindungen sowie chemische synaptische Verbindungen nicht in großem Maßstab abbilden. Die Forscher haben sich hauptsächlich auf Neuronen konzentriert, obwohl die nicht-neuronalen Gliazellen, die Neuronen unterstützen, offenbar eine entscheidende Rolle im Informationsfluss im Nervensystem spielen. Über die exprimierten Gene und die Proteine, die in den kartierten Neuronen und anderen Zellen vorhanden sind, ist noch viel Unbekanntes bekannt.
Nichtsdestotrotz haben solche Karten einige Erkenntnisse gebracht. Bei Drosophila melanogaster beispielsweise ermöglicht die Konnektomik den Forschern, die Mechanismen hinter neuronalen Schaltkreisen zu identifizieren, die für Verhaltensweisen wie Aggression verantwortlich sind. Die Gehirnkarte zeigte auch, wie Fruchtfliegen Informationen in den Schaltkreisen verarbeiten, die dafür verantwortlich sind, zu wissen, wo sie sich befinden und wie sie von einem Ort zum anderen gelangen. Bei Zebrafischlarven (Danio rerio) half die Konnektomik dabei, die Funktionsweise synaptischer Schaltkreise aufzudecken, die der Geruchsklassifizierung, der Kontrolle der Augenposition und -bewegung sowie der Navigation zugrunde liegen.
Bemühungen, die letztendlich ein komplettes Maus-Gehirn-Konnektom erzeugen könnten, sind im Gange – obwohl dies mit aktuellen Methoden ein Jahrzehnt oder länger dauern könnte. Das Gehirn von Mäusen ist fast 1.000-mal größer als das Gehirn von Drosophila melanogaster, das aus etwa 150.000 Neuronen besteht.
Zusätzlich zu all diesen Fortschritten in der Konnektomik nutzen Forscher die Einzelzell- und räumliche Transkriptomik, um Genexpressionsmuster mit immer größerer Genauigkeit und Spezifität zu erfassen. Verschiedene Techniken ermöglichen es Forschern auch, die neuronale Aktivität des gesamten Gehirns eines Wirbeltiers stundenlang aufzuzeichnen. Im Fall des Gehirns der Zebrafischlarve bedeutet dies, dass fast 100.000 Neuronen erfasst werden. Dazu gehören Proteine mit fluoreszierenden Eigenschaften, die sich als Reaktion auf Änderungen der Spannung oder des Kalziumspiegels ändern, sowie Mikroskopietechniken, die eine 3D-Bildgebung lebender Gehirne mit Einzelzellauflösung ermöglichen. (Die Aufzeichnung neuronaler Aktivität auf diese Weise liefert ein weniger genaues Bild als die elektrophysiologische Aufzeichnung, aber viel besser als nicht-invasive Methoden wie die funktionelle Magnetresonanztomographie.)
Mathe und Physik
beim Versuch, die Gehirnaktivität zu modellieren Wissenschaftler nutzen überwiegend physikbasierte Methoden. Dies erfordert die Erstellung einer Simulation eines Nervensystems oder von Teilen eines Nervensystems anhand einer mathematischen Beschreibung des Verhaltens realer Neuronen oder Teile eines realen Nervensystems. Es erfordert auch fundierte Vermutungen über Aspekte der Schaltung, die nicht durch Beobachtung verifiziert wurden, wie etwa die Netzwerkkonnektivität.
In einigen Fällen gibt es umfangreiche Spekulationen (siehe „Mystery Model“), aber in anderer Hinsicht helfen anatomische Karten mit Einzelzell- und Einzelsynapsenauflösung Forschern, Hypothesen zu widerlegen und aufzustellen.
Mysteriöse Modelle
Aufgrund fehlender Daten ist es schwierig zu beurteilen, ob bestimmte neuronale Netzwerkmodelle erfassen, was in realen Systemen passiert.
Das umstrittene European Human Brain Project, das im September endete, zielte ursprünglich darauf ab, das gesamte menschliche Gehirn rechnerisch zu simulieren. Obwohl dieses Ziel aufgegeben wurde, simulierte das Projekt Teile des Gehirns von Nagetieren und Menschen, darunter Zehntausende von Neuronen in einem Nagetier-Hippocampus-Modell, basierend auf begrenzten biologischen Messungen und einer Vielzahl synthetischer Datengenerierungsverfahren.
Ein großes Problem bei diesem Ansatz besteht darin, dass es mangels detaillierter anatomischer oder funktioneller Diagramme schwierig ist, zu beurteilen, wie genau die endgültige Simulation das Geschehen im biologischen System erfasst.
Seit etwa siebzig Jahren verfeinern Neurowissenschaftler theoretische Beschreibungen der Schaltkreise, die die Berechnung von Bewegungen in Drosophila melanogaster ermöglichen. Seit seiner Fertigstellung im Jahr 2013 lieferte das Konnektom des Bewegungserkennungsschaltkreises und später das größere Flugkonnektom detaillierte Schaltpläne, die einige Hypothesen über die Funktionsweise des Schaltkreises stützen.
Allerdings verdeutlichen die aus echten neuronalen Netzen gesammelten Daten auch die Grenzen anatomiebasierter Methoden.
Zum Beispiel umfasste ein in den 1990er Jahren fertiggestelltes neuronales Schaltkreismodell eine detaillierte Analyse der Konnektivität und Physiologie der etwa 30 Neuronen, aus denen das orogastrische Ganglion der Krabbe (Cancer borealis) besteht (die Struktur, die die Bewegung des Magens des Tieres steuert). . Durch die Messung der Aktivität von Neuronen unter verschiedenen Bedingungen stellten die Forscher fest, dass selbst bei relativ kleinen Ansammlungen von Neuronen scheinbar subtile Veränderungen, wie etwa die Einführung eines Neuromodulators (eine Substanz, die die Eigenschaften von Neuronen und Synapsen verändert), ebenfalls vollständig zum Tragen kommen das Verhalten der Schaltung ändern. Dies deutet darauf hin, dass die heutigen Daten selbst mit Konnektomen und anderen umfangreichen Datensätzen zur Führung und Einschränkung von Hypothesen über neuronale Schaltkreise möglicherweise nicht detailliert genug sind, damit Modellierer erfassen könnten, was in biologischen Systemen geschieht.
Dies ist ein Bereich, in dem maschinelles Lernen den Weg nach vorne weisen kann.
Durch die Optimierung von Tausenden oder sogar Milliarden von Parametern, die auf Konnektom- und anderen Daten basieren, können Modelle für maschinelles Lernen trainiert werden, um ein Verhalten neuronaler Netzwerke zu erzeugen, das mit dem Verhalten realer neuronaler Netzwerke übereinstimmt – gemessen anhand von Funktionsaufzeichnungen mit Zellauflösung.
Dieses Modell des maschinellen Lernens kann Informationen aus traditionellen Gehirnmodellierungstechniken einbeziehen, wie zum Beispiel dem Hodgkin-Huxley-Modell, das Aktionspotentiale (Änderungen der Transmembranspannung) in Neuronen beschreibt, die initiiert und verbreitet werden, und mithilfe optimierter parametrischer Konnektivitätskarten funktionale Aktivität Aufzeichnungen oder andere Datensätze, die für das gesamte Gehirn erfasst werden. Alternativ können Modelle für maschinelles Lernen „Black-Box“-Architekturen enthalten, die wenig explizit spezifiziertes biologisches Wissen enthalten, aber Milliarden oder Hunderte von Milliarden Parametern enthalten, die alle empirisch optimiert sind.
Forscher können solche Modelle beispielsweise bewerten, indem sie Vorhersagen über die neuronale Aktivität eines Systems mit Aufzeichnungen tatsächlicher biologischer Systeme vergleichen. Wenn maschinellen Lernprogrammen untrainierte Daten zur Verfügung gestellt werden, ist es von entscheidender Bedeutung, dass sie den Vergleich der Vorhersagen des Modells bewerten – wie es bei der Bewertung maschineller Lernsysteme üblich ist.
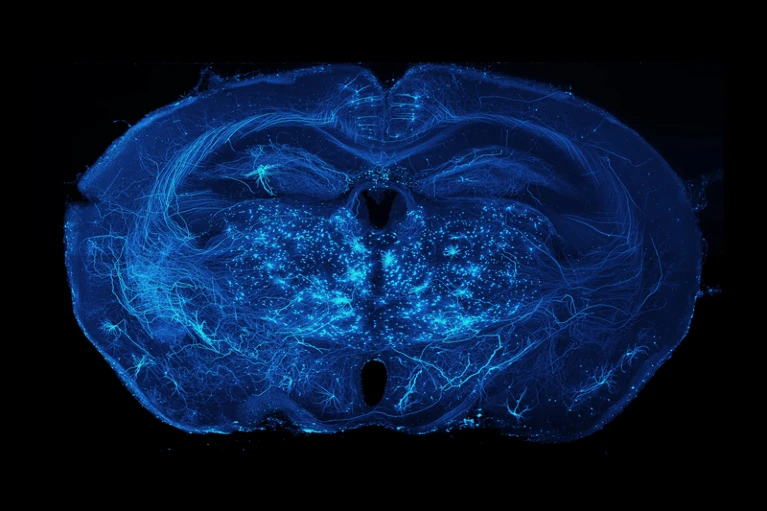
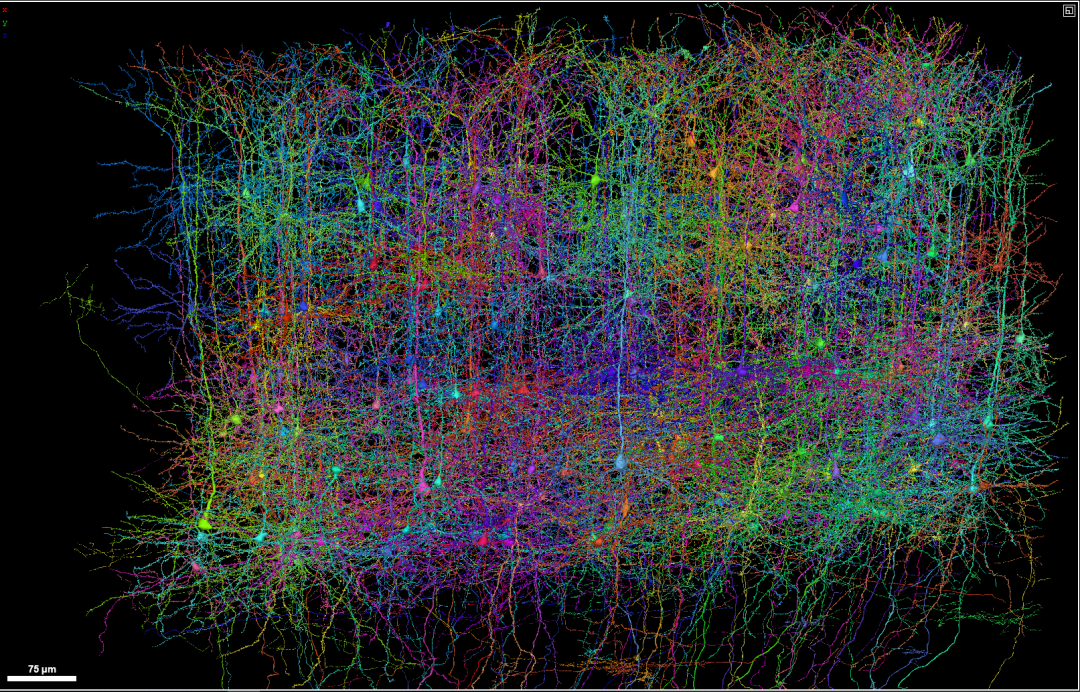
Axonale Projektionen von Neuronen im Gehirn der Maus. (Quelle: Adam Glaser, Jayaram Chandrashekar, Karel Svoboda, Allen Institute for Neurodynamics)
Dieser Ansatz ermöglicht eine genauere Modellierung von Gehirnen, die Tausende oder mehr Neuronen enthalten. Forscher können beispielsweise bewerten, ob einfachere Modelle, die leichter zu berechnen sind, neuronale Netze besser simulieren als komplexere Modelle, die detailliertere biophysikalische Informationen liefern, und umgekehrt.
Maschinelles Lernen wird auf diese Weise bereits eingesetzt, um das Verständnis anderer äußerst komplexer Systeme zu verbessern. Beispielsweise basieren Wettervorhersagesysteme seit den 1950er Jahren typischerweise auf sorgfältig konstruierten mathematischen Modellen meteorologischer Phänomene, und moderne Systeme sind das Ergebnis der iterativen Verfeinerung solcher Modelle durch Hunderte von Forschern. Allerdings haben Forscher in den letzten etwa fünf Jahren mehrere Wettervorhersagesysteme entwickelt, die maschinelles Lernen nutzen. Diese enthalten beispielsweise weniger Annahmen darüber, wie Druckgradienten Änderungen der Windgeschwindigkeit bewirken und wie die Windgeschwindigkeit Feuchtigkeit durch die Atmosphäre bewegt. Stattdessen werden Millionen von Parametern durch maschinelles Lernen optimiert, um ein simuliertes Wetterverhalten zu erzeugen, das mit einer Datenbank vergangener Wettermuster übereinstimmt.
Diese Vorgehensweise bringt einige Herausforderungen mit sich. Selbst wenn ein Modell genaue Vorhersagen macht, ist es schwierig zu erklären, wie es das macht. Darüber hinaus können Modelle häufig keine Szenarien vorhersagen, die nicht in ihren Trainingsdaten enthalten sind. Ein Wettermodell, das darauf trainiert ist, die nächsten Tage vorherzusagen, hat Schwierigkeiten, Vorhersagen auf Wochen oder Monate in die Zukunft zu übertragen. Aber in einigen Fällen – bei der Vorhersage von Regenfällen mehrere Stunden in der Zukunft – haben Methoden des maschinellen Lernens herkömmliche Methoden übertroffen. Modelle des maschinellen Lernens haben auch praktische Vorteile. Sie verwenden einen einfacheren zugrunde liegenden Code und können von Wissenschaftlern mit weniger speziellen meteorologischen Kenntnissen verwendet werden.
Für die Gehirnmodellierung könnte dieser Ansatz einerseits dazu beitragen, einige Lücken in aktuellen Datensätzen zu schließen und den Bedarf an detaillierteren Messungen einzelner biologischer Komponenten, wie beispielsweise einzelner Neuronen, zu verringern. Andererseits wird die Einbindung der Daten in das Modell einfacher, wenn umfassendere Datensätze verfügbar werden.
Think Big
Um diese Idee zu verwirklichen, müssen einige Herausforderungen gelöst werden.
Maschinelle Lernprogramme sind nur so gut wie die Daten, mit denen sie trainiert und ausgewertet werden. Daher sollten Neurowissenschaftler darauf abzielen, Datensätze aus dem gesamten Gehirn einer Probe zu gewinnen – oder sogar aus dem gesamten Körper, wenn dies möglich ist. Obwohl es einfacher ist, Daten aus bestimmten Teilen des Gehirns zu sammeln, ist es unwahrscheinlich, dass die Verwendung von maschinellem Lernen zur Modellierung hochgradig vernetzter Systeme wie neuronaler Netze nützliche Informationen liefert, wenn viele Teile des Systems in den zugrunde liegenden Daten nicht vorhanden sind.
Forscher sollten auch daran arbeiten, anatomische Karten neuronaler Verbindungen und Funktionsaufzeichnungen (und vielleicht in Zukunft auch Genexpressionskarten) aus ganzen Gehirnen aus derselben Probe zu erhalten. Gegenwärtig konzentrieren sich beide Gruppen eher darauf, das eine oder das andere zu bekommen, als auf beides.
Mit nur 302 Neuronen verfügt das Nervensystem von C. elegans möglicherweise über genügend feste Verdrahtungen, um Forschern die Annahme zu ermöglichen, dass die von einer Probe erhaltene Konnektivitätskarte für jede andere Probe dieselbe ist – obwohl einige Studien etwas anderes vermuten lassen. Aber für größere Nervensysteme wie die von Drosophila melanogaster und Zebrafischlarven ist die Connectome-Variation zwischen den Proben signifikant, daher sollten Gehirnmodelle auf strukturellen und funktionellen Daten trainiert werden, die aus derselben Probe gewonnen wurden.
Aktuell ist dies nur in zwei gängigen Modellorganismen möglich. Die Körper von C. elegans und Zebrafischlarven sind transparent, sodass Forscher funktionelle Aufzeichnungen vom gesamten Gehirn des Organismus machen und die Aktivität einzelner Neuronen genau bestimmen können. Im Anschluss an solche Aufzeichnungen können die Tiere sofort getötet, in Harz eingebettet und geschnitten werden sowie anatomische Messungen der Nervenverbindungen durchgeführt werden. In Zukunft könnten Forscher jedoch das Spektrum der Organismen, für die eine solche kombinierte Datenerfassung möglich ist, erweitern, indem sie beispielsweise neue nicht-invasive Methoden entwickeln, möglicherweise unter Verwendung von Ultraschall, um die neuronale Aktivität mit hoher Auflösung aufzuzeichnen.
Um solche multimodalen Datensätze in derselben Stichprobe zu erhalten, sind eine umfassende Zusammenarbeit zwischen Forschern, Investitionen in die Wissenschaft großer Teams und eine verstärkte Unterstützung der Finanzierungsagenturen für umfassendere Bemühungen erforderlich. Aber es gibt Präzedenzfälle für diesen Ansatz, wie zum Beispiel das MICrONS-Projekt des U.S. Intelligence Advanced Research Program Activity, das zwischen 2016 und 2021 funktionelle und anatomische Daten von 1 Kubikmillimeter Mausgehirn erhielt.
Zusätzlich zur Erhebung dieser Daten müssen sich Neurowissenschaftler auf wichtige Modellierungsziele und quantitative Metriken einigen, um den Fortschritt zu messen. Sollte das Ziel des Modells darin bestehen, das Verhalten einzelner Neuronen anhand vergangener Zustände oder des gesamten Gehirns vorherzusagen? Sollte die Aktivität eines einzelnen Neurons der Schlüsselindikator sein oder sollte es der Prozentsatz von Hunderttausenden aktiven Neuronen sein? Was macht außerdem eine genaue Darstellung der neuronalen Aktivität in einem biologischen System aus? Formelle, vereinbarte Benchmarks sind für den Vergleich von Modellierungsansätzen und die Verfolgung des Fortschritts im Zeitverlauf unerlässlich.
Abschließend müssen Forscher der breiteren wissenschaftlichen Gemeinschaft klarstellen, welche Modellierungsaufgaben die höchste Priorität haben und welche Metriken zur Bewertung der Modellleistung verwendet werden sollten, um verschiedenen Gemeinschaften, darunter Computerneurowissenschaftler und Experten für maschinelles Lernen, Herausforderungen bei der Gehirnmodellierung vorzustellen. WeatherBench, eine Online-Plattform, die einen Rahmen für die Bewertung und den Vergleich von Wettervorhersagemodellen bietet, bietet eine nützliche Vorlage.
Komplexität der Schlüsseltechniken
Einige werden – und das zu Recht – in Frage stellen, ob maschinelle Lernansätze zur Gehirnmodellierung wissenschaftlich nützlich sind. Könnte das Problem, zu verstehen, wie das Gehirn funktioniert, einfach durch das Problem ersetzt werden, zu verstehen, wie große künstliche Netzwerke funktionieren?
Es ist jedoch ermutigend zu sehen, dass in Zweigen der Neurowissenschaften ähnliche Ansätze verwendet werden, um zu bestimmen, wie das Gehirn sensorische Reize wie Sehen und Riechen verarbeitet und kodiert. Forscher nutzen zunehmend klassisch modellierte neuronale Netze, in denen einige biologische Details spezifiziert sind, kombiniert mit maschinellen Lernsystemen. Letztere werden an großen visuellen oder akustischen Datensätzen trainiert, um die visuellen oder auditiven Fähigkeiten des neuronalen Systems, beispielsweise die Bilderkennung, nachzubilden. Das resultierende Netzwerk zeigte verblüffende Ähnlichkeiten mit biologischen Netzwerken, war jedoch einfacher zu analysieren und abzufragen als echte neuronale Netzwerke.
Im Moment geht es vielleicht nur um die Frage, ob Daten aus aktuellen Gehirnatlanten und anderen Arbeiten maschinelle Lernmodelle trainieren können, um neuronale Aktivität zu reproduzieren, die dem entspricht, was in biologischen Systemen beobachtet wird. Hier kann sogar Scheitern Spaß machen – was nahelegt, dass die Mapping-Forschung tiefer gehen muss.
Das obige ist der detaillierte Inhalt vonGoogle Scientist Nature kommentiert: Wie künstliche Intelligenz das Gehirn besser verstehen kann. Für weitere Informationen folgen Sie bitte anderen verwandten Artikeln auf der PHP chinesischen Website!
In Verbindung stehende Artikel
Mehr sehen- Erläuterung theoretischer Kenntnisse zur PHP-Performance-Optimierung
- Welche Programmtheorie liegt dem virtuellen Speicherverwaltungssystem zugrunde?
- Wie kann man mit einer theoretischen Bandbreite von 56 Kbit/s auf das Internet zugreifen?
- Analysieren Sie Theorie und Praxis der Bewertung der Netzwerkinformationssicherheit

