Heim >Backend-Entwicklung >Python-Tutorial >Python-Bibliotheken: Ein umfassender Leitfaden zum Schreiben, Verpacken und Verteilen
Python-Bibliotheken: Ein umfassender Leitfaden zum Schreiben, Verpacken und Verteilen
- WBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOriginal
- 2023-09-03 17:37:011153Durchsuche
Python ist eine großartige Programmiersprache, aber die Paketierung ist einer ihrer schwächsten Punkte. Dies ist eine bekannte Tatsache in der Gesellschaft. Der Prozess des Installierens, Importierens, Verwendens und Erstellens von Paketen hat sich im Laufe der Jahre stark verbessert, ist aber immer noch nicht mit neueren Sprachen wie Go und Rust vergleichbar, die viel aus den Schwierigkeiten von Python und anderen ausgereiften Sprachen gelernt haben Sprachen.
In diesem Tutorial erfahren Sie alles, was Sie über das Schreiben, Verpacken und Verteilen Ihrer eigenen Pakete wissen müssen.
So schreiben Sie eine Python-Bibliothek
Eine Python-Bibliothek ist eine zusammenhängende Sammlung von Python-Modulen, die in Python-Paketen organisiert sind. Im Allgemeinen bedeutet dies, dass sich alle Module im selben Verzeichnis befinden und dass sich dieses Verzeichnis im Python-Suchpfad befindet.
Lassen Sie uns schnell ein kleines Python 3-Paket schreiben und alle diese Konzepte veranschaulichen.
Pathologie-Kit
Python 3 ist eine enorme Verbesserung mit einem hervorragenden Path 对象,这相对于 Python 2 笨拙的 os.path Modul. Es fehlt jedoch eine wichtige Funktion: das Finden des Pfads zum aktuellen Skript. Dies ist wichtig, wenn Sie Zugriffsdateien relativ zum aktuellen Skript positionieren möchten.
In vielen Fällen kann das Skript überall installiert werden, sodass absolute Pfade nicht verwendet werden können, und das Arbeitsverzeichnis kann auf einen beliebigen Wert festgelegt werden, sodass relative Pfade nicht verwendet werden können. Wenn Sie auf Dateien in einem Unterverzeichnis oder übergeordneten Verzeichnis zugreifen möchten, müssen Sie das aktuelle Skriptverzeichnis ermitteln können.
So geht's in Python:
import pathlib script_dir = pathlib.Path(__file__).parent.resolve()
Um auf eine Datei mit dem Namen „file.txt“ im Unterverzeichnis „data“ des aktuellen Skriptverzeichnisses zuzugreifen, können Sie den folgenden Code verwenden: print(open(str(script_dir/' data/file.txt').read())
Mit dem Pathologiepaket verfügen Sie über eine integrierte script_dir-Methode, die Sie wie folgt verwenden können:
from pathology.Path import script_dir print(open(str(script_dir()/'data/file.txt').read())
Ja, es ist etwas schwierig auszusprechen. Das Pathologiepaket ist sehr einfach. Es leitet seine eigene Path-Klasse von pathlibs Path ab und fügt ein statisches script_dir() hinzu, das immer den Pfad des aufrufenden Skripts zurückgibt.
Hier ist die Umsetzung:
import pathlib
import inspect
class Path(type(pathlib.Path())):
@staticmethod
def script_dir():
print(inspect.stack()[1].filename)
p = pathlib.Path(inspect.stack()[1].filename)
return p.parent.resolve()
Aufgrund der plattformübergreifenden Implementierung von pathlib.Path können Sie direkt davon ableiten und müssen von einer bestimmten Unterklasse (PosixPathoder pathlib.Path的跨平台实现,您可以直接从它派生,并且必须从特定子类派生(PosixPath or WindowsPath)。 script_dir
WindowsPath). Beim Parsen von <code>script_dir werden Inspektionsmodule verwendet, um den Aufrufer und seine Dateinamenattribute zu finden. Test-Pathologie-Kit
Jedes Mal, wenn Sie mehr als ein einmaliges Skript schreiben, sollten Sie es testen. Das Pathologiemodul bildet da keine Ausnahme. Hier sind die Tests mit dem Standard-Unit-Test-Framework:
import os
import shutil
from unittest import TestCase
from pathology.path import Path
class PathTest(TestCase):
def test_script_dir(self):
expected = os.path.abspath(os.path.dirname(__file__))
actual = str(Path.script_dir())
self.assertEqual(expected, actual)
def test_file_access(self):
script_dir = os.path.abspath(os.path.dirname(__file__))
subdir = os.path.join(script_dir, 'test_data')
if Path(subdir).is_dir():
shutil.rmtree(subdir)
os.makedirs(subdir)
file_path = str(Path(subdir)/'file.txt')
content = '123'
open(file_path, 'w').write(content)
test_path = Path.script_dir()/subdir/'file.txt'
actual = open(str(test_path)).read()
self.assertEqual(content, actual)
Python-Pfad
sys.path 中可用。这是我当前的 sys.pathPython-Pakete müssen irgendwo im Python-Suchpfad installiert sein, damit sie von Python-Modulen importiert werden können. Der Python-Suchpfad ist eine Liste von Verzeichnissen und wird immer unter
gefunden
>>> print('\n'.join(sys.path))
/Users/gigi.sayfan/miniconda3/envs/py3/lib/python36.zip
/Users/gigi.sayfan/miniconda3/envs/py3/lib/python3.6
/Users/gigi.sayfan/miniconda3/envs/py3/lib/python3.6/lib-dynload
/Users/gigi.sayfan/miniconda3/envs/py3/lib/python3.6/site-packages
/Users/gigi.sayfan/miniconda3/envs/py3/lib/python3.6/site-packages/setuptools-27.2.0-py3.6.egg
Beachten Sie, dass die erste leere Zeile der Ausgabe das aktuelle Verzeichnis darstellt, sodass Sie Module aus dem aktuellen Arbeitsverzeichnis importieren können, egal um welches Verzeichnis es sich handelt. Sie können Verzeichnisse direkt zu sys.path hinzufügen oder daraus entfernen.
PYTHONPATH 环境变量,还有一些其他方法可以控制它。默认情况下包含标准 site-packagesSie können auch einen
So verpacken Sie Python-Bibliotheken
Da wir nun den Code und die Tests haben, packen wir alles in die entsprechende Bibliothek. Python bietet einen einfachen Weg durch das Setup-Modul. Sie erstellen eine Datei namens setup.py
im Stammverzeichnis Ihres Pakets.
Die Datei setup.pypackages 项的补充,该项使用从 setuptools 导入的 find_packages() enthält viele Metadateninformationen wie Autor, Lizenz, Betreuer und andere Informationen zum Paket. Dies ist ein Paar
Dies ist die Datei setup.py
des Pathologiepakets:
from setuptools import setup, find_packages
setup(name='pathology',
version='0.1',
url='https://github.com/the-gigi/pathology',
license='MIT',
author='Gigi Sayfan',
author_email='the.gigi@gmail.com',
description='Add static script_dir() method to Path',
packages=find_packages(exclude=['tests']),
long_description=open('README.md').read(),
zip_safe=False)
QuellenverteilungspaketEine Quelldistribution ist ein Archiv, das Python-Pakete, Module und andere Dateien enthält, die für die Paketverteilung verwendet werden (z. B. Version 1, Version 2 usw.). Sobald die Datei verteilt ist, können Endbenutzer sie herunterladen und auf ihren Betriebssystemen installieren.
python setup.py sdistUm ein Quellverteilungspaket (sdist) zu erstellen, führen Sie Folgendes aus:
Lassen Sie uns eine Quellcodeverteilung erstellen:
$ python setup.py sdist running sdist running egg_info creating pathology.egg-info writing pathology.egg-info/PKG-INFO writing dependency_links to pathology.egg-info/dependency_links.txt writing top-level names to pathology.egg-info/top_level.txt writing manifest file 'pathology.egg-info/SOURCES.txt' reading manifest file 'pathology.egg-info/SOURCES.txt' writing manifest file 'pathology.egg-info/SOURCES.txt' warning: sdist: standard file not found: should have one of README, README.rst, README.txt running check creating pathology-0.1 creating pathology-0.1/pathology creating pathology-0.1/pathology.egg-info copying files to pathology-0.1... copying setup.py -> pathology-0.1 copying pathology/__init__.py -> pathology-0.1/pathology copying pathology/path.py -> pathology-0.1/pathology copying pathology.egg-info/PKG-INFO -> pathology-0.1/pathology.egg-info copying pathology.egg-info/SOURCES.txt -> pathology-0.1/pathology.egg-info copying pathology.egg-info/dependency_links.txt -> pathology-0.1/pathology.egg-info copying pathology.egg-info/not-zip-safe -> pathology-0.1/pathology.egg-info copying pathology.egg-info/top_level.txt -> pathology-0.1/pathology.egg-info Writing pathology-0.1/setup.cfg creating dist Creating tar archive removing 'pathology-0.1' (and everything under it)Diese Warnung liegt daran, dass ich eine nicht standardmäßige Datei README.md verwendet habe. Es ist sicher, es zu ignorieren. Der obige Befehl erstellt eine Archivdatei im Standardformat für das aktuelle Betriebssystem. Für Unix-Systeme wird eine komprimierte TAR-Datei im Verzeichnis dist generiert:
$ ls -la dist total 8 drwxr-xr-x 3 gigi.sayfan gigi.sayfan 102 Apr 18 21:20 . drwxr-xr-x 12 gigi.sayfan gigi.sayfan 408 Apr 18 21:20 .. -rw-r--r-- 1 gigi.sayfan gigi.sayfan 1223 Apr 18 21:20 pathology-0.1.tar.gzWenn Sie Windows verwenden, wird eine ZIP-Datei generiert.
Sie können auch andere zusätzliche Dateiformate angeben, indem Sie die unten gezeigten Formatoptionen verwenden. 🎜
python setup.py sdist --formats=gztar,zip
例如,上述命令将生成一个 gzip 压缩的 tarball 和一个 zip 文件。
可用的不同格式有:
-
zip: .zip -
gztar: .tar.gz -
bztar: .tar.bz2 -
xztar: .tar.xz -
ztar: .tar.Z -
tar: .tar
二进制分发
要创建一个名为“wheel”的二进制发行版,请运行: python setup.py bdist_wheel
这是一个二进制发行版:
$ python setup.py bdist_wheel running bdist_wheel running build running build_py creating build creating build/lib creating build/lib/pathology copying pathology/__init__.py -> build/lib/pathology copying pathology/path.py -> build/lib/pathology installing to build/bdist.macosx-10.7-x86_64/wheel running install running install_lib creating build/bdist.macosx-10.7-x86_64 creating build/bdist.macosx-10.7-x86_64/wheel creating build/bdist.macosx-10.7-x86_64/wheel/pathology copying build/lib/pathology/__init__.py -> build/bdist.macosx-10.7-x86_64/wheel/pathology copying build/lib/pathology/path.py -> build/bdist.macosx-10.7-x86_64/wheel/pathology running install_egg_info running egg_info writing pathology.egg-info/PKG-INFO writing dependency_links to pathology.egg-info/dependency_links.txt writing top-level names to pathology.egg-info/top_level.txt reading manifest file 'pathology.egg-info/SOURCES.txt' writing manifest file 'pathology.egg-info/SOURCES.txt' Copying pathology.egg-info to build/bdist.macosx-10.7-x86_64/wheel/pathology-0.1-py3.6.egg-info running install_scripts creating build/bdist.macosx-10.7-x86_64/wheel/pathology-0.1.dist-info/WHEEL
病理包仅包含纯Python模块,因此可以构建通用包。如果您的软件包包含 C 扩展,则必须为每个平台构建单独的轮子:
$ ls -la dist total 16 drwxr-xr-x 4 gigi.sayfan gigi.sayfan 136 Apr 18 21:24 . drwxr-xr-x 13 gigi.sayfan gigi.sayfan 442 Apr 18 21:24 .. -rw-r--r-- 1 gigi.sayfan gigi.sayfan 2695 Apr 18 21:24 pathology-0.1-py3-none-any.whl -rw-r--r-- 1 gigi.sayfan gigi.sayfan 1223 Apr 18 21:20 pathology-0.1.tar.gz
要更深入地了解打包 Python 库的主题,请查看如何编写您自己的 Python 包。
如何分发 Python 包
Python 有一个名为 PyPI(Python 包索引)的中央包存储库。 PyPI 可以轻松管理不同版本的包。例如,如果用户需要安装特定的软件包版本,pip 知道在哪里查找它。
当您使用 pip 安装 Python 包时,它将从 PyPI 下载该包(除非您指定不同的存储库)。为了分发我们的病理包,我们需要将其上传到 PyPI 并提供 PyPI 所需的一些额外元数据。步骤是:
- 升级您的 pip 版本。
- 在 PyPI 上创建一个帐户(只需一次)。
- 注册您的包裹。
- 上传您的包。
升级您的 pip 版本
确保您的操作系统中安装了最新版本的 pip。要升级 pip,请发出以下命令
python3 -m pip install --upgrade pip
创建帐户
您可以在 PyPI 网站上创建帐户。然后在您的主目录中创建一个 .pypirc 文件:
[distutils] index-servers=pypi [pypi] repository = https://pypi.python.org/pypi username = the_gigi
出于测试目的,您可以将 pypitest 索引服务器添加到您的 .pypirc 文件中:
[distutils]
index-servers=
pypi
pypitest
[pypitest]
repository = https://testpypi.python.org/pypi
username = the_gigi
[pypi]
repository = https://pypi.python.org/pypi
username = the_gigi
注册您的包裹
如果这是您的软件包的第一个版本,您需要使用 PyPI 注册它。使用setup.py的注册命令。它会询问您的密码。请注意,我将其指向此处的测试存储库:
$ python setup.py register -r pypitest running register running egg_info writing pathology.egg-info/PKG-INFO writing dependency_links to pathology.egg-info/dependency_links.txt writing top-level names to pathology.egg-info/top_level.txt reading manifest file 'pathology.egg-info/SOURCES.txt' writing manifest file 'pathology.egg-info/SOURCES.txt' running check Password: Registering pathology to https://testpypi.python.org/pypi Server response (200): OK
上传您的包
现在包已注册,我们可以上传它了。我建议使用麻线,这样更安全。像往常一样使用 pip install twine 安装它。然后使用 twine 上传您的包并提供您的密码(在下面进行编辑):
$ twine upload -r pypitest -p <redacted> dist/* Uploading distributions to https://testpypi.python.org/pypi Uploading pathology-0.1-py3-none-any.whl [================================] 5679/5679 - 00:00:02 Uploading pathology-0.1.tar.gz [================================] 4185/4185 - 00:00:01
该软件包现已在 PyPI 官方网站上提供,如下所示。
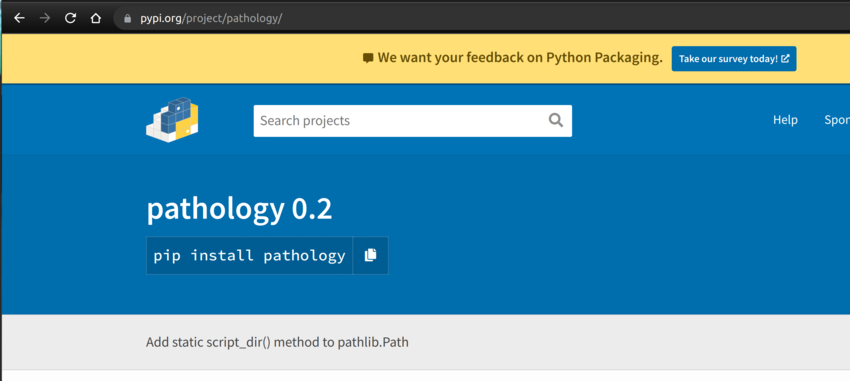
要使用 pip 安装它,只需发出以下命令:
pip install pathology
要更深入地了解分发包的主题,请查看如何共享您的 Python 包。
结论
在本教程中,我们完成了编写 Python 库、打包并通过 PyPI 分发它的完整过程。此时,您应该拥有编写库并与世界共享库的所有工具。
本文已根据 Esther Vaati 的贡献进行了更新。 Esther 是 Envato Tuts+ 的软件开发人员和作家。
Das obige ist der detaillierte Inhalt vonPython-Bibliotheken: Ein umfassender Leitfaden zum Schreiben, Verpacken und Verteilen. Für weitere Informationen folgen Sie bitte anderen verwandten Artikeln auf der PHP chinesischen Website!

