Heim >Technologie-Peripheriegeräte >KI >Tsinghua AIR und andere schlugen ESM-AA vor, das erste Proteinsprachenmodell von Aminosäuren bis hin zu atomaren Skalen
Tsinghua AIR und andere schlugen ESM-AA vor, das erste Proteinsprachenmodell von Aminosäuren bis hin zu atomaren Skalen
- WBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOriginal
- 2024-06-28 18:10:061272Durchsuche

Forschungsteams der Tsinghua University AIR, der Peking University und der Nanjing University haben das ESM-AA-Modell vorgeschlagen. Dieses Modell hat im Bereich der Proteinsprachenmodellierung wichtige Fortschritte gemacht und eine einheitliche Modellierungslösung bereitgestellt, die Informationen auf mehreren Skalen integriert.
Es ist das erste vorab trainierte Protein-Sprachmodell, das sowohl Aminosäureinformationen als auch Atominformationen verarbeiten kann. Die hervorragende Leistung des Modells zeigt das große Potenzial der einheitlichen Multiskalenmodellierung zur Überwindung bestehender Einschränkungen und zur Erschließung neuer Möglichkeiten.
Als Basismodell hat ESM-AA die Aufmerksamkeit und ausführliche Diskussion vieler Wissenschaftler erhalten (siehe Screenshot unten) und es wird davon ausgegangen, dass es das Potenzial hat, auf ESM-AA basierende Modelle zu entwickeln, die mit AlphaFold3 und RoseTTAFold All-Atom konkurrieren können . Es eröffnet neue Möglichkeiten zur Untersuchung der Wechselwirkungen zwischen verschiedenen biologischen Strukturen. Das aktuelle Papier wurde vom ICML 2024 angenommen.
Forschungshintergrund
Protein ist der wichtigste Auslöser verschiedener Lebensaktivitäten. Ein tiefgreifendes Verständnis von Proteinen und ihren Wechselwirkungen mit anderen biologischen Strukturen ist ein Kernthema der Biowissenschaften, das für gezieltes Arzneimittelscreening, Enzym-Engineering und andere Bereiche von erheblicher praktischer Bedeutung ist.
Daher ist die Frage, wie man Proteine besser verstehen und modellieren kann, zu einem Forschungsschwerpunkt im Bereich AI4Science geworden.
In den letzten Tagen haben auch große Spitzenforschungseinrichtungen, darunter Deepmind und die Baker Group der University of Washington, eingehende Untersuchungen zum Problem der Protein-All-Atom-Modellierung durchgeführt und Methoden vorgeschlagen, darunter AlphaFold 3 und RoseTTAFold All-Atom-Modelle usw. für Proteine und andere Lebensaktivitäten können mit hoher Genauigkeit genaue Vorhersagen der Proteinstruktur, der Molekülstruktur, der Rezeptor-Liganden-Struktur und anderer atomarer Skalen erzielen.
Obwohl diese Modelle erhebliche Fortschritte bei der Strukturmodellierung auf atomarer Ebene gemacht haben, sind aktuelle gängige Proteinsprachenmodelle immer noch nicht in der Lage, Proteinverständnis und Repräsentationslernen auf atomarer Ebene zu erreichen.
Multiskalen, der „notwendige Weg“ für die nächste Generation von Proteinmodellen
Die von ESM-2 dargestellten Lernmodelle für die Proteindarstellung verwenden Aminosäuren als einzige Skala zum Erstellen von Modellen, die für Situationen geeignet ist, auf die sich der Schwerpunkt konzentriert Die Verarbeitung von Proteinen ist ein vernünftiger Ansatz.
Der Schlüssel zum vollständigen Verständnis der Natur von Proteinen liegt jedoch in der Aufklärung ihrer Wechselwirkungen mit anderen biologischen Strukturen wie kleinen Molekülen, DNA, RNA usw.
Angesichts dieser Anforderung ist es notwendig, die komplexen Wechselwirkungen zwischen verschiedenen Strukturen zu beschreiben, und es ist für eine Modellierungsstrategie auf einem einzigen Maßstab schwierig, eine wirksame umfassende Abdeckung bereitzustellen.
Um dieses Manko zu überwinden, durchlaufen Proteinmodelle eine tiefgreifende Innovation hin zu Multiskalenmodellen. Beispielsweise führte das Anfang Mai in der Zeitschrift Science veröffentlichte RoseTTAFold All-Atom-Modell als Nachfolgeprodukt von RoseTTAFold das Konzept der Multiskalen ein.
Dieses Modell ist nicht auf die Vorhersage der Proteinstruktur beschränkt, sondern erstreckt sich auch auf breitere Forschungsfelder wie das Andocken von Proteinen und Molekülen/Nukleinsäuren, posttranslationale Proteinmodifikationen usw.
Gleichzeitig verwendet DeepMinds neu veröffentlichtes AlphaFold3 auch eine Multiskalen-Modellierungsstrategie, um die Vorhersage der Struktur mehrerer Proteinkomplexe zu unterstützen. Seine Leistung ist beeindruckend und wird zweifellos einen großen Einfluss auf die Bereiche künstliche Intelligenz und Biologie haben .
ESM All-Atom, eine mehrskalige Proteinsprachenmodellbasis
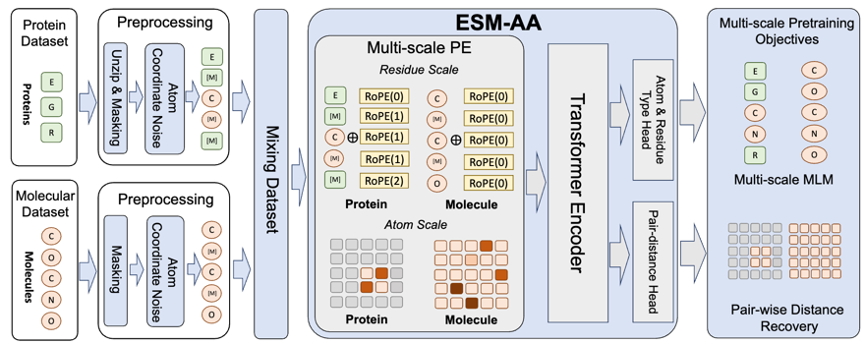
Die erfolgreiche Anwendung von RoseTTAFold All-Atom und AlphaFold3 auf das Multi- Das Skalenkonzept inspirierte eine wichtige Überlegung: Wie sollte das Proteinsprachenmodell als Proteinbasismodell die Multiskalentechnologie übernehmen? Auf dieser Grundlage schlug das Team das mehrskalige Proteinsprachenmodell ESM All-Atom (ESM-AA) vor.
Kurz gesagt stellt ESM-AA das Konzept der Multiskalen vor, indem es einige Aminosäuren in die entsprechende atomare Zusammensetzung „entpackt“. Anschließend wurde ein Vortraining durch Mischen von Proteindaten und molekularen Daten durchgeführt, wodurch das Modell in die Lage versetzt wurde, biologische Strukturen unterschiedlicher Größenordnung gleichzeitig zu verarbeiten.
Um dem Modell dabei zu helfen, qualitativ hochwertige Informationen im atomaren Maßstab besser zu lernen, wird ESM-AA außerdem molekulare Strukturdaten im atomaren Maßstab für das Training verwenden. Darüber hinaus kann das ESM-AA-Modell durch die Einführung des in Abbildung 2 gezeigten Multiskalen-Positionskodierungsmechanismus gut zwischen Informationen auf verschiedenen Skalen unterscheiden und so sicherstellen, dass das Modell Positions- und Strukturinformationen auf Rest- und Atomebene genau verstehen kann.
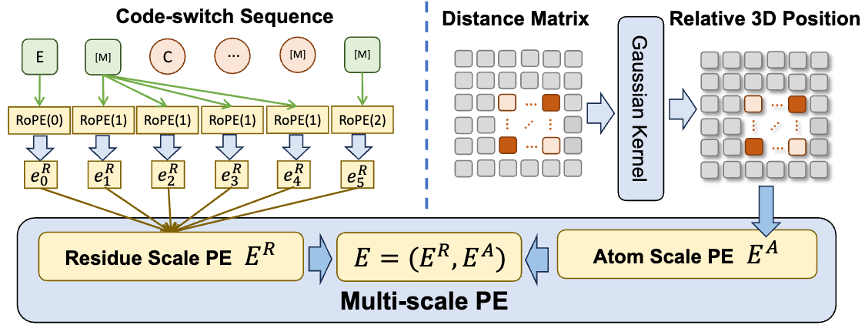
Multiskalige Vortrainingsziele
Um das Modell beim Erlernen von Multiskaleninformationen zu unterstützen, hat das Team verschiedene Vortrainingsziele für das ESM-AA-Modell entworfen. Zu den mehrskaligen Vortrainingszielen von ESM-AA gehören Masked Language Modeling (MLM) und Pairwise Distance Recovery (PDR). Wie in Abbildung 3(a) gezeigt, erfordert MLM, dass das Modell Vorhersagen auf der Grundlage des umgebenden Kontexts trifft, indem es Aminosäuren und Atome maskiert. Diese Trainingsaufgabe kann sowohl auf der Aminosäure- als auch auf der Atomskala durchgeführt werden. PDR erfordert, dass das Modell den euklidischen Abstand zwischen verschiedenen Atomen genau vorhersagt, um das Modell zu trainieren, Strukturinformationen auf atomarer Ebene zu verstehen (wie in Abbildung 3(b) dargestellt). Abbildung 3: Multiskalen-Vortrainingsaufgabe -Substrat-Substanz-Affinitäts-Regressionsaufgabe (Ergebnisse sind in Abbildung 4 dargestellt), Enzym-Substrat-Paar-Klassifizierungsaufgabe (Ergebnisse sind in Abbildung 4 dargestellt) und Arzneimittel-Ziel-Affinitäts-Regressionsaufgabe (Ergebnisse sind in Abbildung 5 dargestellt).
Die Ergebnisse zeigen, dass ESM-AA frühere Modelle bei diesen Aufgaben übertrifft, was darauf hindeutet, dass es das Potenzial von Protein-vortrainierten Sprachmodellen auf der Aminosäure- und Atomskala voll ausschöpft. 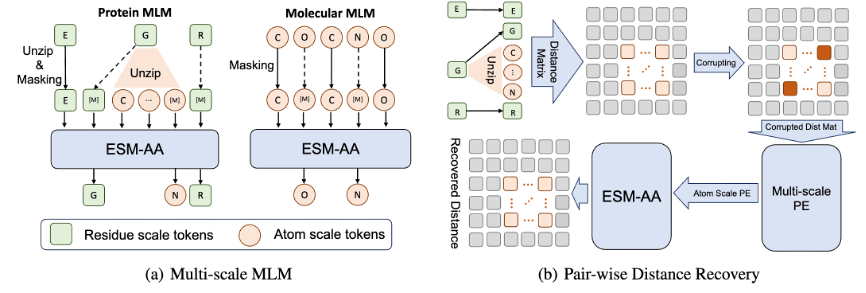
Abbildung 4: Leistungsvergleich der Enzym-Substrat-Affinitäts-Regressionsaufgabe und der Enzym-Substrat-Paar-Klassifizierungsaufgabe
Abbildung 5: Leistungsvergleich der Arzneimittel-Ziel-Affinitäts-Regressionsaufgabe
Darüber hinaus die ESM-AA Die Leistung des Modells wurde auch an Aufgaben wie der Vorhersage von Proteinkontakten, der Proteinfunktionsklassifizierung und der Vorhersage molekularer Eigenschaften getestet. Die Ergebnisse zeigen, dass ESM-AA bei Aufgaben, die nur Proteine betreffen, eine gleichwertige Leistung erbringt wie ESM-2; das ESM-AA-Modell übertrifft die meisten Benchmark-Modelle und ähnelt Uni-Mol. Dies zeigt, dass ESM-AA seine Fähigkeit, Proteine zu verstehen, beim Erwerb leistungsstarker molekularer Kenntnisse nicht opfert. Es zeigt außerdem, dass das ESM-AA-Modell das Wissen des ESM-2-Modells erfolgreich wiederverwendet, ohne von diesem ausgehen zu müssen Neuentwicklung, wodurch die Kosten für die Modellschulung erheblich gesenkt werden.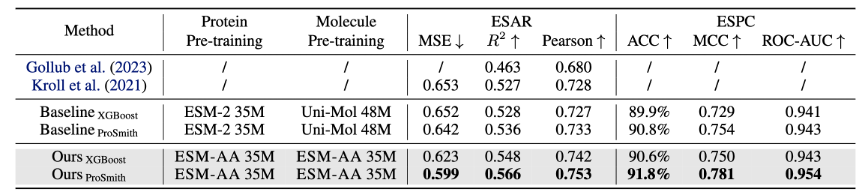
Um die Gründe weiter zu analysieren, warum ESM-AA bei der Protein-Kleinmolekül-Benchmark-Aufgabe gut abschneidet, zeigt dieses Papier die Extraktion des ESM-AA-Modells und des ESM-2+Uni-Mol Modellkombination in dieser Aufgabe Visualisierung der Stichprobendarstellungsverteilung. 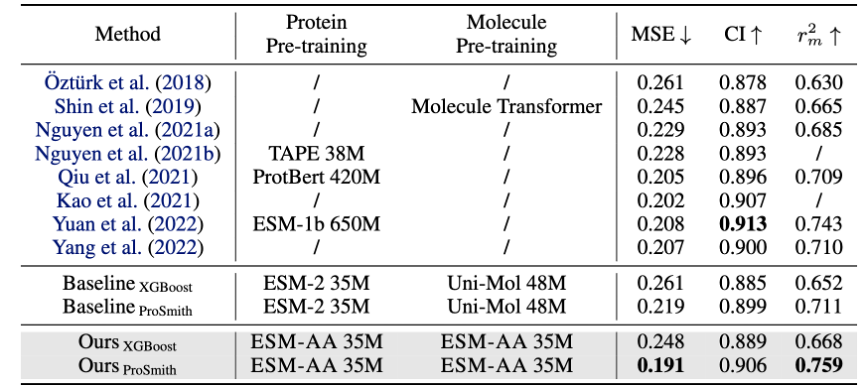
Multi-Scale Protein Language Model for Unified Molecular Modeling
Github Open-Source-Adresse:https://github.com/zhengkangjie/ESM-AA
Link zum Papier:https://arxiv.org/abs/2403.12995
Das obige ist der detaillierte Inhalt vonTsinghua AIR und andere schlugen ESM-AA vor, das erste Proteinsprachenmodell von Aminosäuren bis hin zu atomaren Skalen. Für weitere Informationen folgen Sie bitte anderen verwandten Artikeln auf der PHP chinesischen Website!
In Verbindung stehende Artikel
Mehr sehen- Detaillierte Erläuterung der theoretischen Grundlagen des neuronalen Netzwerks und der Python-Implementierungsmethode
- Welche Programmtheorie liegt dem virtuellen Speicherverwaltungssystem zugrunde?
- „Aufruf zur Innovation: UCL Wang Jun diskutiert die Theorie und Anwendungsaussichten der allgemeinen künstlichen Intelligenz von ChatGPT'

