Heim >Technologie-Peripheriegeräte >KI >SOTA-Leistung, mehrskaliges Lernen, Sun Yat-sen-Universität schlägt KI-Framework für Protein-Arzneimittel-Interaktion vor
SOTA-Leistung, mehrskaliges Lernen, Sun Yat-sen-Universität schlägt KI-Framework für Protein-Arzneimittel-Interaktion vor
- WBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOYWBOriginal
- 2024-06-04 15:43:571346Durchsuche
Herausgeber |. Violet
Wechselwirkungen zwischen Proteinen, Medikamenten und anderen Biomolekülen spielen eine entscheidende Rolle in verschiedenen biologischen Prozessen. Das Verständnis dieser Wechselwirkungen ist entscheidend für die Entschlüsselung der molekularen Mechanismen, die biologischen Prozessen zugrunde liegen, und für die Entwicklung neuer Therapiestrategien. Proteine gehören zu den wichtigsten Molekülen in Zellen und erfüllen innerhalb der Zellen verschiedene Funktionen. Medikamente regulieren häufig physiologische Prozesse, indem sie mit bestimmten Proteinen interagieren. Diese Wechselwirkungen können bestimmte molekulare Signalwege fördern oder hemmen. Aktuelle Multiskalen-Berechnungsmethoden stützen sich oft zu sehr auf eine einzelne Skala und sind für andere Skalen unzureichend. Dies kann mit dem ungleichmäßigen Multiskalen-Tropismus und der inhärenten Gier des Multiskalen-Lernens zusammenhängen.
Um das Optimierungsungleichgewicht zu mildern, haben Forscher der Sun Yat-sen-Universität und der Shanghai Jiao Tong-Universität ein mehrskaliges Repräsentations-Lernframework MUSE vorgeschlagen, das auf der Maximierung variabler Erwartungen basiert und mehrskalige Informationen effektiv zum Lernen integrieren kann. Diese Strategie vereint effektiv Multiskaleninformationen zwischen der Atomstruktur und den molekularen Netzwerkskalen durch gegenseitige Überwachung und iterative Optimierung. Dieser Ansatz ermöglicht einen besseren Informationstransfer und ein besseres Lernen. Diese Strategie vereint effektiv Multiskaleninformationen zwischen der Atomstruktur und den molekularen Netzwerkskalen durch gegenseitige Überwachung und iterative Optimierung.
MUSE+ übertrifft nicht nur die aktuellen hochmodernen Modelle für molekulare Interaktionsaufgaben (Protein-Protein, Medikament-Protein und Medikament), sondern auch die aktuellen hochmodernen Modelle für Proteinschnittstellenvorhersagen auf der atomaren Strukturskala. Noch wichtiger ist, dass das Multiskalen-Lernkonzept auf die rechnergestützte Wirkstoffentdeckung auf anderen Skalen ausgeweitet werden kann.
Die Studie mit dem Titel „
Ein Rahmenwerk zur Variationserwartungsmaximierung für ausgewogenes mehrskaliges Lernen von Protein- und Arzneimittelinteraktionen“ wurde am 25. Mai in „Nature Communications“ veröffentlicht. 🔜 Wechselwirkungen mit anderen Biomolekülen. Das Verständnis dieser Wechselwirkungen ist entscheidend für die Entschlüsselung der molekularen Mechanismen biologischer Prozesse und die Entwicklung neuer Therapiestrategien. Der erhebliche Anstieg der Anforderungen und Kosten im Zusammenhang mit experimentellen Wechselwirkungen erfordert jedoch Rechenwerkzeuge, um Wechselwirkungen zwischen Biomolekülen automatisch vorherzusagen und zu verstehen. Um diesen Anforderungen und steigenden Kosten gerecht zu werden, werden Rechenwerkzeuge benötigt, um Wechselwirkungen zwischen Biomolekülen automatisch vorherzusagen und zu verstehen. Die Vorhersage dieser Wechselwirkungen rein aus der Struktur ist eine der wichtigsten Herausforderungen in der Strukturbiologie. Aktuelle Berechnungsmethoden sagen Wechselwirkungen meist auf der Grundlage molekularer Netzwerke oder Strukturinformationen voraus und integrieren sie nicht in ein einheitliches Multiskalen-Framework.
Während einige Multi-View-Lernmethoden darauf abzielen, mehrskalige Informationen zu verschmelzen, besteht eine intuitive Möglichkeit, mehrskalige Darstellungen zu lernen, darin, molekulare Graphen mit Interaktionsnetzwerken zu kombinieren und diese gemeinsam zu optimieren. Aufgrund des Ungleichgewichts und der inhärenten Gier des mehrskaligen Lernens stützen sich diese Modelle jedoch häufig stark auf eine einzige Skala. Die Fähigkeit, Informationen aller Skalen und deren Verallgemeinerung effektiv zu nutzen, ist mangelhaft. 
Hier schlug das Forschungsteam der Sun Yat-sen-Universität MUSE vor, ein Lernrahmen für mehrskalige Repräsentationen, der auf der Maximierung variabler Erwartungen (Expectation Maximization) basiert mehrfach verwendet Verschiedene Skalen werden in einem iterativen Wechselprozess optimiert. Im Vergleich zu bestehenden Methoden, die stark auf Einzelskaleninformationen basieren, löst MUSE das Problem des Optimierungsungleichgewichts beim Lernen auf mehreren Skalen effektiv durch gegenseitige Überwachung und iterative Optimierung.
MUSE ist eine mehrskalige Lernmethode, die die Modellierung molekularer Strukturen und das Lernen von Protein-Wirkstoff-Interaktionsnetzwerken durch ein Variationserwartungsmaximierungs-Framework (EM) kombiniert. Das EM-Framework optimiert zwei Module, nämlich den Erwartungsschritt (E-Schritt) und den Maximierungsschritt (M-Schritt), in einem abwechselnden Prozess mehrerer Iterationen. Während des E-Schritts nutzt MUSE die Strukturinformationen jedes Biomoleküls, um mithilfe bekannter Wechselwirkungen und Verstärkungsproben effektive Strukturdarstellungen für das Training im M-Schritt zu lernen. Als Eingabe werden Protein- und Arzneimittelpaare und deren Strukturinformationen auf atomarer Ebene verwendet, ergänzt durch vorhergesagte M-Schritt-Wechselwirkungen. M-Step verwendet als Eingabe die vorhergesagten Wechselwirkungen des Interaktionsnetzwerks auf molekularer Ebene, der strukturellen Einbettungen und des E-Schritts und gibt die vorhergesagten Wechselwirkungen aus. Die iterative Optimierung zwischen E-Schritt und M-Schritt gewährleistet die interaktive Erfassung molekularer Struktur- und Netzwerkinformationen mit unterschiedlichen Lernraten auf den beiden Skalen.Die gegenseitige Aufsicht stellt sicher, dass jedes maßstabsgetreue Modell auf angemessene Weise lernt und ermöglicht so die Nutzung effektiver Informationen in verschiedenen Maßstäben. Dieses Framework wird über mehrere Multiskalen von Wechselwirkungen zwischen Proteinen und Arzneimitteln demonstriert. Es wird analysiert, dass MUSE die unausgewogenen Merkmale beim mehrskaligen Lernen lindert und hierarchische und komplementäre Informationen aus verschiedenen Skalen effektiv integriert.
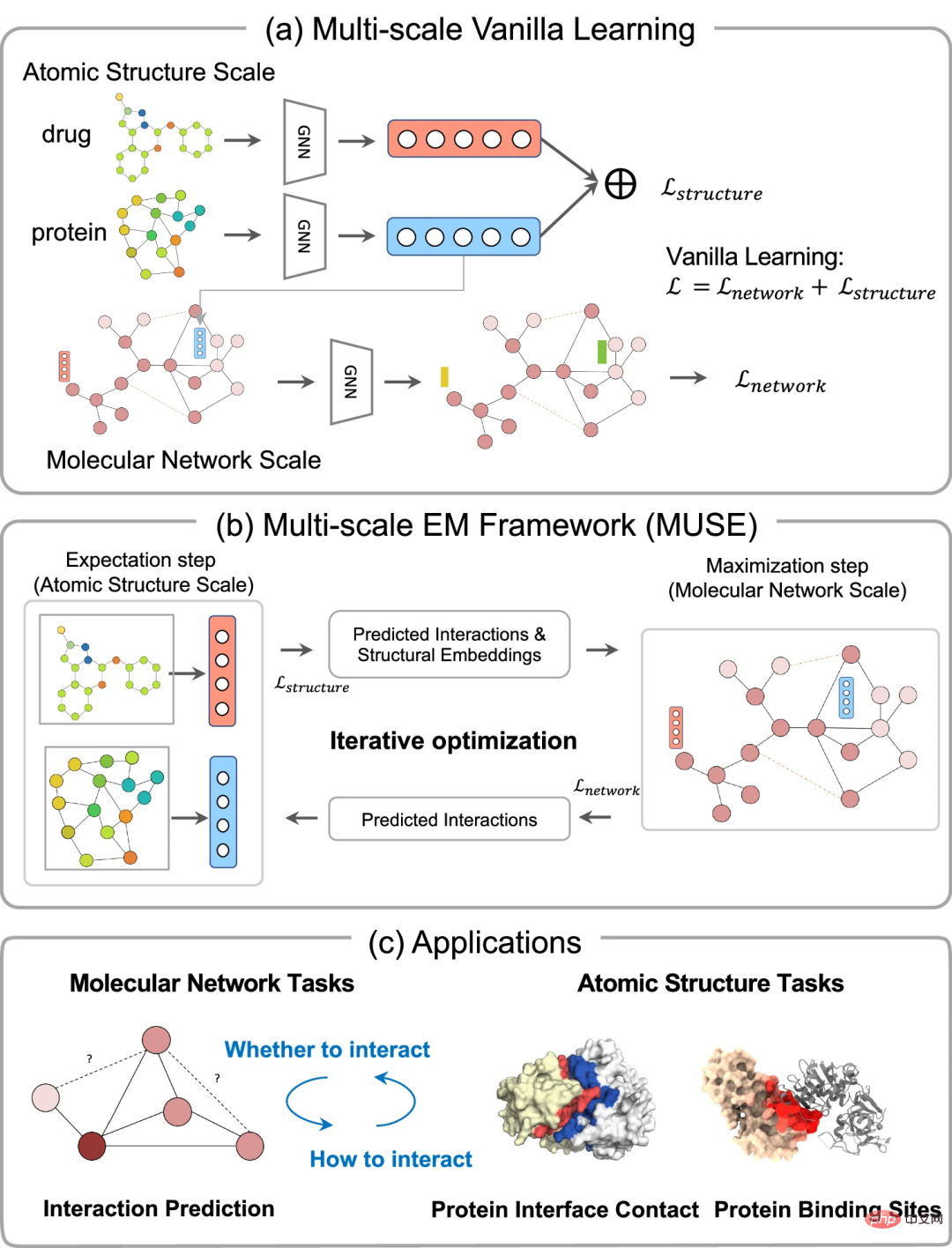
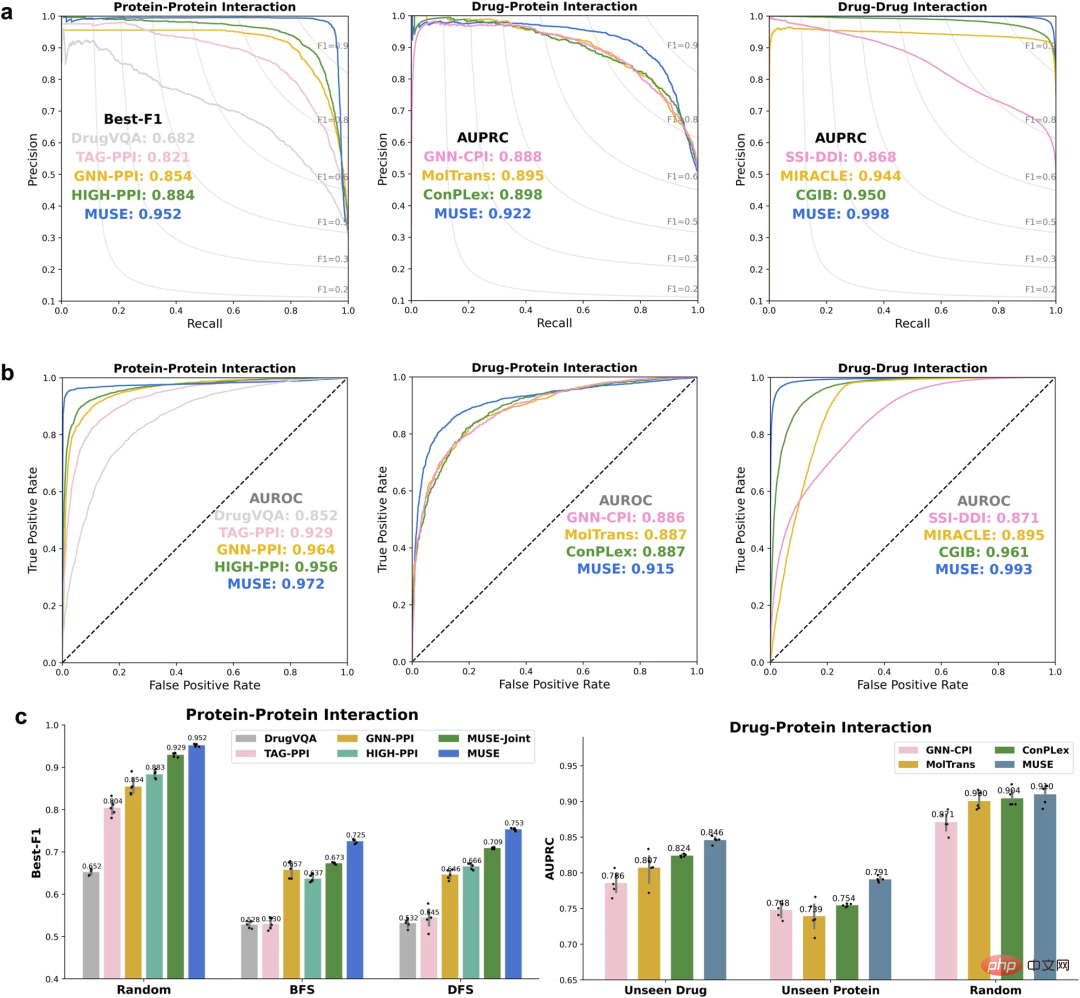
Übertrifft aktuelle Modelle auf dem neuesten Stand der Technik bei Aufgaben auf mehreren Skalen
Nutzt atomare Strukturinformationen, um Vorhersagen auf der Ebene molekularer Netzwerke zu verbessern
Um ihren Ansatz zu bewerten, werden zunächst die Forscher nutzten MUSE, um Atomstrukturinformationen zu integrieren und so Vorhersagen im Maßstab molekularer Netzwerke zu verbessern. MUSE erreicht Spitzenleistungen bei drei Aufgaben zur Vorhersage von Wechselwirkungen auf mehreren Ebenen: Protein-Protein-Interaktion (PPI), Arzneimittel-Protein-Interaktion (DPI) und Arzneimittel-Wirkstoff-Interaktion (DDI).
Verbesserung der Vorhersagen der atomaren Strukturskala auf der Skala des molekularen Netzwerks
Zusätzlich zur Verwendung von Atomstrukturinformationen zur Verbesserung der Vorhersagen der molekularen Netzwerkskala untersuchten die Forscher weiter die Fähigkeit von MUSE, Strukturen auf der Atomstruktur zu lernen und vorherzusagen Skala Die Fähigkeit zur Charakterisierung, einschließlich der Vorhersage von Grenzflächenkontakten und Bindungsstellen, die mit PPIs verbunden sind.
Um Vorhersagen zu Protein-Interkettenkontakten zu bewerten, wurde MUSE mit modernsten Methoden im DIPS-Plus-Benchmark verglichen. MUSE übertrifft durchweg alle anderen Methoden und bestätigt seine Wirksamkeit und Anpassungsfähigkeit bei der Vorhersage der Atomstruktur.
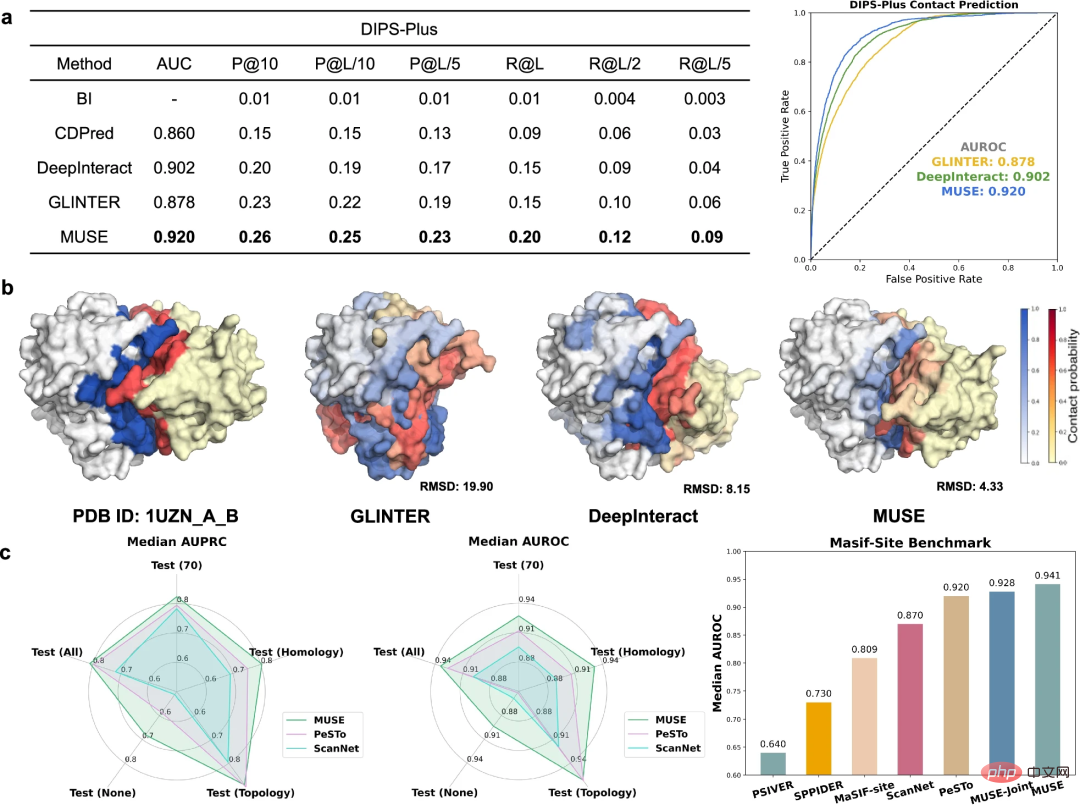
MUSE wurde weiter evaluiert, um vorherzusagen, ob Rückstände direkt an Protein-Protein-Wechselwirkungen beteiligt sind. Die Ergebnisse zeigen, dass das Lernen im molekularen Netzwerkmaßstab in MUSE wertvolle Einblicke in Vorhersagen der atomaren Strukturskala liefern kann.
Verringern Sie die Ungleichgewichtsmerkmale des mehrskaligen Lernens durch iterative Optimierung.
Um herauszufinden, warum MUSE bei der mehrskaligen Darstellung eine überlegene Leistung erzielen kann, analysierten die Forscher die Lernfähigkeit von MUSE basierend auf den Ungleichgewichtsmerkmalen des mehrskaligen Lernens Lernen.
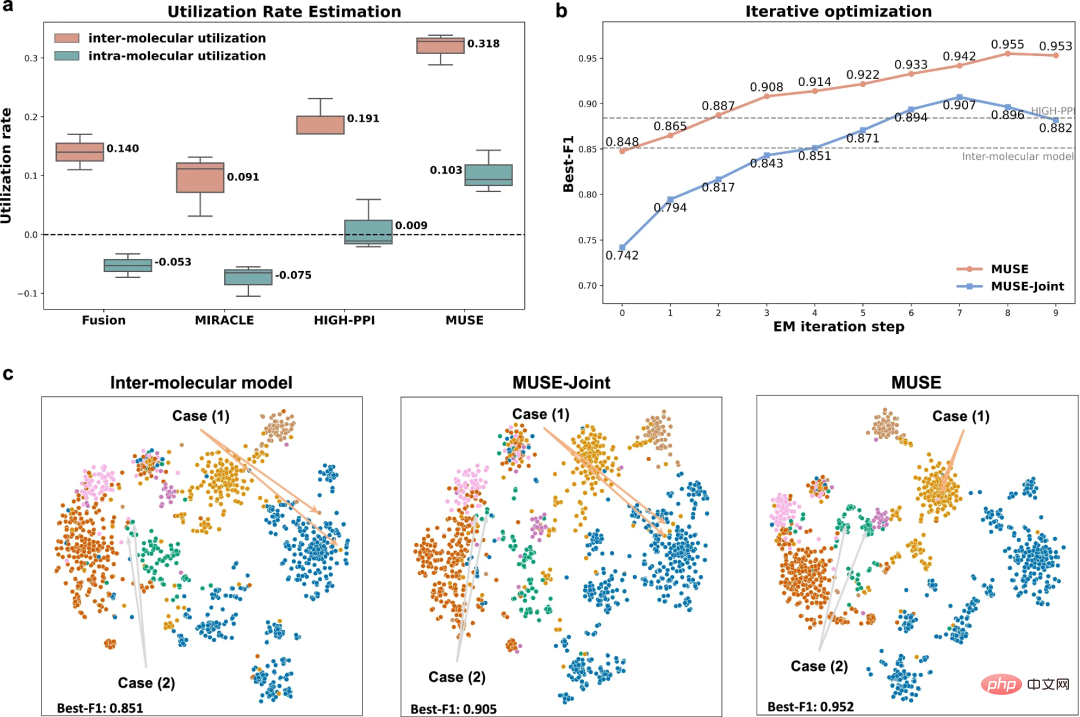
Die Ergebnisse zeigen, dass MUSE die Ungleichgewichtsmerkmale und das gierige Lernen beim mehrskaligen Lernen effektiv lindert und die umfassende Nutzung von Informationen auf verschiedenen Skalen während des Trainingsprozesses gewährleistet. Darüber hinaus ermöglichten Experimente mit der Analyse der Nutzungsrate den Forschern, konkret zu verstehen, was das Modell gelernt hat, und zeigten, dass die Verwendung von MUSE zum Ausbalancieren des Modelllernens auf verschiedenen Skalen die Generalisierungsfähigkeiten verbessern kann.
Visualisierung und Interpretation der erlernten mehrskaligen Darstellung
Um die erlernte mehrskalige Darstellung besser zu verstehen, untersuchten die Forscher die von MUSE erlernte mehrskalige Darstellung aus verschiedenen Blickwinkeln, darunter (1) MUSE-Erfassung Die Fähigkeit von Atomaren Strukturinformationen (d. h. Strukturmotive und Einbettungen), die an PPI beteiligt sind, und (2) gegenseitige Überwachung zwischen erlernten Atomstrukturen und molekularen Netzwerkdarstellungen.
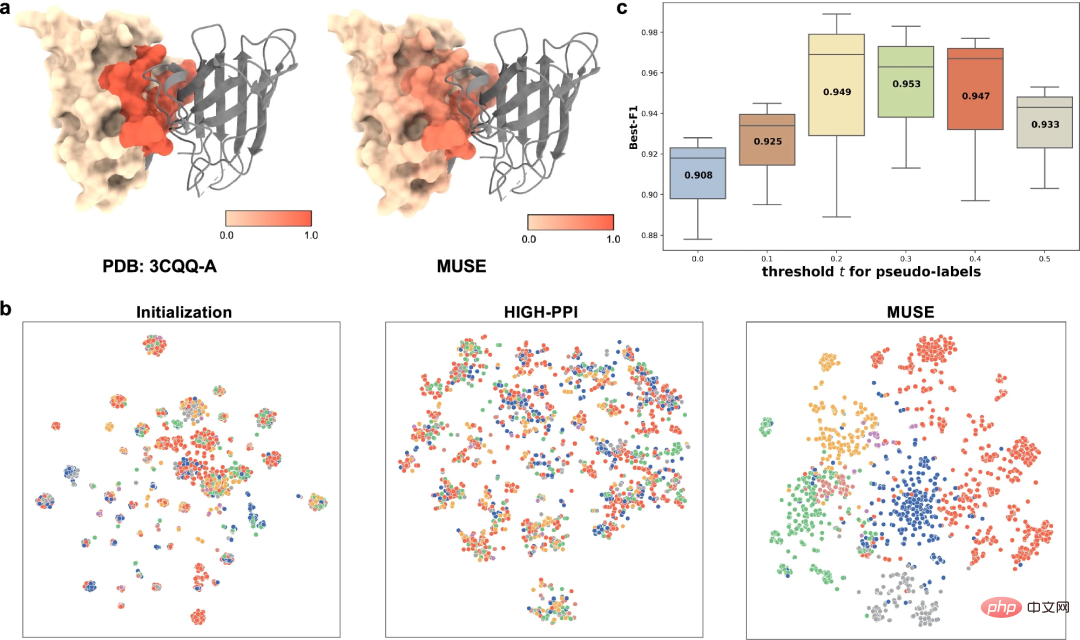
Als Beispiel für die Vorhersage der Bindungsstelle (PDB-ID: 3CQQ-A) kann MUSE Reste, die zur Bindungsstelle gehören, mit einer Genauigkeit von 97,7 % genau identifizieren. Dies deutet darauf hin, dass die gegenseitige Überwachung in MUSE atomaren Strukturmodellen dabei hilft, wichtige Unterstrukturen zu erlernen, die für Wechselwirkungen relevant sind.
Schließlich führten die Forscher auch Ablationsstudien durch, um den Einfluss von Pseudomarkierungen zu untersuchen, die auf der Ebene der Atomstruktur auf der Ebene des molekularen Netzwerks vorhergesagt wurden.
Während MUSE in Benchmarks eine Leistung auf dem neuesten Stand zeigt, ist es dennoch möglich, seine Fähigkeit zur Bewältigung lauter und unvollständiger Multiskalen-Downstream-Aufgaben zu verbessern. Dies kann durch die Kombination von Vorwissen durch Wissensgraphen und erklärbare KI-Techniken erreicht werden. Andererseits kann dieser konzeptionelle Multiskalenrahmen auch auf die rechnergestützte Arzneimittelentwicklung auf anderen Skalen ausgeweitet werden.
Das obige ist der detaillierte Inhalt vonSOTA-Leistung, mehrskaliges Lernen, Sun Yat-sen-Universität schlägt KI-Framework für Protein-Arzneimittel-Interaktion vor. Für weitere Informationen folgen Sie bitte anderen verwandten Artikeln auf der PHP chinesischen Website!

